class: center, middle, inverse, title-slide # Modelagem de distribuição de espécies ## Uma introdução com exemplo no R ### Marília Melo Favalesso ### 31-10-2020 --- background-image: url(https://www.mmfava.com/marilia.png) background-size: 300px background-position: 90% 5% <style type="text/css"> pre { max-height: 600px; overflow-y: auto; } pre[class] { max-height: 600px; } </style> <style type="text/css"> h2 { color: brown; } </style> ### Marília Melo Favalesso **Formação** - Técnica ambiental (CEEP, 2009) - Bióloga (UFPR, 2014) - Mestre em Ciências Ambientais (UNIOESTE, 2018) - Doutoranda em Ecologia (UBA - Argentina, atual) **Projetos** - [**GECD** - Grupo de Estudos em Ciência de Dados](https://github.com/gecdfoz/GECD) - [**Soma dos quadrados**](https://linktr.ee/somaquadrados) - [**Grupo de Estudos em Modelagem de nicho e distribuição de espécies**](https://linktr.ee/niche_group) **Contatos** - ✉️ [mariliabioufpr@gmail.com](mariliabioufpr@gmail.com) - 🌎 [www.mmfava.com](www.mmfava.com) - 🥚 [Twitter: @mmfbee](https://twitter.com/mmfbee) - 💻 [Github: mmfava](https://github.com/mmfava) --- class: clear background-image: url(figuras/Slide6.PNG) background-size: 500px ## O que é um modelo? --- class: clear background-image: url(figuras/Slide7.PNG) background-size: 600px ## O que é um modelo? --- class: clear background-image: url(figuras/Slide8.PNG) background-size: 1000px ## Modelos de distribuição de espécies --- class: clear background-image: url(figuras/Slide9.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide10.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide11.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide12.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide13.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide14.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide15.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide16.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide17.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide18.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide19.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide20.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide21.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide22.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide23.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide24.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide25.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide26.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide27.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide28.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide29.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide30.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide31.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide32.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide33.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide34.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide35.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide36.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide37.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide38.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide39.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide40.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide41.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide42.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide43.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide44.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide45.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide46.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide47.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide48.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide49.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide50.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide51.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide52.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide53.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide54.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide55.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide56.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide57.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide58.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide59.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide60.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide61.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide62.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide63.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide64.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide65.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide66.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide67.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide68.PNG) background-size: 1000px --- class: clear background-image: url(figuras/raster.gif) background-size: 500px ## Raster --- class: clear background-image: url(figuras/Slide70.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide71.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide72.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide73.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide74.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide75.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide76.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide77.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide78.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide79.PNG) background-size: 1000px --- class: clear background-image: url(figuras/Slide80.PNG) background-size: 1000px --- ## Modelagem de distribuição de espécies com o R ### Exemplo com *Trigona spinipses* na América do Sul ### Pacotes: ```r ## Instalar o pacote ENTML devtools::install_github("andrefaa/ENMTML") ## Carregar os pacotes install.packages(maptools) # pacote para abrir mapas diretamente no R install.packages(raster) # para trabalhar com rasters install.packages(dismo) # função gbif - download de ocorrências de espécies. install.packages(tidyverse) # trabalhar com as planilhas install.packages(ENMTML) # pacote para modelagem de nicho install.packages(leaflet) # pacote para mapas interativos - usaremos nos resultados ``` --- ## Modelagem de distribuição de espécies com o R ### Dados de ocorrência do GBIF - Vamos baixar dados de ocorrência de *T. spinipes* do [database GBIF](https://www.gbif.org/). ```r # - Vamos usar a função "gbif()" para fazer o download dos dados. pontos = dismo::gbif('Trigona', # gênero da espécie species = "Trigona spinipes", # nome completo da espécie sp = TRUE, # Retornar como um SpatialPointsDataFrame? removeZeros = TRUE, # Remover linhas com dados de latitude e/ou logitude faltantes download = TRUE) # Para realizar o dowload dos registros # - Colocar o nome "data" na guia de valores (que são as datas de amostragem). names(pontos) = "data" ``` --- ## Modelagem de distribuição de espécies com o R ### Salvar os dados de ocorrência para utilizar com o pacote ENMTML - O arquivo precisa ser salvo em .TXT (separado por tabulação)! - Precisamos apenas de três colunas em nossa tabela: - 1o nome da espécie - 2o longitude - 3o latitude <div style="width: 1050px; height: 320px; white-space: nowrap; overflow-x: scroll; overflow-y: scroll; border: 0; padding: 0px; display: inline-block;"> ```r library(tidyverse) # Primeiro vamos gerar um objeto com o endereço do nosso diretório de trabalho: d_ex = file.path('C:/Users/mmfav/Dropbox/nichemod') d_ex ``` ``` ## [1] "C:/Users/mmfav/Dropbox/nichemod" ``` ```r # Extrair as informações dos pontos para uma tabela: tabela = as.data.frame(pontos) tabela = as.data.frame(pontos) %>% # transformar os dados em data.frame select('lon', 'lat') %>% # selecionar apenas long e lat add_column('species' = c(rep('Trigona spinipes', length(tabela$lon))), # Incluir uma coluna com nome da sp ... .before = 'lon') # ... antes de coluna "lat". # Mudar o nome das colunas para facilitar incluir na função depois names(tabela)[2:3] = c("x", "y") # Salvar os pontos de ocorrência como .txt # - criar uma pasta para os pontos no diretório chamada "occ": dir.create(paste0(d_ex, '/occ')) # - salvar os pontos de ocorrência em um arquivo chamado "occ.txt" utils::write.table(tabela, file.path(d_ex, 'occ/occ.txt'), sep = '\t', row.names = FALSE) ``` --- ## Modelagem de distribuição de espécies com o R ### Dados de ocorrência do GBIF ```r knitr::kable(x = head(pontos), format = "html") ``` <table> <thead> <tr> <th style="text-align:left;"> data </th> <th style="text-align:right;"> lon </th> <th style="text-align:right;"> lat </th> </tr> </thead> <tbody> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -46.99378 </td> <td style="text-align:right;"> -22.94122 </td> </tr> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -46.63464 </td> <td style="text-align:right;"> -23.45814 </td> </tr> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -48.45403 </td> <td style="text-align:right;"> -22.86630 </td> </tr> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -46.57087 </td> <td style="text-align:right;"> -23.73215 </td> </tr> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -46.92160 </td> <td style="text-align:right;"> -23.28016 </td> </tr> <tr> <td style="text-align:left;"> 2021-04-08 </td> <td style="text-align:right;"> -44.87107 </td> <td style="text-align:right;"> -23.15217 </td> </tr> </tbody> </table> --- ## Modelagem de distribuição de espécies com o R ### Área de projeção do modelo - Polígono do mapamundi ```r library(maptools) data("wrld_simpl") ``` - Recortando o mapa para uma extensão mais próxima dos pontos de ocorrência (background). 1 - Olhar a saída de 'pontos' e ver a dimensão geográfica dele ```r pontos ``` ``` ## class : SpatialPointsDataFrame ## features : 12496 ## extent : -75.9972, -34.7965, -31.395, -0.519111 (xmin, xmax, ymin, ymax) ## crs : NA ## variables : 1 ## names : data ## min values : 18725 ## max values : 18725 ``` 2 - Salvar a extensão em um objeto chamado "ext". ```r ext = c(-85, -34.81973, -40, 0) # colocar um pouquinho mais amplo que os pontos ``` --- ## Modelagem de distribuição de espécies com o R ### Área de projeção do modelo 3 - Recortar o mapa mundi para a extensão selecionada. ```r map = raster::crop(wrld_simpl, ext) ``` --- ## Modelagem de distribuição de espécies com o R ### Background <div style="width: 1000px; height: 400px; white-space: nowrap; overflow-x: scroll; overflow-y: scroll; border: 0; padding: 0px; display: inline-block;"> ```r plot(map, # nosso limite territorial - America do sul axes = T, # Pedir para incluir os eixos das coordenadas lwd = 1.8, # Grossura da linha do mapa col = "#CCFF99", # colorir os países de verde bg = "#CCFFFF") # cor do background da imagem em azul # Plotar o nome dos países no mapa text(map$LON, map$LAT, map$NAME, cex = 0.7, font = 2) # Os pontos de ocorrência da nossa espécie points(pontos, # chamar o poligono de pontos pch = 20, # estilo dos pontos de ocorrência col = 'red', # na cor vermelha cex = 1.5) # tamanho de 1.5 # Incluir legenda com o nome da espécie legend('bottomright', # do lado inferior-direito pch = 20, # mesmo estilo dos pontos que antes legend = c(as.expression(bquote(italic("Trigona spinipes")))), # Nome da sp em itálico col = 'red', # Cor do ponto em preto bg = 'white', # Cor do background em branco bty = 0, # Tipo de caixa ao redor da legenda cex = 1) # Tamanho da legenda ``` 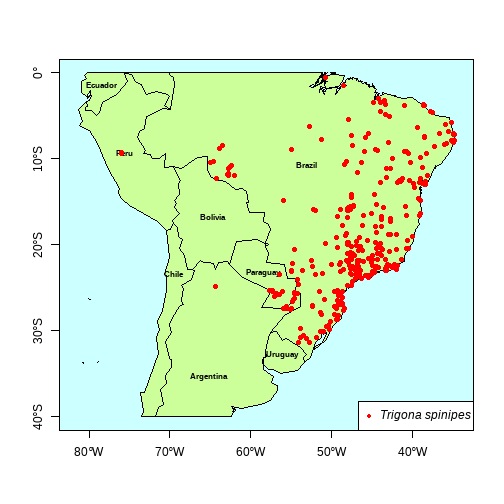<!-- --> --- ## Modelagem de distribuição de espécies com o R ### Dados biogeoclimáticos - Chamar os dados para o R com a função `getData()`. - Vamos usar os dados da plataforma [wordclim](https://www.worldclim.org/). ```r # diretório de trabalho setwd('C:/Users/mmfav/Dropbox/nichemod') # download data r = raster::getData("worldclim", # banco de dados var = "bio", # var. biogeoclimáticas res = 10) # resolução = 10m ``` - Recortar os rasters pela extensão da América do sul ```r rr = raster::crop(raster::mask(r, map), map) ``` --- ## Modelagem de distribuição de espécies com o R ### Dados biogeoclimáticos - Visualizar as variáveis biogeoclimáticas ```r plot(rr) ``` 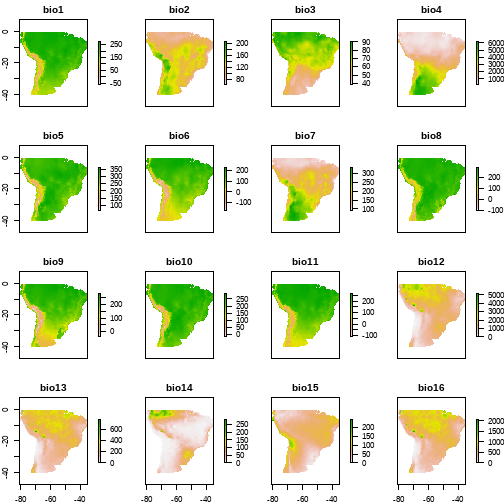<!-- --> --- ## Modelagem de distribuição de espécies com o R ### Dados biogeoclimáticos - Salvando os dados bioclimáticos em um diretório próprio ```r # Criar um diretório chamado 'env': dir.create(paste0(d_ex, '/env')) # Para salvar cada um dos rasters individualmente no repositório: for(i in names(rr)){ # para cada raster em 'rr'... raster::writeRaster(rr[[i]], paste0(file.path(d_ex, 'env'), "/", i, '.tif'), overwrite = T) } ``` --- ## Modelagem de distribuição de espécies com o R ### Modelando a distribuição com o pacote ENMTML - Criar uma pasta/diretório para salvar os resultados do(s) modelo(s) ```r dir.create(paste0(d_ex, '/resultados')) ``` - Vamos criar objetos com os endereços dos diretórios. Vamos usar os objetos na função `ENMTML()`. ```r # endereço das variáveis climáticas d_env = file.path("C:/Users/mmfav/Dropbox/nichemod/env") # endereço dos pontos de ocorrência d_occ = "C:/Users/mmfav/Dropbox/nichemod/occ/occ.txt" ``` --- ## Modelagem de distribuição de espécies com o R ### Modelando a distribuição com o pacote ENMTML <div style="width: 1100px; height: 400px; white-space: nowrap; overflow-x: scroll; overflow-y: scroll; border: 0; padding: 0px; display: inline-block;"> ```r ENMTML::ENMTML( pred_dir = d_env, # Endereço para o diretório das variáveis proj_dir = NULL, # Quando trabalhamos com projeções - diferentes locais no espaço ou tempo # result_dir = res, # Pasta para salvar as saídas/resultados occ_file = d_occ, # Pasta onde estão nossos pontos de ocorrência sp = 'species', # coluna que contêm o nome das espécies x = 'x', # coluna que corresponde a longitude y = 'y', # coluna que corresponde a latitude min_occ = 10, # Número minimo de ocorrências para cada espécie thin_occ = NULL, # Inserimos os métodos de filtragem de ocorrências dados redundantes eval_occ = NULL, # Tabela com dados de ocorrência para validação dos dados colin_var = c(method='PEARSON', threshold='0.7'), # Onde inserimos os métodos para evitar a colinearidade entre os dados imp_var = FALSE, # Calcula a importância das variáveis em curvas de resposta sp_accessible_area = NULL, # Método de restrição de área acessível para as espécies pseudoabs_method = c(method = 'RND'), # Método de seleção de pseudo-ausências aleatório pres_abs_ratio = 1, # Razão presença-ausência (valores entre 0 e 1) part=c(method= 'KFOLD', folds='10'), # Método de partição para validação do modelo. save_part = FALSE, # Save partitioned data? save_final = TRUE, # Salvar o modelo final em .tif (?) algorithm = c('BIO', 'MAH'), # algoritmos de modelagem thr = c(type='MAX_TSS'), # Para binarizar o mapa, qual método utilizar? msdm = NULL, # Métodos de restrição no modelo para próximo das ocorrências conhecidas ensemble = c(method='PCA'), # Qual método ensemble, ou de combinação, realizar entre os diferentes algoritmos? extrapolation = FALSE, # Se TRUE, a função calculará a extrapolação com base na análise de paridade orientada para # a mobilidade (MOP) para as condições atuais cores = 1 # Defina o número de núcleos de CPU para executar procedimentos de modelagem em paralelo (padrão 1). ) ``` ``` ## Registered S3 methods overwritten by 'adehabitatMA': ## method from ## print.SpatialPixelsDataFrame sp ## print.SpatialPixels sp ``` ``` ## Checking for function arguments ... ## Loading environmental variables ... ## RasterBrick successfully created! ## Performing a reduction of variables collinearity ... ## Loading and processing species occurrence data ... ``` ``` ## Warning in ENMTML::ENMTML(pred_dir = d_env, proj_dir = NULL, occ_file = d_occ, : ## Result folder already exists, files may be overwritten! ``` ``` ## Results can be found at: ## C:/Users/mmfav/Dropbox/nichemod/Result ## Performing partition of species occurrence data ... ## Adjusting fold....1 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....2 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....3 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....4 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....5 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....6 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....7 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....8 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....9 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Adjusting fold....10 ``` ``` ## Total species to be modeled 1 ``` ``` ## Fitting Models.... ## Models fitted! ## Performing Ensemble.... ## Ensemble created! ## Models were created successfully! ## Outputs are in: ## C:/Users/mmfav/Dropbox/nichemod/Result ``` --- ## Modelagem de distribuição de espécies com o R ### Visualização dos resultados - Vamos plotar os resultados usando o pacote "leaflet" <div style="width: 1100px; height: 400px; white-space: nowrap; overflow-x: scroll; overflow-y: scroll; border: 0; padding: 0px; display: inline-block;"> ```r library(tidyverse) library(leaflet) library(raster) # Abrir o resultado em objeto chamado 'res' res = paste0(d_ex, '/Result/Ensemble/PCA/Trigona_spinipes.tif') %>% raster() # Fazer uma palheta de cores variando de verde até vermelho; para a legenda de 'suitability'. pal = colorNumeric(c("#003300", "#006633", "#009300", "#FFFFCC", "#FF9900", "#FF6600", "#FF3300"), values(res), na.color = "transparent") # Plotar em mapa interativo com LEAFLET map = leaflet(pontos) %>% # Usaremos a função 'leaflet' para produzir os mapas e já chamaremos os pontos de occ. addTiles() %>% # add um mapa de fundo addRasterImage(res, # adicionar o raster 'res' (com nossos resultados) col = pal, opacity = 0.5, group = "Suitability") %>% # com cor = pal, transparência de 50% e nome suitability addLegend(pal = pal, values = values(res), title = "Suitability", group = "Suitability") %>% # add legenda para raster addLayersControl(overlayGroups = c("Suitability"), # Incluir opção de ligar e desligar o raster options = layersControlOptions(collapsed = FALSE)) %>% addCircleMarkers(radius = 1, col = "black") %>% # adicionar os pontos de ocorrência como circulos addScaleBar() # Adicionar uma barra de escala ``` --- ## Modelagem de distribuição de espécies com o R ### Visualização dos resultados - Vamos plotar os resultados usando o pacote "leaflet". ```r map ``` <div id="htmlwidget-c00bdb0b98315807359e" style="width:504px;height:504px;" class="leaflet html-widget"></div> <script type="application/json" data-for="htmlwidget-c00bdb0b98315807359e">{"x":{"options":{"crs":{"crsClass":"L.CRS.EPSG3857","code":null,"proj4def":null,"projectedBounds":null,"options":{}}},"calls":[{"method":"addTiles","args":["//{s}.tile.openstreetmap.org/{z}/{x}/{y}.png",null,null,{"minZoom":0,"maxZoom":18,"tileSize":256,"subdomains":"abc","errorTileUrl":"","tms":false,"noWrap":false,"zoomOffset":0,"zoomReverse":false,"opacity":1,"zIndex":1,"detectRetina":false,"attribution":"© <a href=\"http://openstreetmap.org\">OpenStreetMap<\/a> contributors, <a href=\"http://creativecommons.org/licenses/by-sa/2.0/\">CC-BY-SA<\/a>"}]},{"method":"addRasterImage","args":["data:image/png;base64,iVBORw0KGgoAAAANSUhEUgAAARcAAADwCAYAAAAq5SBaAAAgAElEQVR4nOy9ebQl2VXm9ztjRNzpzTlnVmbWrNJcEhIqQAIEhiXaCJtBhoYGhKFNw8JAA27aqxHd6mbohWiGxgsbcBsaWsLCLBCDYBkEAqMBqSRRQlVSZWVmVc6Zb7pzDGfwHyfufVmoJCEmL4wi161X7977IuKeiPOdb3/72/uKGCMfc/vNL4ry/FuJv3MYrl89eF5IOHacf/J1d/GmK2/nPV/287zwp/8F9Vsug5D8x5/4Zv7HX/ldmt977GPve7Gtb0KnA2uriEKh8ga3LxFWI61HWofQHmQgrAwRUUAU4DUiSAgq/V4bYq3xpSF4CVUNIYDSoBVCQXQRnIeqgtkcxhOYz6AuP/F5fqKtO4D1NVgZgNFQVuA9yPZcYoB5Cf0eKJUeMYJWgEj7WJ7XGMYjyDuwsQ5rq6guCBUR2iPyGrQDEcEraDRhnuFmGoRAd2pkXkNWE/M5wVQEPcfqhg3VpSdzAHJlMUKjhQQhONk9RNfkZMqSKUPlGwRw9/odnNu7hFWGXFvmTcWkmbE93+fabIdCZ5S+Zu4rcmmRQnL3ygnmrqJnCjJlyXVGJDCuZ0zqGUoqCp2RKYsSkmkzZ1RPccFjlEYg0FIBEGJgv5os/9/FwNxXZNIw8xVPlNeQCDb1gEjk8W+8JZ7pEtnveiAiIDYK99hN2N0mvss943s/tf31N/2xXnjX5Z+JL+kfRlUruLp++otnzqBeeovv+PSv5xd//hF+6c9+nfrXngKlsK88w7e97U24c52Pf2QhwOawugJFkYDFeAgSfCAOJ3hrCJ0MlTmE8shJnzjPELaBKIhOEb0EAbHWBC/S742DCBiDMBIhIjGINNHxabIvJv/fBLAATEegJGQWYpaAQwjY3QOtQYoEOq4FHCEQPYtUAaEioVaEdtKjFOQ5hAh1DeMJ3qxiVmeIoiSaJoFLkOAVQkREFGgZ0tAaD9oTTUPQNVGXGF1zSA2oYwPA+aEnmB1etnaSma/YtCuEGKhcTYgRLRVaKkKMnN+/TIiBED3zpmK3HFK6mhvzXd4zexIRFbLuEVVFFAGEZ+SmZNJwvLPFWtYnxIAQAiUkRmrq4FBCYpVGiQQic1dR+YamKTFSIwAhJAAdnbNfj/Ex0ARPFRokghgjR/QqTfScr7dx3xIEQPav7orVvz4nxDe8IMpMgIioXIAI6d5YX4OyRLxyM9qHVoh5iXAaZgXVDzz2KcD5G9ieEVz+1/d8RzwxOArrp5HDdXDbT3vdPuD5ohP383996HeQ8y4//Ft/nl7oDjhzx4jzH96EW099/CPbHA4fhm4H2VVI2yBkJDQKUVjiZAreE53HFQVm1RMmBUD62RKu2DKZGNID7xNjsQahQKiQmA2kydo06T1NA+Xs6eek0sqP94lpfLJb42A0hsKBNQkkrIFuF6yFyQSyDNErEDokwNQedEBpD9IQRJH+plMkEFISjEF3K0TLRKJuCGaGKgf4YohsCqTsI2T7WbUHLxFVhtA1SEMjay77KXq2wY4MBFPx0Popnpzd4Fi+wYLB1sGBENS+QUvFpJmzng8QQrBXjildxaie8cT0KufrXaQvkE0HURcgOkTdEKXnI35G1LMEHDGymvXJtQUESio0kULnFDpHS41RBiUV9fg6o3qKEAIdFbId2kiko3PGzZwmOAa6gxQCKQSRyBHT5aWbz1peiupfnxPiGx+MCEFwEqkDMQiEishOhT0T4Z61xP7wIAJRBoSMqG9+YfQ/9fCnAOavuclnevIbX/QGcWV8Hfes/4qYlzAaPu31Zu0Khc54/a8+Qn15A/f2D6YXVgacv2lp3nURZuOPfVQhwNj0M7OorEFmDcK4NNkEkGfgAzgHRJpRhpsa/NwQGkUMkhgOTl/IiJARpETkBqkjUgeEoAUeEguYz2E6S+GQaw7OSRvoD2B9Azq9FPp9spuroW7Sw7kEZiuDdFwlQWtEv0jhzeJ8BUvwk9ojrYA8Q3RzZF+jB2AGc2RRE4s5UdcQJVbXHF9tuKPICGYO0oOMaV9OpUkTJHLWQzQ5sh6gx4eJyhN1RVQ1T85usmEGlL6miY7K18xdxdyVjOophc4ZVhMuDq+xX465Nd/n0vQmj06e4uKsRtUrSJcjmgxRZog6Q5YdZNlFzQfIeoX3TS+zU43YrydUviHE0LISgRQSLRVWGbomZyXrsVWs0TedFEopi5EaIxVSCEKMFMqyansYmRhPBDazFVZsFyEEr/21z4sAX/BLz488533oQQMhEL0gVCaxPRXA1oTuGDfYxnfGBFMSshnkJQtgsf/uRLSvPxWz151dagfii45/HB3hU9vt20cxl+9865fEkytHuG/zLqbNjDe88tV869t/GXZuHrxJBN70x7v49z4B991zsLOHxjTvXIFy+vGPKmUbokSkCmlSKJ8mhopI4wmDDrHIU3gRY2IjUhIRCBFaIGl/ivZ6B0mMHDCVCNHLxGjKMmke0xnM/oLOIiQUXSiKFMJAAp5q/smNpmsSIzI6TXAdIKqksyASa5ExfV5YSi0EkZiYAGk8IoSlvoIKCOPAOIKdI5zh9FrkOasPIhCcG19BOvDdIYqVxL4WoNuOiyy7EEQLTiVBNiA8Wkhc9AgEM1dR+hotFLGJZMqykvVogmPuaurQtA/HZTdEii6y7iXtq7bgdBrw2iS2KCLKaYhw2WyTKYOVhrWsh5IKJRU+eowy9G2Hytf4EOiYnKPdLSBilKEJjsrVgMCFQOnrg2Fr6etSh3EVLvjl5Sikoj56FbvSJ046+GlOqAySxGijdETZELI5Ilhk0yHYCgDzw4eiECaNofKYHzoSxfYGrK8hvu55UXaSPlj/yCOfYjgfY3sauPzcw98dD3U36JiCL+hugO3y1vN/grr3KP5PDsDlCzefy2+98bE0+ebtJM07yMkqXDz/8Y8oxDKkoVMk7PAS4VWabCIgTRIro1DEKJcTT8h20qmQQoDbJypACIiQwGQBKiG0guqsZSzTGdR/ATSUTiFMuw8QYMwnDy4xJoBxNu3H+1a4TbqQKAwQEcsBiEsgjKEVqkXLwlRIwKJ9At5GE2UDJnA8P0rPFPgYkxgbJcJLgq3SGPn2mMT0M7IEnHYoIRgu1Pts6Zz9MKcQmlXZJZcGLRWxTpM2iaYlVajZrkfLiS2CIiqHbLI0AfMKZIDQ6maNBi+RqsM1s826npCrjEJnDJSlozNmTQkxkmlLBGrvEAjWi0H7e0PtG3bLEYXOKLTFR8+4qZAI6uCY+hKBYK+ecKXe4aab8+Y3rMSoao5nXa5FSSimUEzRnT5hv0+oDGowgShQdR9Z7HHcdnkylghvUD9WRKE6S/CPEaKpkP0ptqtSCDXqIWxD9v1n4+I6Vq87/ymguW1bgsvPPfzdEeCezTPct3knb5vc4vN+9POQ14/hn3x6iPPND76G33L/EgB7OlI/LmFtjfr62ic+opApJIIUNgSRGIdTLWhEog7Idq5FL5Ku8lH7aR+L6RJu0168JHpJcDIxiXmZwqH5DKpZultu35RKoBdanSWG9LtUabJ8MltTg89SWKR12ufieAfkuhUWxZJpHYBLTAI0IGVIWbJRFyEjerxBKKZcKbfpmoJpM+d94xuouEJUDhElKJ9E1aBSZs0vBlIgGoOUBVGaxPpcwbZzQJex8AzVEIEgisBddpNr8x2kkPR0kbIy9S0iAumSWB9VWvVRjth+OBEUwhmwEhHSoiFchw/O0uKkhGyBIsPHkt1yRKYzfPAIoGsKjNKEGPHBo4Rk7lN26mh3E4BhPcHFyDzUzHzFbjPhqh8hXYGZHyHqmqgark4lssmIuuH4SsMlcxPtFbFMTEuWPVAeX/e46IegIs33TIT6sSL6bA/pOoigiSKQ2Tn9Lc/uuAMiJj1BRKJ2yzE3P3QkyrKAIKm+/4l/8ECzBJdB1sMqw3+dDQibd/LAG78aMzuWdI3RbeCyvsXbLrxrORHdsAMPvgDTK2ne+8QnPuJCKG1DnRgSgAglkxApW50kktLHXidKv1zhIyIIhJct/W5DgCVjEcQoiU1MoU9VpzBoXsJsegAgt28iMYsDcGnDMG3A8ckBTPAJWLw/COli2mdiYfE2XLwdDNNnFDJpRf6WxxwVCBGRRY2f5MhJF+kVF8OUC+MLCG/RfgOILZNoCLZGOIsIIe0vipSujgIagwySqHzLMhIYIQJROYiKqGfIpsd5P+FUXjD3FfNQMQ81RIUu14iqIaqK41mGFZ4V0+VmPcQIxSzMuOFmHNFdbtQNIlg6tqSMnkfKK1ShoWcKOianawq25/tooeiYfCnqlq7GKpNCpqzL6cFRZk3JpJkvtRYXk36zZnpcq/bIhaJSNcGURDPnrqLPPFRctjd4TnGYM/076MhLnCuniDYEF40hRoEMfdz3Dpdg8PVnPpPr0x1ulHu8Z/IURMUddoPHqptIkxhhHNRE4VmznrPFUXJlecfocWiOQxRk338mVt934R80wCzB5UhvC6MM8cTzkftX+NnP+A5ijHzo1jl+XHua3x+nCbcy4Nr0FmxsQuPSpH/8AuKhNRjvf+IjxpgAJraeE+eIRhGjT3qEChBASJHSs14SGxIDaSd/lCKxDW0RIoKE6Np9Owehndxllbwj8xLm02cGidbjcQAC7XOq9Z801Sc/qiEcPBaf2SfNKLGVNhyKEJ0k1G3KOUaiUvjMwmiXJnSxZwTNbneJyTIIZGWX4JBON0BeETs1UVUQJKJJK2xUPjEaSMcMEoEAJ8DLtB/lQWmUN0RvCXoORJ6sk5AfmSMQiGAIJmXYhKrYtIfo6w7r2YCz/ePkKnlcINIETx0SACih2K1GXJ/vMAsV7987xwuE4PzoKkc6G1waX+fs6gkybVFSo6RiUk+JMdI1BR2dU7oaHz0qSv7on3xYAHzef35OHDZTjmZrDHwHhaDXK+ibY6zYLkalEK+rE4jNQ00UmljMWoalEVFQfe9lAaDfsBLvH/Q4kq+zng84Jjd4tq/588kue27CEdWlkQ0rqsuWXaFnCvqmg5YKIw2vygb8Rv1h5LwPCOy/PRnrf3lJfOkvf3p885e/4x8c0CzB5eTKMVbzAW99/A841j+EVYYYI3ev38F/95I9fv5cBRcvwGzGyf5hkOega4kXrsJwF+L6X/6ooWUGroHGEaxBOEWsDCIDQlL2Q62IpYd5lQTZqkrpXilSyKE1caGVCJYMYflwLmVuyjKFK8+0iYX/RaSMjhCwswu+OdjfJ7v5Fty8P/C1tOcZI8sQDhEJVYDhKH1GQfLJ+E4CRgH1xR6oGqTCzyXBWcRtt6lQSaMSKiDLTgKPoBBeJT/M7eJum0HCLdieABRIhcgrYpvKFkETVYPwFhEMUdXE9t9WJtgyA6Y+43K1ja4UanaNOzvHWM/6ZNpipGLSzNjIV6h8s8w+geB4vokSimE95UR3CykkPkZuznaZ1AnUkmgrMEpzbbpD3Zr5XPDslCNe9p/uii4GerqgUJY6OLLo6aicvinIVYZVhp7t4IPn0vg6l2e3qEMDmJQtExGhMprvvpGA5UfWo4iGG80+R4uNNixLmS1E5IabI6IkSsfZ4ihWpqmzX0/S+5Rm5kqeNejzISbIuiDYGV/zq6+Io2bKy3/+/viHX/PoPyiAWYLLtJ7x6K3HeWL3Sd51+f1cn2wTSRf056/8IfSfk95oDD/42d/OG971Lpq3l7CbPDCxNpAVf0kRNB4wl6oGrQkinYpYCLG1Js6TgYzxJHlEmupAtxC0DEOn8EWnSYIQTweapv742auFW1arFmRI4PBM4dNfdnOuTaP7dl+K5YkHiE0g1m1maWcP9ncO/rY7SOddVa3Tt0wO5oW42IaIQqQ0dnCaUCtUFIjaIE3eMiKN6M4SoERaYBGtoxlirRF5Q2xU0rlETGFVy0wS+AWiqhEuTzqQqhioAonkaLaBAN4xuoiIhiv1OaLwICKHVYeeKvDDwJrucaPe43i+SU/nZNIysB3W8gFdU3BztksTHNNmzpHuJuf2nmKQ9ZYp64HtUvuGSDL2zVzFxM3RQqGEbH0uoEVKV+cq49de86fitb/2yigQfHD3Au8Z3kAEkzJ3ukp6FxClWw67LLtEWzEOFbVvlin5mS+JIiBiEtRP6lUmbs6tMCQTBiUkubKUdc2j1TWImn4uGesZa1ohEGTKMHcVX/zGF8db5T5/8rWP/4MAmSW4/OS7f4FMWRAJUBCCEDzb8z0e7JzikTvG1Jc24PJT8L0P8ZpPfwW/8HvvAyIoQ7Obw6FDcOlJlktrXP7nYFuEIKE1suk0gWK0+JClSeQ8lPOk9YzHMBl9NIOIQPTtfqqn71/pAy3mdi/LR316AyZLk7hlQgBsbsHN63+F4bxtv1onJkTLslRbguBJrGRnL6X3/+LnKmdQdQ7CqoXQLE3y8LQMJJkDgZhCo1BrpJdLj0v0EmEM2Kb1v4RUIuFTaIbx1Nt97KEh2BpMMr+BIIo06QQy8RUzQ7oC4XM6KqejMozUvH30YUS0LdvJU1glIrdUzU0xQUTFPKs4ZjdYMV36tsPV6TYvOfoARmq00gyrSQsMkpuzXbY667jgyI0lxIBsXb1zVzGqp9wodxFCcCRfR7Telzo4Ygy8e3oBERX6R9ZjMNP2vEx6uGyZmo8yZSNFFNgfPBpTZi2Jz3WT8cHJk2zqAfNQ8VQzQsaszfh5hn7GQBdApI6OnszZbyZcqPeJQiB9hpMVUTqGZcHDOx/h+Rt3Y6VhVE8JfxVz5t/TbekU+6kv+i0xrMZoqXjo5Au4a+0kLngOdzd4YO0sMStTerbT59s/54v5hSt/AOMUk8sXPwCzOfL4Wlr9+6tpBS66ic3YHGyWskRtvUhyybqkNSzs+LMZNB7G0xQqjEYwGX9yockiHdxULdP5CxdTiAQ8Nm+BxST3bGaTM9ba1vT2MSsjPsGIttmwTpH8LSsD5MBiVht0r0QVLjGlsnzmz+Ua2NtbOnPT+RmEFqjMIW16CLXwwgSUCcl8qNrQJwqEdaBCMs3ZimgbyOr0PBCmWfIYGZdYi2wQQbcP22aNGqJM4WQwE6JwaCGTziAkus16pcma9rMQrgRwIjc00XOp2ubS7CZ3DI6yVaySaYvVySB378Zp7t88S+VrZk3JXjlKZr6monI1jU/n27MFfdvFEzFCU/mGYT1huxoybmZcr/aQCERImUgRFKK9vUV4+rUU3rSit07nvHg4gx5vMtpf5/yu4OpEoeoVhMuXGbdZdLgQyKUlEilDzS03SpfeZ0TZ0MRAITTCZTw6nDNrSiIRKzVdnX/MW+eL/ssL/39l0Hu6z+XVvyd+4I9eG190/Hl0bQeB4Dsf+mnx7b/96tj0bsDa3fCq6zxr6074d30oIgz6CBkxxyTNLQcbh+DoobQyV9Vt4mYbCnnXiq5tNmWhebj293mZdJLptJ2Af0WkXwoT4sCshkisQi2YioIsS488a2t+gH4//f1w/y+XKVp8BiGTu7dTpP0VObIjUd0K0fpA5KSDN+YgBLt9kzKN1amTCeAGfURhk7FOJQYiFmEOKY0tbktfszAUyogoqgQqpkrhigVhTfLAdBSqMUTTpGygbkiqeKu3mBlB1py2q/RUToyRQKSvOwxMhxgjw2bKCbPCk80ex/SAjsySNyU0uKjoqz6FstDANFQooXhs9yJHOxsYmRy5UipiWyuUKcN2OUQLRaFX8TGgpVrWI4UYyZTh/sEprsy2uVJuo4Sijg1N9KyoDkdUj2uhRAifDH4iHqTkEUR8Oz7pnvK6QsSUXRTBIKo8aVa+XQCNRmiXsmMipZt70izDsY6UzEO1/P8ZKXw7pHtsmRXGes4dnWP0TMFuNaKJT7+XvuRNL4m/+hXvEl/x5ofizFVIIfniN744KimJMfKrX/Huv9fhk3imquifeOe3RKM0mcr4uhf+kLg0fEs82dvk9P/2Kn7u81/H537lP4fDR+DIIcyZqymlJwPN22bo5x7DTWwKaap6mQVZrtIhJD0ihjSprV1a45OI4GEybf9+9lHn9vE/jVwWBS5DIyGSAIxon1fpeEqncGgBLNYsdpLOYTb/aD3ko47XljEsGNAivMosdDqIboYqamRRQV4TpUdMetR7Hbh2KwnHrkljozUUnQSoWxuwtobqgsxcWjVlWIZDi3oqvFxSfURbTqA82IaoHSGfEvWcIGue3znOB2ZXoZ1IarJBKMYIbw/YjtdE6Qn5kLuKARt2QKEzjNAEAlJI5q6kqwsCkXE9xcWwFDcDESUkXZ2jhWLua3aqIVNfMvQzzuSHeejY89jsrHG0t8WsmeNjQLUh7NuefDd922VUTalCTc90yJVNldNS4oOn9DXjesZ+PWHmSurQYKXBCE0Zas7Pr7HbRKTrIJ0Ff5tj2dTJAyTDssAySo8IChaszWvErAPaEW1NNBXeTOibVCDZ0wVayGXxpIueJjqs0FTRIYCOyjmSr9EzHfqmg5GKUT3Fx4gSgjd/+TvFV/7KZ0YlkkvZx0DlG4xUSTEgEGIkxEATHDFG3vpVH/h7BzTPyP2/9aU/KQD+94e/JzL8jXhS5/BfvpaTxRaf++XfBmsb2OdkxLJETLvEYpZW/G4X2S1hZBDrPeLcJaAIyxxvYi1CJFBZpJSFSCFS07T1PyU0n2S1sjbtRDcHIq28DWxu3xYC7iIksgZhuK3eRxA7fYIx6XyeqU5KmxT2rQwSmCzAi9gyogXjCGlsfHLRJonK41cG6fhl2UYSAQb9pKsMuqlQ0brk/VGBA0FYtq0mWu/PohQC0nu1I9oKgsJn+xgheGn3LJv5CoXKEELgY+C99aidZKlWSTa29b00RBFYt33WswED20UKwbwtD+joVAOkhaRQliZ4muAofY0LDT1dsJYNEAJyb4kxEuvInpswDzWPbJ/j/vXTzJo5R3qbKCHp2Q5CCNayQcroiCTQxhjx0VP5mlxkSCGxyrQTVlOHhnE9wyqTJnXICDEQucE+c2LQiNg6lhHJM6VTRX26KCpdk5RoT1qVdJDPQcZUb2SmHM9yjmRrrJguhcqYuPmysBOSRmml5khnI1WW+5pcZWzkK+Rt+OdjpAkH+t+pwVFuTneSwB8jHZ2lcyS21eE1TXCEGBEIPvc/PzuWrmLqS97/2ksfF2g+5xeeFY3USCH57a98//9noPSMzOVp2zu/NRIjmBzxH38XHnkEXvAC7NaU+orl9MuucvVmD6osZSB0IJQGIWPqrVLLFlDaCt8QQEukjAfGOE8KhWatPX84+sT1SbdvNk+rfmYPhFStD1LASx9L+1kXz+vEXqRJ1clCBdABoV0iMNMcd8vB1etJYK7LxHiyArod2NxADrL0GW6vyhYSaWPSRoxLLRBUS8kbTawNfm6TgzhGdMfhpgqhBbpXIrImCaymSZmNZfarpexeJdYSWnG2rbNKbRhUqpruDomi4VhuefH6vXR1wY3ZLkpKDhXrnBtd5h3DCxA10meJwQRJNCUyG/M5a89hM19Z9lvZr8ZcnW2zXY84mq+zla+ipKJyqY/Lfj2hq3P6psNmsUrpaob1hP16QulrrlY7uOhZUV3O9o5ipOYf3f1yzu0+hZGaQmc8sXeZOqTQQiDawsSk32TKLltA+BgS6LiaqStRQibQDJ7aN2xXQ949vYB0PaSzScxtLfpRLUKTuHAy3nYjpawZLZvxdsK9xTpb2Qob2QAtU/1UEzyNb1lL8JxdOUbtHYe768vapto3SzuHFJJJPWuT+cmlvF6sMK3nVL6m9g2qrfPyMbS6UxqHutWcmpgYjBCCTBo2i1UEqWQinUdiTVLItD+pqHzNWtYnVxlv/NI//jsHmU+sWlZjuPp+vjCCvXOfeOh+mlueeruHePARXrj2Qq6cH+GGBXF3iDljkb05NBoZWudtSLRemibVymh/4LmIpJu6UTR25aCJkiAZ3z7RlnWg102T3doD/WPBTm639i/ARUiQom3J4FP1tG7reGxDtPXyfI3u0HTOwHCcNKS2kptOge6DyOfQlh9EL9OkJ9U9pSZXt1v/RaoXsg1KBRQkraQosWukldPWBNWkojrVenNESLQ9KoTLkvnL2eQejiIBV9tmgSBaANLIaLhh9hjVU071j6BkMtAJITiUr/Icd4Qnq5uMqBAiFTAGPeeFnTvom4LKN0ghMTqnCY4PT6+w4xxjN+MPho9yf3aEzWwFIxQdnadwqZ1MD+98hA+VN7gn22TDDuipgkv1DoHITjWipwved+1RLo1vLKujU8q2xkhFppJfxkVPiBEXPTGksMtH3+pAqfZpWXogU9bJNoazZoN9OWVP1siWkQhvWsF6oXe1gnQ7xlEGkI6gKoSU3JH1OJytspb16dsuTXC44DBSYdvj3rd+mhuzHe5ZP4ULHi01ugXdyjeM2wxRz3ZSL5p2TJWQdG3BbFYuwz4VFiAhKHSGFopgAjEmLWvmSqw0aSxixKokvHuXfs+0XYJRjJFCZcvw6vV/+LXxf375f/o7BZiPCS7f97Z/HD/z1It4ZVPC6AP83489B47N+WcPvoCfeMslYoQnvvpNnP3F/xYT7idevJzSrXZhEKOl6wF0msRCe+iUROVSLYxX7U+DaAy2WxJXLU3vKOwWcO3ax27mpE1iLJ0i+UD63RRmtNXTqOTfWKRukwCaxlYIUjghSOUGbZHgQq9AN2n1z2bIrMT2cjhkiE2XWLd+HOVbhtF6JZZhj2x16rjUSQ78/qTfjUNkCcCibojK8+ChPn+6f61tuOQ5pVfZtFsMTAct0iS7VQ350PwGsukjG48Q2cFqHAVCpuwQAmRdEO0c3xS8d3SOjs4pdJas8yGtdoXKOKLXUAzZcxVEjZZJQxEIrEriZeVrdqoRu1VqV9BVOcLN+PB0SK4s63aQdJa2NcK54WXOXexh+pGPiOu8zK4w0B2Ox8DVZo9hM6X0NX+28wRF6+o1Ud8WBoCRAa00uciolyzBUbXhRekUIukAACAASURBVIypsNLHkEKvFmJccGipOJKvs+p79Oohl+Mc4bPUliLo5f25YDPLWtoWhKKqeCA/xrFiEyMVA9sDIi54lEhNtBY60cmVo3RsQaEzXHD4GFqw1NS+IVOWxjc0wdF4h5Jy2WkvhIAAVvIes6akZzNmzZy+7dAER9cUFC2wl66inqbxSQJ3Cr+uzXbom2KpdykhsaZACbFswnX/xhk2ilV+5r3/PPoY+KYXveHvBGSeEVz+2W++Kg5sl935PnzGN/EDtgOPvQd//hQ/Kd9O5Az/4kvv5mcf/mXushtcdAp9/+Gkt0gPThFrncIE2usoSKxAJmDxxW57bUNrT0+2cT3bxIgVGrmewqlrV5PAKgRom3QVrROQaA25Ta0SjEnAhk6d2aQ/SDFyIIcsN8HB67dlWaL0ROUIekZUNcEIRJ4DAeFyZJMvw5HYFhYm9qXSihglItxm5FvcxLdTcBlTcZ1u8HbMXfkKn3nseXzWsefz20+9gxXT42hnnYHtsVGsoNt6m535kOOzDZ6YXONCOULKbnvctq5INykFW3ZSFzrVAJFxmfGW7Yd5Se8sHZUhEMx9xbiZMQlz9pxvU7gR7yw3q32KVjOIMWkAe/U4+V6i5vH6FtJ36JqGDZt0GasMRiZt43233gfqBFEGjusBK7ZL6esUIiBpokNH1eoj4KKHEDEtGyh9zX49QQjBatZPIBclw2q6DAUgaR3DeoKPgUwZOjonApm0S5FXCYlnh+tuRHQdRMySn+f26bU0ZiZwucce4nC+xsB2UUKipWTmqiQua7PsRxOIuOBYyweUrsJIg3cVpa+YNyXjesqknnPX2smlcK3a0G53PqRpyyMm9YxMWazUzBAc7x/i1myPxjvWMstGscKsKRO4+gYtJCcHR7g+3SFThv16QhM8hbJ8cHSBLbvKic4mYzcjU4a7N05z7+ZZPnTrHPNyzM8+/F3xtS/893/rAKNe97rXPe2Jr/yVz4y5ztBScax/mN9/6r1Urua1L3sp53kH25fWUJ2al50+yg8+8puM9lOvDHl4J9mjp6kqtBnlyYfRCmXL/iRtSCSiBjPn+d2TnMrWuLOzyT3dw5xzTyKlQQVNoEhCr9LQ66cU8WCQ/COdImVnrAVrEVYtwxupExtJP+NBcyZ126NlVom93JbK1a6d+CWnsj6fsX4fm9bSU5KgJsxUmd5jEuOIyh2kNxfsZ1EYuHy+FWVlElwTY6lx+R6f1ruD070j3Lt+mn7W5Z7VU8QYONU/yqHuOuvFCoOsnwpLpU6CqukykIqroxTKRFPj8z3u6Q64xW5qOWnnSZxVNQiB8BlX3Dalm7PXjJm4OefqXSaOttGUa6MEy17T4JmiELjokyvWl9yI28hoAMl9nVVetHYPG8UKXVOQKUOhM+rgyDGc1+c42+1wpjiSOtkBkUhPF7iYdKSezunoHBccIaaevj4GRs0MFz1aKHJtkfIgbPLRM/epHGG/njBqpsx8ao+ZK4NsrQdKKrSUaKGwQnPCrnOlHiFdtkxF3774ANCGoqfzdVZtj47OWM37NMEzcyV928FKg5aa5x++jxuTbU6uHKXxTcpmtaa/aT1n2Pb8Pb16PAnOLZNYmAe7tuDy6AaidRgPqwmRyErWI9MWH9K4z11yDC+29XywFMEb7/iNW+/hoc0HKF2FlYZTncOpFsvXXC93+ePhOT5t7S62Z7vsz8c0oUEiuTT809c969DLv/9vEVuezly+/P98KObaooVECcVr/uD7id/0R7zyTf+Uf/X+N6KunubHv/QL+Ke9Q/zg9rlkUhp3kUWNqC1h2MWNJLIQ0DREkxomHTTVVqn1otLEIHnW6nE6KqOnC6SQjJopL1+7m7eHi+jaouoB/tjRlLHRqhVTDVKlODQ2tAKxTvU1+iDEWdjjl8vSQjy+rZEUcMAw2icXRX5ReNZ0amy0ng2WNu51XfKR+haiTekSFQidhFTR/n1QB4PaZnRuP5coPT7b59n5UQqVsZYNuLh/lfs2z9CzHVayHh2T08+6FCYnV5YmOHzwBNLESD1ub/H/3LpB1CVbOm8LIttixNv6uUQCMlrwGTdCkzSFYBBojljDIbuFRDD2M85VuyjX46l5hRG7DHSaUD2V89z8BC561myPM/2jFCZvqXfSWWRrhT/a2eBLshdjVMpYCCDXGZVv0sRp++nWrQh5QCMiRqoWZDxKSprgUFIh2s8cSRpCbPv8liExIqOSsS4qMCLZ7qWQrX5j0iK3YJCtN2hJLRfOXRGJhCWATJo5hc5T03CpEUIwyHrcv3Una/mAr37eqxnXU25Ndtgrh2TKUvmajskJBMqmagG3QbYZSxc9GoUPnrmrls/3TAcfPfvliEPd1Hb05myPU/3DDLIuu/MRc1dhlWG9WEELxVre5ws3X8Cbzz/GdzzvFbz35qOs5wNcPaUJnl034dWHXsSwmlD5Gi0186ZkUs85tXqM3fnvxZvTbe7b/Iq/FRajAeSPm/j5G89iYLv0bIfaNzx47Nm8/Np9mP/war79ea/gjx6f86HveT13bl/k+X/0H/j6B76YVx46ze+X24hxjzDsEZ0C5wi1hcmIYNeSA176dCEXPV7b7v0fmF3h0/tnl0h8vdzl0cn5VLmaVajNfWTWJTYpI4MMCD1PVcBAbFRbZ5PcqELGZTr2aSsSLI8rnDo4B1h6PBYp36gbUI7DuqCJqZ9I1lbX9l3NdjXkPiHYc1PGoWQeGqJM+xRRpdxGFAlkFg8Rk2AoPEjPppUM1CYdlXOqf5gQAycGh9jorOKCp2s77M6Hbd+TnCY0+BCQUtI1HWrv+Knzb02hjEoh2nYd2A4zpO+DV0hhU+sJ1aRQrT2PiFta4o0pOZlvspYNkAjWfB8jNI/Nd5BNj3PVLg9IixKKQlm6piBXlr7pYJVFItrJkVbfEFMB31o+wLVV0SlLQvomASFQQrCW9RnV01YjUBzurDFuZnR0Th0cfZOEZyuT98gHj5BJpLRtM6vKN1ipWdFdJj45YJvgyJQhEJEtCDUt8/rAeAcRzcH1jjz9HmmvESwT02wWqxiZChK1VHzWqRehpWI1HzDI+5SuYlJNGVUTpEilElYZGu8w0nB4dSPtLaQwbvG+jimWx/AxIBCM6ik922FSTwkxcm54mdLXVL5mWE0ofQKqlazHE/uXuW/9NP2sx12rJ+GpS/joccFTupquzhnWU641U+7fOEPPFoyrKb0sic25znju4fvb8grJIzd+MT7n8Ff9jQOM1j+yFjGC/Sb1TPXBE2LgHZffz5ff9/l878vu4Afe8TN89YNnufPKB+HIfXzoRuAbvuY1/B8fegvRVAh6hEYRGgV163lxqVH2IvGzXDEWACMiIhhcdO3qJjjdO0IhLe8cXiHYGULrlM3x6sAI1Xagi6TakIX7Mq1KMekgzwAsiVGIZUZGlHnb3kG0YU7dfgXHjEw77u7cCZBaP4b0NRhSS7aEIG8sWigGoeBys4vHsSoLbrp5e5+mFVbJgA+aKBybKsdKzYnsEIXK6OqME/1DbHbW2hRj2xXfN8ybktJV7M6H9ILDtt3y9+ZDXPD89OO/ngxgwqcxCLqtYFaJNYmIqApQDqENUdcEM02d7FgAq6AjDLmyrNguWqQJO/Mlur5JcCmlqtvVv9DZ0tCWa4sUYlnbE4nLBt8hBlzwRFJVsYue0qdO/UZq5q7Cx8jUlaxl/eW3DGzmq0sxFFg6cwWpRUWaCAqtko4iESip2cxX2YiBsZvT1TlWmqXxzLWmu0P5Kkeafa6XzbLM4cDrsrhFArSscCEOL3w2jXds9bY4t/sUn3HqQdY7q9SuIcbApJ5yuLfJ6dUTnNu9yM5sn9xkdG1B45vEwERiYMNqwuHuBvvliJ7tEIjkKn3lSunqNpSK7FVjuiZfmhOTp0ZTtiUS58dX+YwTL2BST5m7kpceXuVHH/1dPnvzNONmRq4sr77rFbxs9twkGGd9cp1RuoqNYpX7tu7kjtUTfHj7CR69dY7KVTy+8/q4M9vjv3/Rj/yNgYwGED7nw7PLHM7X2CtHy4v7xN4lXPC87aH/gf1jD8DuJT7/d/4t9Xf+Emxf4NMOPcD7Jr+HZCOJt7v7aSVtXHK9CsFBod1t1msZ2tRfQxM8ob05Q5vHD7JK/hCfI6RLKVivD8IN2fY/hRZ0bmMhS7rLciKJ2PbRFelrOKJuwLbhAzGJn2bOim04mx/lz+ZPUYW0MjbBISRUvkaJRNkLZenplKo9kq8vJ9adMaCF5I+HF4nC848OvYgL42sYqTnR2UIIQdcUrGZ9NjurzJoSHwJbnTTuISZL0cX9K3RMzrSepXYCyiCF5N986I2c1APWVY/gJ8yEB9rCvDpH1DZ5Y5QnFFOCmRPMmON6wECtIoVk6ksu1vsIIvvOM3UltXdorZYp3iaGlL4VvgXCpI1YZbAyTe5I8pvQTsIEKoE6NJSuXhbo7dfjZcPtRTbo6nybTCZ9JBKZNSVWWZQQaKmXXf3FAlxI98Zi8i9StRGQQix77y5eU21phat9qjcSguPZBjv+Keq2wllE9TSBP4FLYp8Lz4hVhnnro3nVPS+ndBXHBocZVRN25nsIBEd6WykFrTR3b5whch4Xkha2X46IvmHcTGm8Yz0fcHO6S9cW+OA50t2gZzqEGLg0vtGK4glQJs0spZPb73aSQlKHhqvTW/R0wWM7F9pQTXK4WOPeesSHxk/xwtW7WM8HSXdqQ1UfPVvdDXZn+8h2bB6+9gjXxje5Od2haT05TXD85Lu+NX7LS37ibwRgNAiEN+yHCTvViM18ZZmKNFKx0VmFW+dYPfZsfmn7HJ918oXwc6+As/8N3/jgV/D2a+/n8d6EeLPT9oyVbfsCi9Rtv1vdip0tsESdOvqLqJn75Ebs6Ixpk1b+E6a/pI1PubY4sukmMW5RhBZlEivbYr1Ia+de3iQCokosYhEWeN2GKSG5WNsJFM2cDSs4ZNZZz/p8mrorTaB29ZOINkWaVpBMGXqmoGeKZSweYqDQOZWveXFwHC7WOLNynJcdf16a1M2cXGc0vmUPxKWl/Y7V49y4stMargQPbN3F1fHNZVr2g7fOMbBdnl+cQAuVsiPScLXZYxobojCtpNMyM10T7JTn9DbYyu7ASL0MG0pfs6q7vH9+Gdl0eXh6mRAja1mfSTPnWrWD9DlR1tybHaZnCga2S6FzTDvxF5mSRQo4tOPkgqPxDWVrqksglEIiHwN1cDTBMQsVK6a71GMWzbqVSBkn0bKixb9AaO+GNjRSBqsMPgRKXy2dsokhC6wwSJGYUi8U6atITIfndU5xsx5yw42Yh7TYDaRl5B0y2HS/BM3IzVowS+fuomdaz8jbsoa+7TGppxzrH8Z5x9yVS2YQYuRob4tJPWNap/18eOdiYr5Cps4DbQau0DkzV1K5ehkCH+ltYpTGhwT0o3rKuJqS64x5mUTbvunw57vnecHWPQxMh3lT0lEZz149Q64td6+f4mhvi0HWY1wlYLs0vJZCfCyXhte4Nj5oO+qFZHu+DzFiteH73vaPYwiBf/O5v/TXAhkd9CwJs1EwcXOyxjBoVxotNYe7m3DoPvjzt/J1v/kmqm/9MRhdh5uP8ILsqzjR2eKx7uOgji7Tw3pdEmqVHKrWpS8xW2RJWgYThSfKhuPFJiBwwTNpwworNGumR6EyTnGIiZvzAX8TyFO61+slQ4m6IqiSgYYTdhMjUopz7OdcbHaTBTwYYtQI6VKrSRYp5wqE57AuOJ5t4GKip7oNUXKdLf0CySMik9BqDybGoh1joTMm9YxhNeH0yjH6WZfGO/pZL03CGOlnPWrfsF8OscpyZvUET+xd4sroBludNQKB65NbHO0fYlLPuHfzDC443nfjMS5NbrBu+2zXI9ZtH9Wk4rnHqhsELaCICFu2Kek5d2RdBjqliA86xEETHD1X8VJleef4AtJ1eP/kJsyuJFD2FpBEPeNItrZkLQu2kiZdO9EB35rcUjidOvbnMbDXTOioDIlsF5CGJjr23RQpBHVI9nkrDYXJl+xvIQybthNgAjC5BBZHCtdc23N32Go3AFbqtPJLAJmMbjHiCWSt4SxTlkN+NYV6KmPmK2au5EJ1g3GTFtoL9Tb3cnI5Xt3W8PfZZ16aqrkjrOUrFDpnTsmqTnaB2tcc6W1ileXmdJdLo2uoVoTOlMW0IV0ywdF2sNNEFZk0c6ZNiVWaruksSy7EckwEg6xL1xTLrNLF0TVeevS5TJuS450tMmXxIfC+G49x59op+raL855Lo2tJw2rbts7knK7pYJReGgMXDc+v7d9itxzRtx2+63e/NP77z3/zXxlgdPi2Uhz5X3pxFOSyaU9HZVgVuTXbY2e+z/bRB7ghJW7lx1PGZutuuPg78L438z+97Bu4/vs/zGNH94irralr8wZ61k029daWvujbuuilEVXDabOGkYY61OQ6Yz0bsFON0EKRSdPWZAS0uC374nX6Zrw2BIq65ojJeKB/B4c76/jg27y/47jfZOZL9popt9yISUtXu1Jy1KzSVaupXkRnzNtVZ6UVtQc2XUghUtah0JZCpwxOrjOsSr6Qvu22fV8rGp+++ye2FvXFVZm5klMrx1q3pscqu7SGG6l4dOc8Lz/1onb1T5rXvZtnIE13Tg2OUO6lr/3o+CrVCCHo6JyeSs2z69gw8WULzEnXWWRWKt9gVZp4mTLoNiX6afE0756eR7ouuLZwUwQOZ5LTxV2sZr1l+JEcsgd6RAo3WLZEoGUbxMitcp/tZshOVaaQzecHWTgp2MogV1krgiZQEC1YR5L+pOVBIjPGVCRZ6JxZPaXy9RLg+iZlGhufUuk+BmSUeMKSFVli6ofeMkMrdav3aBrvqENDXxecm13jRl1zWPfbey7pRqdXj/GK0y9JhrlWjK18Ra4zjNKp2L/9ihYjDVfHN3l85yLn9i6j2gZWhU51XS54QvCLvOHSqKiEREgoXc1K3mdYjnHBk7fpcCM1pU/36Hh/xkrWa3UnmZpulXt0TcpszV3Nn179IIe6a/gYyXWGj54ro5sEAvdtnEUwR7oUMs1dxbAaM21KhtWUrsl5cnSdTBm+6S1fEPu2y/Z8j17b0nO9WOFwd4NvevGPflzgWV7BvrTpYghJHRw2eParMQ9f+3NWsj6b3XW+7PiDvPHJd/Gaepqu7YVf57PveDEvPfQAmfwI+802dXSc7Rxltx7xgfkFhM9SU+e2AjWqBmTK2yt6uOgY2F5KtypHoSydIsWiu9WIK+UO81AnX8zilrrNnxBFYMuucLS7wZmVE8zcnGxmU6Gdq1iJXQ7la5xtaaYQYKXhkdEFHty4l1lTth3MLIc6a3RNh77tUCwYijyI+2lXm9V8QK4zNjqr9G0PgNJX3JruMq1nzJtyWeuyqPxd9IRdCLc+eG5Mt2l80lSeHF5l1pScXTuJb+tTfAyoVt3vtGFJz6YYvRMyfAys2C4+BlxwbWo3TfIYA29/t23Ngg5OXeBVx+/kN64/SlBzvuzIS9jKV3ipuJNr1S5Xmn20kNxhNjhWbKYetG147EOqoYkkR+wiy6Da6mAtFSLCpcnN/5e1Nw+3JK3KfH8xR+x57zNPeXKqrKzMmicpSigKRAFRwEbEGcEJutWLNF7ai0JJS6PXbvHaVxpUrl6H1n5AWwUEBbEYqhiqoMbMrKqch3PyjHveMUf0H+uLOEl7+2nt636e8+Sw99k7dkR861vrXe/7LiZpyFOTTbnuyQxa6KLFahiZHZE5PhvGDvOJTyV20IBR7FNVQUI0RVqZyWgKf7ENiyiN1ahZs8RSRFekY+upwoDyUpOUl2xfCQCaoZfs5IZdExUy0HCqbI53mXKbXJlss686VwbV5y3eRKfS4lBnlf3NZUhD0C0qthxvmETyngrE3hhvc657iX44YqE6pYzXJPgkaabKPwlcQZKqa5XTcRsMowl1u0LV8vBjkQWsj7Y5NnNQzk2sseP3pAzMc/wk4vTuRcI0xjOc8hiEnJirrUna9KRwdbJNlucc6awSpnuY2Y7f48pwEw2NtltnR3UqgyRkx+8zU2ljagZz1Sl2/B5VyyPJUv7q1Pvy62cOcaQ2yx8/82kmsc+P3vFrZcAxAQI1PzjKE6mlVX2uobEx3uGRtSd553X38yff9MN83O9CpQ0zt8PibfDwB/jgy38ZLA+/0uLSYJ2rw022J7uc2b3IM7sXWBttYeoGH9t8glxPaKh0ddGbYhj71EwZ03qwtcRKNsckllk0cZYQTq4yZ7XYCncV1T2R1raWS4fHHFMxZmk6dTzLoWK5dNymlB/hkEkciMJU7bDP9i+xrzbHDbV9eKZDxXSo28InadhVqraHY8r7VEy31Nek6sLpms5iY46GXaPu1PAsV4nFIq4MrjJbnWJtuImh6UzV2zy3c47Z6hQAo2iigE+hiUepKMQ902XX79MNBqw2F8UzVl2iDI39rSWuDDdlp89zMjR0XcMorlOmEeVCNY/SmM9d2SY5vYi1uk38lRxME7vT5BOXzmNELY7PtcgUdtG266Lotdvoyi6hahX4iqTwSSbK5IKXEqTxNeCrjovFqcElOpbs+EbYQIs8tIlHNnGJJza6mWHWfXRAt6o85V/hVhUEkixVGImlWtsmuqGXwSZV2d4wlV218JfRoMx4bENmS8cqOy05Req1uqLMR6lO3ZAMDtUaNzSDplOnalfouM0ykIVJzIH2CrqmcWb3AtOVDrU4gCSk6tQgS/HsCiGCF5q6yUpzkRNbp2m5NbI8ZxL70oZHgmTNrpXfqx+O2FWL11AbR0Ggy/KMpttgv2mX3kp+HJDnwomJsxjXtNkO+hxuLTPlNdnx+wRpiKUb7Pp9VhrzBIlYwzacGrOVDuujbc71rigulay7QThmpTHPrt+TzCieqDneFtOVFqZuUneqNJwqdadKokb++okEQMi5YeYwJ7ae+4eZS+8todb8v508yGL8VGjOeZ6DCXnuMghHTGYOUXnwP/KbGyf59sWboTINlge3fC985HWc+Bd/zLFwyJGHf5cjs0dh5TbSo9/GM9tnWBtt0PMH3N+/i6+uP4WhG9StiuAUsU+e5yw35qjZFbrBQDgSacSU2+T6dJmNoKs4Ihk5SrFsiajxjuoKVdOl44pqtWZ7mCozmKlKJydMYyJVrnS8Jrqm07AroEC/jtcSHotTpapSv6p6n4JTkeeZuvl1pisdGk4dW6XERfv1cGc/25MuR2cOcWrrNP1wyHJzgUnsk+USCJpOTQ0Dy3BNhzwWJW6aycyec73LLKgOhPAlTBzT4s6F4yRZykNXHivT8EQJ3cZJwBl/jW63BbmOHtbg4hXiYAr0McQx0ekmZC7W85/l5M4MJ7unefnKKqZu4Bl7wcQ1bEzN5Es7J7i7cwNxFovrWzzGTyN0TbC5LM9pWBU83SbULcG6yHmyO0b3a+QTj3hQId/swmBENt0hNVwMK0GPKmR6wsVgizTPaFgV4jyl7dSxdatsbxfamFSZLBUYRfE8OQr4pQSALcWNKTo+RUu5AMJNXawcDPVnnoOfhOWUgNhM2JzsSiaYxjScKjU1KnZtuMGRSkfRLXQe6l9mZ9IjTELuWLyJ2eqUaI80A8uwSLJElcKSPTSsmupkyc7R1huygeQ5Bjme5aJpGqNoQs2u0nYbJckOwDaFBmBoOn6WyjoFVltLDMMRw2jMOPbRNJ1+OMIxxC3P1AxM3aRqeRzpCMA/CMfs+H0msTCPhdNj8MzuhbI8vWnmOqXuNgmTSIKRBruTPhpQsTxWW8vs+F3FiP5GNVH5r0EeY6shU05i07AqBElEYMr8YHm1w5H2KnT2g9+DW14jLvner3Fs/STv2j7LA+1VuOv74fdfiXHXv+RYZz/HWquwPMXJ/hodr8nmeIe6U6MXDJjKpbsj3AmdQHlZFDjLnNdhNxxw0K2zbQ0YxHIT5UbIi5o3oAGHmst0Ki3qCi8xlLAsB7IsJVGMzkuDdZbqs8qhXnZJx7CZrYnbu2s6ZelRd2rqpkLsFlUmB5TitTSTLGQS+1iGSdWukOU5V0ebJFlKXbWQt8ddGk6Nhl1lx+8RpZFgAbqJbZiMI5/js9fx9fUTXBis0/GaqjwQkZtt2Hx17Sk2Jl3Oj9exNMGiJmnIVjoQIDLXMSdVkunLZJ+riVHXxtbeJMltMb3SNvdLJ81I+eTZq7x4dUrKCwxMBRymecrR+j7Gia90RSN2k6G4xOk2lmayluxybhIqnpEuJktpTub66P402dgl743FshTAc8V6QplSaYnDNgFb2SWuS2eoGi5+EtJS17BI7TU0PMstdUtS6qgWuJaX10RTi7Ag9RXt6DTPSLMMV3fK1wzDMaZuMIomZTlWdO40hGK/GwyY9locaO/DNmzSLOFgexVGW5CEXLBcdiY9huGQUeSz2lpWnUDKjKkoOyqWiEYdUw0DVJh4okpKQ91Lhc2EZVg0nCoNp0bV9uj6A5l+adh0vCa7QZ9YCTRffugFBGnIJPJpODVGsU+cJuiaxhcvfx3bUNmgUlZr6GgaDKOJbCymy0pjnizPqCpJga7p3DZ3lIZTw48DDN1gqT7LMBrTLTg6CgdjtEUC1O0qq62lbwguxrvf/W70/8vKtcxiooVUtL0h4WEmtZypm5zdvcC9N7yMlz/z13DsFbDvDi6S0Vx7Cs49BK0V7s9iLtz4Cjq//Uqy61/F/V/+j6C5cOlRuPI4M/U5llZuZ2O8zb7mIie2zggj066Sk7M13i2JQkESoWkadVvcvJIspaLZNEyTtuWwz+7gGjar9QVuXzgGwGx1iqpdoRcM2fX7LDcXqNoV6naVhiudmprtMV+boe02aHtNpqsdKmpI11SlxWJ9jqbbUICrYB7FLB4RnWUYugSuOIvxY79kCSZZojodliKHSTZgmxa2YTOJfRz1Z6E3MXWTJE+5NLhKkESEScTl4QYrskXV8QAAIABJREFUzfk9rAf48vpTfOv+e/jDSw+yEY/YjH16SUIcO2oyoAWjOlTH5I/0Ic/3xtQGgZoRlaLPtCE1iBfOsFTVZRwslMrgv7l8icWKaFN68ZirYZfNpMei3aFt1WjaNSqGi4PJrOOw6FXY6pnosYMWuujDNllokfqmmtWkQaeN1cnQbZl5jTLQkm1CZzcb00+HmIgCO1EYT+H6X7M9ZQi1R9orSHzFDyof2Pu7Vt7Hhi4/RQbhWQImu6YtZZWiEqTqZxiNabsNfuLO72WuOoNrOqxoBnoSiomZprGjyeiTQul8Y20GkwwzTfj69mkcxRsx1PV3TZua6tAUHcYsS6nZFSaxL9opU3DPLM/xLIc7l27i2Z3zyp6hwjAcM1Ntl2VfmmfcNHuEUTTBNMTmoeAXFeW8WFYIDaB4rqAOiClVzOXhJsdmDpEo7s2SGi0kmZJD3akyjMbkwEJ9horl4ZoOo3DMmdEmy80FRtGYml3l78595N3HZ1/0QBlc3vOFX3930doNibAQf1ZbNxnGE6oKE/nU2pP8UZbzXRsn4PSDNFurrM1fT72zyq+tP8n7rzwBecbHLz3CQ0+P+PhCix+98AUIz8Hu4xDJ7h405oiymIrlEitaeJZl2IZFNxgozYWuAptRBpi6VcHVbZpWlTtmjzLjtdnXXMDQDa7rrFKxPHYmu9TsClOVNjkZYRLR9lo4psNcbRrPdNW0vwotr8G016HmVJmudmi5jdKpLVX09VyVM7lqt4ZJWHI8ojSSGTtqty+sAaI0xjJMxgrEFdAvVUbNGeNoIt4q6gat2RUFjoo0f9vv81z3Ast1OU/kOU/vnOVjlx9iFIu4UE9tMdLOLCUzMKAygUvLaEvz5BfXlYexihyAccuqAKuZDo0xk0tL6I1tTl4xaVVjvnxlzC2zLoN4win/ChfiLr0s4IA9TduSGr1hVfm7S5scaTVlNKvhcGVLg8jm2IGQjfUKeWyCBrprQtXDrGdi9WklStSpMo68GEhnkOYaO8mY7bSnJnHKAlitz5dZnK4J5yXPc0UGk+DbC4bKIlICj5+IWDNVZWOiApWUuXsAfYFtRKkQ/wp8xlISg9fd+EqSLEHTwOtelHIoCSFL6FkuaZ6WXZ2v75zl5s5+/vTs5/FMmVxQBIyK5dFyG7S8Bi2vqbRR4vGi62IcNV+bIUqla1qzK7S9JsvNBQ629zGOxmUn17O8cqOL0pj7DzyPKA0ZR36pP9M1TVToiP5KR8MquEGKKGkaQha0DYtuOKAfDLlz8ThN1ZkyNDG4irOknMCwNelyYvsMF/pr3L10M0ESMo59nt05y60Lx1gbbDCJfW6YeeEDZVmUvK2rARi/4eVRlnAp7zOXJ8R5wk4yZNZtk+U5dywc542f+QB/8Op3wxfez7vOP8wDc0cYTq3yrw2b+Sf+M3csHONFnUOsrM5y69xR+OIHoBeBDVz5LDQXufv5b+KDX/1DNie7NOwqo2hCy22w4/fIyanbFVAAWJEW1+yK9P91jSQVBN41HTpuE8swmaq02Z50ma1Oc6izysX+2jeQkdJc2n+dSku1luU9rXJHlBtRgDzZAWRAuqDvtmFJx4eccTQhUcO4TN0gSpRDPjnJNWY9RVvRTEIJLHlWYi91oyZqWjVvx08CWm5TvFnTNV64cjtfvPIYdy/cSETMKw+9gA+f+EuuEiltjKH0SwULWYNMR28PwUgpR8AdPijZwxNPko51dAc0I0e/cADMjAtxD6015tFRBjWNp/yMMMuxeyuY7UsccxeIculEWZnFhr9LZoY8tNYVQ+sEbtgfc/K0xYVwE2iKBizX1WjaFM1K9mwtcjGzKkbNarmmZlen5HpCkkecybpcirssmA0GGxNslQF+0/xxGUOi62RkTHlNTncvqYDckyCkG0y5TUbRhIZTJcllRI5tWmrGUU6uMpS4JEiK9YOlSojC8MrWTWy7ApoO4x3QlS9zfRZVDErJoWlULI8/O/+wMIVzaDh1uZfSiJpdYb42g3BNbJ7bOYfhNoizhN1Jn1vmj+KYDl+5/JiQ9rKUKIn4+LOfZbE2y3JznmF/jfnaNBXLI0ljBuFYcavGitgpTn2DaMyOAmZFuJnjWm4pqtTQSMmIE+lShsqu89XXv6RcCzW7wuXBVVUi56VmSWxY+oyiCfO1GSaRz47fQ9N0PnvuYeaq098wOuUbLBfe9bx3PvDAl977bg2dcR6xk42FL5Dm6Lm0qV60eD1bTpWD9/9rPvS1/8Lbv/SfmEyGeM0F3Ezn/332EzzR6/G161/M8x77APzwX8DDfyLEJtcCowmTPsdvfhWuabMb9MtFHKUJg2hcIuueKe3g6UqHudqU4pjIjOGaXaXlNrh14ViZnjadGtdPHyr9TTXkogdpRMdr0fFatNwGHa9J3alRUMGKDk6UxkRpzCTxpWOWCuM0zVJ81VIubAmTPMUyTKX8NcqsI0pjYjVl0I8DYUhmwuIs+DBhGjEMx0RZIvwIdUNP4oAdv8dzg8tc39rH4c4+XNMWkNkwuHv+Rk5un6IfKxZyZqhbXCtFksnaNEYlJN3SxfpzOITNLfHB2e3CzDSalqO7sbjWeTHmziJGVMMIGiS2j5bZJJUd5pUq/MbmAaa9JjNei7++eInf+ZY38dD2F5mEQkzb7hlkE5dwcxbDi4SRnRhoulheoCwuSl2TkmRomV4KPLVcF5lHLt8pyzV6qc+cXWMt3JFxqk4HUze5ae462m6Ttx95MWtpyFy1w4zXZsprMu212Zp0S76RpkB7XdPQVTlbwB5plor1hmp1G7oA2mIxmXHj7BFcy8OcdAVbdGpgeWw1F2RH1w2SLCXNU8USlo1wrjZDy5OuU8Vyma/NYuoGFVuUzxf7azScmsqyAm6YOcyXLz/Gye2zTFfanO5eLK0vgzRSm2uV62cO0Q369ALxdI6zhC9dfpzjs9cxiX2CJKKnMv9MqcalrBGlfdttlJynotwugt+Z3Uvsay5ICzoNxXw9Sznc2cdifY6tyS5RGtEPxBj9xQeex9XRFqN4QsORDpiuCwP5uql7HwD+xx66+m+4OVqGETZltII94CXtG3nRyh38/De9gSvhkFd/5Kd44rxB7vqk3pD0hz6M8QdvADTSn/pb+OA94AMTJHN5wU9CMBTAt3eFdOlmznYv0gv6XB1tc7m/zpnuJYIkxNB1alaFw519igHr0nBrjKIJaZZiGRZtr8lMpYOndDjLjYXSCtAyTLbGO8zWZvjQV/+In7zrB7BNoV7n5PhxSJRGRGkk+FIaEaei4p2ogee6MvcxdZMsT0mzTGmMdMyidlbdCT/2Gce+ONQnIVmeCTkpGBIpHKlgWyaZBJSN8U7Z0QiSkF4wZGOyy2bQ5c23fjeTyKdqV2g4NfrhEHJxhv+9p/6KQTzmZN8XcLSweVCjXEuRp7qBotMV9IZDNknBMrGaAdniZYzd2ZLgmNQ3lbARtNzkO2Zv5gu7T3F363recsfruTLY4KWHvpmHL32Nxfoc3/bR9/Dzd3wbZ3uXeap7lscfFaa1cfACuZaRn1tFN9XEAivd05Zp6rg02DPqukYaUrrzicjwpbMH+Zudk/J1tIw3rt7P7xz7djEQc6rgNiBPwbBBN9n2Gvzgf307NcujanlKD2VdIxHQSn/ZIqMtODuWYRIm4s0iLVYhtb3/5b+AHo4YGyZBEqIpfG0UCbV+c7xNmESyo9dnqJhCTyjK56KTlSlm7snt02r+dUg36KtswC87Q9t+j8XaDFeGm8xXp3Ath+ev3I5neawPNznXvVTOdtr1e9y7cjuapnF1tMWV4SZrwy2lELdpOFWaigzpWS6OabM97rIbDIgKE/AsI81FQvMDN72Krt/j0uBqqWI/MnWQZ7bPsuP3uDi4StttcM/yLawPt9jXWuQFq3djGzbrww00NP7mzOdljfx/RhYg+5lAA7D/3VKupSZZrvGZ7lMc6xzgnV/4T/zqVx5Em1RJezXytIF2eEzUWub2xgyPr6VMsoQKwBiIgBowfVha1E4Nw6kDCBHNqbKvucQk9lkbbVLQ6udr00KfJ2ehMYulm1iGRZSIN8VSfZ5RNKJqeexrLmHoBrN2lY3xFlOVNg218I/OHKJqim1DrMEk8hlHE8J0z4gnU/6moQoMjrnnNr/XDtSoKkDX0mWnC5JQ7V5Fqp0o4V6qnkswdZOK5ZU+JEkuBs/SEhzRDQaqpha3sRta+6mYLlvjXfa1lqSrldiqrZrxYzd/F1Ea8eYvfgA0B1JLzS8qm68qQ9DJ+jX0hoE5v0O61SYdxGhuRK5HxDMXyfUII2piGBGH7FmiPOZc3OVHbnkNs891cAyLPIeroy1WmkuMown9cMinv/tdfP7CI7ziuheysjHHibUvEZ1tonenQMuJMx291RcF+6BKjggFNTWIrRSwgjIcN/YErkW5ZOh8euOC2HlqInZ9bPs5SAIYbEB9Rn7fdGQiZZbwt2c+zxtufhV/+cxnsfXCx2WvVV0Q70wFzmu5ECiDVGYruaamwF6HSRzgWQ4/+8l/y5vv+j7Gsc/B9qrwlFS5DSIF2Jrs0nSllV6QH01NlxJacW8KIF+yg4h+OCRKEzbGO0zikKX6jCqTdZVhWOjKue7U1hnmajP4cSDmWplgd8Nowl88+1lunbu+NDqfrXYIk4i62pgM1ZGarrRZqM+KgZZpMY58FuuzjCMfXReCoYDdbSxD2tUa0AsGVG1Pyso859jMYTZG2yw2ZvkXh++DzdOQp2TTB7kyWC+nOfzPDbqL+j61IWrwiUsP88YbvkN2HSMl9QFdw7y4yrf98Rt5+90/zPd/+KPUf+ulpN/zB/DbPwhNYOX5sHCMzSxma/s0hzv7cUabVCoiAUhzAV93Jj1Wm4ss1GeYq82w1JijHwyp2VXhIlgVxvEE15QxErPVaRpuXUnXwQCe3TlHx2uzpdzuv/3QCyFLSHWTfiA1Y5wmBElQalkKaT8K0LUNS/EEKIOJaZiiX1EchhwwNFNhMfKI06TEbwyF7WR5pjoCQuF2DBsdnaolNonj2FfTLTtYusmRzn48y+XG2SOqW6VRs6tqJw1JM5Hx16yMgT5ENxzxIU5l9xaBplqgqYa5sEO60UFf2EIzOuSxKdmFHnPInuZ8mHLUXaBquIRZzM8deDEX+2v82O3fzV+ffpCGW+ONt7+Oc90LtLwmnUqLzfEOVctjymvxlrt/kLfc/YMc+MCbYNDi6AGfU8qB78WH2nz6Kzn6VJ+sW1eTIFMRrxqpWF4YKVpkl/ahWi7+MFquJkYoH5xcS3msOwS7Bi2Ff0QTKVnQQNf53s5BTjken7vwCLZhlRqkgttSdJlMcWcnRygIRUrvmg5+HOAnITkZ/WDCbLXNHz3xlyRZyksO3sMNM4cJk7g0v0aDjtcsnf5zoB8MSfOUnUmPIAml9FcmWZMoYJIIABsmEdNem8AWomeqZXTcBmkmTnwtp14yootsaaY6xYXeFVG0Z1KCf/HyY9y7fKuSJIikwNJNOpVWaZOxUJ+l5Tal+9u9yPH913Gws8oomqAh1g6u0sgdm72OJ66eZGO0LWrw+hxNt8448rnYv8K+1hL3rNwOa0+zPrWfBa9F6HeZqnSUQl37hzaX//3jF17wsw+85/P/4d2Fb0o3iTjemuP1R5/HSsvg8fgsZsOHxOTyTs7PveiV/O6pT2J0Z4kaI1585SmYacJN34O/cJxx5GMZFlOTLugmplPl1M5Zntk+w5cvP87aaJu5aocXrt7NbHWKQ51Vasqj1dANVlvL6JpGxa5Qs6tU7Qqu6aBpOlEaYhgWm+NtoiTi9O55ojSmU5siSGURj8vAIvIAXd1wILwDP/ZLTkEh9pO2nAQb1xILAtd0cVU2JG09XwHBaSlrd00H0zDKmh7AMcV0aabSpunVqVqi2nVNm4ZdZcprsdSYI84SZmtTKoiKs7tr2srVTLosy84Uk8mQH7nuWzjZP8VYV23xaxToWt0XNvO4glb10ao+eW1IbvrssxpUDZfFiodn2LiGzU/f8b188+pd9IIBc7UZvu3wfYrx2ePRtaeoWB6HOvtZqM2yPdllujrFOJqwUJ9jo/8sj3y+z25QgVwnXq9z0Z+Qjlz0SojmxGSNPlpmkjQ2S/Mm8TxWG1kRYDKFJVHYaQB6ykdf/nMc1XUYdyHPxFfZLKZlapBG7Fgudy3dzKPrT2FoRkmYKwJL8X7FqJIojanbgmulysE/zsTwSdM0Ol6r7OY9s3MOP/bZmXTZ11rENqyS7WwZFmgwiSdsT3bpB0N2/B6BmsGVKmFskIai6E5CLN1knPhoiutl6iZ5nguFwqnS8Zq0vSbz9ZmyJGuqgONZDpvjXW6cOczB1jKL9VnqTq20yjR0QxjHps1qa4nFxpzKYnRevno3y2lC1W3ieU1Zk14bx7JZdhvYtrDPDd2g7tRYbMxh6iYdT7qE108fJExCplrL1A0TshjTqdFwajy7c47zvSv/iMwFiH7+sma/dykvrnPBov3y5gmSyg7W5irZ3FVyI+CJjZM8/MYHuOc3/k/RVVSBS31YfBLv6EtpunVR1sabpI15FTETHls/KZHWrTOJA850L3Jk6gD9YFhmKSIk1Gmq4Vl5nrM52uZge4U4S7GVGG6+NgvAdVMHqNlVhuGorLk9y1UkNtGkTJQs3jRMgjhgEgdUbU+1ky0sXccxbVW/1tUZySGL0TRDfDN0A9uwcc1Y7ZZJ6U9TtKgNTacXDHBV7dtyG2R5zoaxxTiesOP3Ja21hEquAVvjXQ60V4RurR6mboKeo2c6LzlwDyuNeW6YOczD60+yGw153L/Me295A0ma8ktf/URp4K3P7cjIEQWo6nqNyEr4rZf+H7zjs7/Ov7nnTUx5bZ6/fBvPdS8yXZHUuO5U6YcD1oebLNZneXLjFFNei7pT48a5o4RJyP72Cl4Ov//ME1irHTQtIbqogaXc/UAmQFb76GGFuHlVsqxr3ABzPUW79m7MdEhMKfcUOP/1H/pNjgdjGF6FzVOCt8wcAbsCwQCsClgO1/sDPuH3SizF0A1CRWorFDcSX/aU2MNogq5ptNwGk9gvTbcrlltqh6JEQPvPXXiE/a0lbpg5TNuT7uMoGrM76ZFkIl7NVPAoglXRRUlLa09phTe9OrtBH9d0aLt1MiBOY+ZrM6CkEU3l7Lfr96lYIhXxlCFVmqUs1mdxlfZssTEH5KwPN/mOo99KnMZUdAM2n+NMEpHlOTd310B3YNIFTcOdOoBrWBAMyNwmnH4Qp7PKqlNjZd+d7Ey6rI82lZ2oxb7WEpqmMQhHPKtpHKnOgF3Bi2XMjnRKw/955lI83vPQr71bS8XvYpRt8oWrj6Oj88KZozy7PSR3Al4yu59ff+av+OmbX8uHHv8UZ6OneOvzfwa+/kVInuHJwy9hY7TFo2tP0ujs43J/jVPbp1UnRdI4SzepOUIYEq1QhSANy5QzVS3is7vn6VRa2IbwCAzdQE9jyBIqTh3HtJmtTjOJZTreXG1GpabSBZLZMoKKD8IRYRKK74dbx9Itak5F+YZIYKk7dYw8RUsjGcGaxpJ5GTaDcIif+Co4eBLINK3kskQKy8mRIDbltelU2vhqRxtHE/I8w1N6piJbCdKQpcY8lmEp/oKObZgls7PmVDnY3odrOjy69qQ4xeUaQRTwzvv+Je9/9M/5vZf9OH9x+utoiQWxxXte+mK++9idvObQXbz++pfx0n138fx9d3Dv8u2sOHVIQzyvxZQKLp7lEaciy9/fXkE0XzGDcMhqa5klp4YVDCCe8J3HX8AHz3wUPB+zZmO0Q9LmDkYlJq12yY2I3Ei5p7WMocU03ZzhxGKlEzHwhRujlQFHK4OPlms885bf5MikJ9lKHEL3Ipgu1Kcl2NsViH35v+EGv33xkXLWdIEVFFacsrRVlzAX/MTQ96QGxa6fZntjS1JFIwiSkEE0VqbihcREyq714aYaiiafNQzHpXShEJMWHKo4TUgU6B+nMa7l0HQbJZGuansiilWn48zuRbZV8PKTgJ1Jj+3JLlGasDXeZdfvCzZnWjTdBrcv3kT19Bex7ApceAT6a3SsKlNJIOdp4TisPw2GJT+aBrbHOAnQpg5g7F6AmcMluXAST2SkizoXVil9SalVOljdS6xpGlcnu1zoX+F8f+0fMXHxmof93uVci2wwE9JqH6M/RdK+Sq6lGOePkO5/Fmt7hcMrE57txWhbM7zzFXfyi5//Awjhd17zNu7ddydb4x1ObJ0G1X5eqM8w5bWxDYsL/Stsjnfw44BjM4ep2WJxMFebIckSpiptZt0m/dgvdSMtt1GSogrHuKJj0w36RGmEbVgcbi6T6QbDcMTlwTpb411G0Zi2J3WodKiqZScoyVJabh3bsKlZ3l59r+lyk1sukW7Q9fsl2SjPkZZdOBSFeSoZVj8c4seBaDxsj7bXRFeM1JNbpxmEIwylnTF1g4PtFRxl7aBpe4sCNPrKLbDh1AVwRQDXYnBXnMbcunCc53bOcWTqII9dfbq0zrxu6kAJLlcV2dAIhvKdkhB0Q3Abw2aQieVmP5RMteO12Jl0S2X0+nCDl88dh2gs58X22LQrLH7w9RSewbmWlGDsQVtEcCveDA9e6HH//g5/f64nSvlUhIflFMniR3WZwh/5BYrxM6QR7FyA2rS0hzeekdKo0gK3Kc93ViHP+ekvfbhc3KZuluZfBfExThNFMDNVF0iuOaC8a4TwFyiOSD8csu33GEUTXnP9S1hpLnLbwnFRuY+2GccT4jSmFwzV4t8TLRaUg1hRHqIsESJmErHSmKfuVJlEPr1wyIHWiow0iQNy4MrgKv1gKIROdd8XBEEU8e9ge5nF+hzTlTZHpg+wrFvSRUsjuaZZAqbLhaDPQn0O+7kHoToFzXmoTjPJEnYmXVbWn4a5o/S8Jn9z+kFG0YQXH3w+V4ebjGOfmqL6JwrUXmzM88TVE7JJkrM22ODLVx7/x5VFxSP6+cua865DObFNrqVklRHZl1ZgMCRJt2DrMHHd5cR6g/f90O2847e/xAOP/TnHvusBXvtf3sVyY54bNIODy7fzhYuPCDFq+VY+c/YhcnK+aekWmk6dmcoUVwbrrA+3aDg+s7UpLg/WZI6L12Q7lFIpUc7wO34XQzNoe0364ZA8z5mptNkY79BwxBKhFwxKsNZPAsYKWxEeQI2aXSHPEdKbbpJkCa4CY6t2RRZPOJJFWNb5JramMVubUS3ogDzPGUVjFRS0Um9Sc6oMgqEEEMOkYnr4SUDX77NYn6NmV+kFAzRN48a560vMp3gPT7GkAXRPxsDOVKdK/c3+1opADmK6gmPa3LZwIwDP33cXRhqBpjNStgVBElB3ahhJLBMUtEwCC0jgzFIaOVQqHSp2RaAcpffq+n16wUCCVH0WffeCBF5yZnWLzBqAWGTTNEzqugx20zUdWxM849g8/P3pAXesWoDNoxeV2l3PId0T6wW/+BG0LJUAkufSgk4T6OyD3hX5iUaidZvaD15LjiUcgSEugaEK8FVLpgigFbmFhqkb+HGApYsEpeAqaWil/idJEzWjqADqBWjteK2y67cz6dJwa1iGyTAaE462oSyAcmRcel6OEpEnRHyZ5Rm3zN9A1+/z1OZzXD91gDAJ0ZTtwa7fZ320VU50LGQOvuougkbbtZVzooC+z+2c56JpM+21GURjUNq5UTwpu6XHZq+D0TaYLoNELFfjLJHzt36Ch0yLrckuo3DCn5/4FG+643uI0piL/SukWYpnuQRxiAHc5ragNsOpcMj53mVabuOfFlzkhMiOYnbnyCpjOP0cVGowHqEdWCJ/5DGoNXjHY4/DWgutfZDHr57itRG8TDP5u8E6L+6scqizj1cffRlfuvw1fuVrnybXMr514xnecsfruXHuCKauc6G/hp8E5a5849xRzu5eZH97haujLfY1l7B06dYYWsrmeBtTN3EtB4KhqKOViY+pm5wfrNMPhtKeU/Tm6WqbugKMG04d0zDxdJNM09HzjFw30NJYSiHdkJanbiFKMNmxNcB0atQMmwjxGxFDo5zcdBiEI7HANKOy/i4GrDfduhCZqHC4s8pjV08IQM2e1WMRqDSg5TV5fP1pbp4/dg0RUJ6zFNBLNCbXDLRwJMdsV2RypQY1wwZdp2FXyQxHvgM5GI7sbFkGQQ+cOmQJhlujblcJk1C8fPUW/WDI7Qs3ipt9nkmaHU8gHHLfp/4tL2hexyV/i4ycuuFhaEaR3SsTK5hxWswcQlkoAHEPrHwvY9HkXnN/6bWEP/pu+YzKlPx/7MvCDIZw4YuweDscvFcyl2Aoz2saaAa/cdvroNLhZx/6bdF2mUL880xH0QKkbZyosjNXmw/XnNc0z8qOnhg7adyzfCs3zBymU2mT5hkdryUeLKZDmEY4pqP4TJQGYMUjTuMywIRq83FNh7bX5Lb5YyUzvQhMV4abijKx13jQNE0cBSj8awRPnKl2GEcTur7Yw0ZJdM1nibzAUZ2jxKljug0yr0UWykyjg5cek8+xHF6x7w5+ffsspmHSC4dc6q9x/OzDTK/cDl6LQZaQmzlXx9vMN2YhGLI52i7dA3T+iY/wl05r4Xue04hN9EkV+xUHZTfXwDh4ERaXYNDD3jqAdX+FdACfufRV/p/X/Rx86K2c2DrNy//kx2k4NZy1J7nv7MPcPdsmPbnCZ87s8r4v/a7IwJ0ajmEziiaK8JbwxNWTbE+6JFnCcmOBs90LrA03iNKIuUqHIAlLs2icGjWnSt0W5LzlNsTD1LRlJEQ0xjEtZqvTWIbFYmNefF00HSIfPRxBGvNrX/iQ7JTBACY96K/BYB26l8Dvi5AtDsAfQBxgG9KyrtkVKpYotKt2RQkaTdWhSNka75bWkMKvEFezOxdvKtvjAB2vxVRF2MVtr8k4mnBk+hBhEjKOJti6ga1o7YRDWXRWBS0YSJkAj4B9AAAgAElEQVSTxnKcqF0/CVQWNkYPh5KlKMIYsJdCp7HwUsIxTHo4SuLgxAGHOqs4uknba8JwQwJUnsOkx4N3v4GWXeem5kFubR1m3u3IrGjTo2q61NQANfHllSBqGSavuLHNSw63ufdgRTgviUH4o+8g/PH3SMBzGxIox7tyvEFfjt1pCpnOa4JmyOvSSDCZ4VVw61Cf5b3f8nY81SIV5qyHqdz58nxvBnXhvSLAZFYyp7cmXXpKR3Rs5jDXTx/k7849xMef+QyGKrXVzABG4Vh1j0whWeaSXRYBo+ZUr1lReRn0Wm6Dw51VZqpTdCotak6F53YuKD7N3nhdcbRLyuy2bleUalweRQfVs4Sr48dC0GurrtBCfY5u0GcUjQjsCv1wAOTM9tdF7DrcEO7K03/NUmNOZnarTOWRfbfB5nMw2JDKoWig+H1+6sHf5PTuBc7sXqQfDv8XMpfikekQWeBEGLeskj67gRZZsLEBQPS1AOMlQ8jbPPynQ77c/QA/ctfd/Ktows8+mvC3V3+ZZGEVjn0nj/ztI/D0SaL+Mo1DVb5w4avcf+Aents5V/b3e0Gfql0p/ThM3eDWuWPCX9F0ojxjpjqFZ6h50eEIR8nvXbdGmESKvjxkutJhqtIWKb5p03QbaMFILa5Ybs4shTzl7Xd9P//hkT+iYnn85NItEI4lbay2IYkkK1D6EnQd0hhpOuv4WTFUXHw6PMtlGI6I0oi212SiDMkt3WKluV+IdNGYltsANHpBX51suSlH0YS6XaPtyfNZnhFnGVYSYGepvK4IMGksiy/PVPBXuUOey3GDBE1N26vHs0yCqG4AkSzYIgsIJyW4aEYTYsuTG9ofgF2VzK7a4a3PfVYJ3YySTp+Tl2pn+TaaWtCGMmvSMQydTzy9q45L2ZgalrSc7YoESCeTYBH58rl5Bp0DElSKx3hHcIRKR/AYtyl+NOGQpvIzLhzbXMMmIlYamoxBOFaufmmZKcaq+2NouiKqWfhJyNObz/GWu3+QYTiCHCp2hWE4IlBNggJAFnmJ/C3N02uCzd5DV2S7UGXBkrnoaMiw+lTZTxT3vq7wo7ptlCrrpfoceZ5LaY10vQr6QyFFMZWzYIEjFeVfqqYyMHcU6nPQXAJy2DqjpDczXOhdVhKbBrSWRF+VSUm46/d47Z+/jcXqNLt+jzO9S3z4VZ/W/smZS/GQElKD0MZoD7Gf78CwBkevlxc4Npzfj3n8KugG5rDD2xpL8PXf5WNvfiMvml3m0fv+N04sHCNPdZidhcuX+dsrlwHBPva3VpSQTHYQHY04TcjJlcNWCoaFkaXY4RAvjeVmjMayKFTtrU36uJrOfGWKI9MHS6HjglOjbVfQRzsw2ZW6PRrJzRkMoDrFD3/i3WiacBA2mwtyYpNQ2nhxoEBeqXvJ0j1MgBzP8iQDU/67YRKJgbUpNOypShtDM1hszKNpsDnepuU1y2DScpuSESpC1Gx1ipbXkM9NQnRlIYDClcgS+ew42MtQklB+4kDOSwFKp7G8JkuU0ldNhay0pbQobv9JT4LRaEsCSDiGLMHSNFnctSk5X61FaC7y68dfyc0z15EouQSIWVHdEusL13DE+Fw3cAwT1DIyNINX3zxHNHuGPNXJ2jtyl9meyqYMObcg2cnyrbB0Cxx4nlyTNJLgbljQuyyB0amCW1elMbzuxm/nO49+C7ZhMQhGbPs9hkr5G6huYZqLvWuoyglHAfvHpg9K94ic6UqLn7jz+8qMIskS/NgnzmLG8YQgDpXExJf7FPEgtlQ5XYSd4hSLtuci42hMmqWKAxNxevcCTaeufH8sHKVsNjQDx7CpqQxlymuVCu+uP1CD6EThHaaiN9r1+4xC8dYpsJsi0F7qrxMmEZx7GE58Uu6J6jQkAS9u7+O+8S4/dPi+sqv2XGMWv3BFzDN+7m/ex7HOAZmqmUR8+FWf1oB/ellUPKL3nRSyeSp8BBKTeN1Ec5XR84VzpKfWxUxb10h3mrz/5Kf43294Ld/6Z29jI+jyyJUnOPbJd5LNr8HaZahW0Xsdnt05z6dOP8i+1hKu6TKJA7I8o+ZUy97+md3zJCCLfLgpOEg0kZ9ikce+3HS5AqmCAY5mCCkqiWXBDTZkt4t9WXyjHd77xQ9Bc4Hv+Ohb2Qn65HnOSnOR2QJYjCYw6UsASqO9kyI9SVl0xQK+hltRtMI9S2bRFKlzXwnRlhsLCmspkiFJpat2hbrlyaiRLNvLSorOyWRXAOYskYCbxirIxXvnIlGm1n5fXpMEAnoWGEXsq4CjFrKmyfdLwj2MIwnkXEW+BB2/rw5UlxawW4fGHA/c9t3ljh+l0t2QsRoWVctTYzRsVf9bZRtYQ8M8e6PMGn/9r8jxarp8fpaC5UqWkqUCqluuPO/U1HGbqjzSVdCUayPZYiydmGDAvfvuUDOUxG6h7TYZK8qCH4flOJGaJfaSK415hBWT03Tr3LvvTvTClD2L6QZ9diZdtsdd+sGAmlNVA9GyPf8Z9uYvCYs7Kwl+tgo6XTWyNckSnt05BwjjW0zBKe1CCkFk0xE8TLKuEYNoVDouhkmEH/tsT7oMwzGOaTOOJ2yOt7kyuMrWZIfzvUuc2HpOFPleAy59TTJfw4LuJZI7Xg8Xvsq5xRs56fcwdIO1wVWuDK7yn5/8Cz5/4St84Ct/yGx1irlqh4YtpL/i8b9eFgHh+05qzjtuyEmUy3+akn/1azA7D5tXYWqK5GIF64aA+OwQu7nArxx7OXz2I/z9v/p9fvLjv8BPdM9yR/sAX6EP/S55JMYzfhwyiX2arsyIcRUAN1edEVGW6bDr95i1PFkAoy1J6Yob0rRlMYCk0JYrtpx+Dz3PZUFWp2VxJYG46+k6ZCnXDa7wuyc+ye1zRxlGY1YaC9yxeDNceRJ2L8ric+tyEUxXbmrd2GOJqtRTgkyOZYpncN2u4phOydg0DbPk7xRs0GuBu2LekK4Wi21Y8t0Ma6/0Kcqh0ZYKcJksLDTFxVGLMc+kC5NnsuiKDCsOVPfL3Ttmy5NzEvlSg0dCjiIJ5bP9npRCRUlIoWtCzkWe813XvwQdjY+d/hyFgBIgJyvBU9RcZul4KEJbc0TWq+99R00XjMV0VeBM9p6rzcB4W5V1KoBqOrRWJNhqOqgMpBf06Qb9cpbQi/Z/U6mG1zStFPZ1gwHDUOwim26NRl6jG/SZqrTQ0Tg2c5iGU2MSTaQrpAy++uGQnj/ANm2mK3XVMbQIk4jCD1rXxbxJOFBGaT7mJyGb492SfNZ06wzCoTJ2l66mzJYOcExbsYIz1fHMlZGWgtsMi6pdIckSBqHPOBLZScttMFVp0/MH7KpAMVedklLHbVBfPwXHXyGbdRozXr6FNPZpHHw+dVMIpF2/x2fPfYlTO+cwdYNTvQtoaNw5ewNtd5GDbXEb+GcJLuVDyyExsQ9GRKeAqTYMR3D+PHkUEHem0Y8sk/Zy6ULYMPXBlzJ93cvgjR/ny1//KNqtLXjs62j1MXct3cSL9j+P+dosR2qz/MpX/oDIFIR9FI3peE1mq1NCQnObEjjsihxLohaLHNjewrq2HDAseS4aS9rdXODUeKfUEH3zvrtKUDVKI5bq87BxSkDccABuk88257m/qPU15EZGk8VakJKUVoksVZhIjqOb6JZHksmO1DY9Im3Pu0aMi0I6XqvMdPRUsizTcsVZTtMhiyCO5Lv5KoMqFqOGlDkFs7fIcopdv7AtLUHeUB0/e5lYlu1hNpnqdIQj+fekB04grzUsYcaS7/2OpvOmQy+ELOVjpz+nyGs5hqaRZAVTNSXTNAzDKHd0gNyOuPsWf+/c5bkKWOnesebXtM0rbfn/YKDKZFfKNKcKusGVyS5BEjAKJ6VB+rUYiK+6YPcfeB63zB/n8asnOLl1ms3xDpqmcbC9wpXBVfa3lsv2r6lwhjCNSLOUfjAo9UPF6BFTN3AMh27WxzKs8loWkx2qpTm2mHQPghGuadMLh7TCOlvjLkkxrsQwS+V+MSO7mF5ZYDeappWmY8IKF+a7BBuvVPkvNeaJs4TF+hyL9TnCNGIS+UTLt/DM9hluWrwJkJZ5ksZs6AbdyS6e6fLk5jPs+n2eHVyiYVW5e+4YX944QTFXyTUdfuCWXy7hpP//waWcy6zaiAtLcPY8zM3CxQvqNTrZU+egVsP5rbcTvuYXwDD5wOffBQ/X4CMf4Xtedi9/emGa3Iq5b//zONu9xMpn/j1EQ5zV59ELhtc4tsnON1Od2tvZ7AqDcEijOiU35GhHxkBkqQom7O28WaZAyhFM7ef0cIO6U2WmOo0djiR6T7pSu9tVSB9T3aJdeW+EVYvTkM8vMpcik1C7N6Yl2Qxc83yMZ1eEFm6oUs6wSt2ITO4LcHUD4gDtWnA2GOxhIgXonEZ7IK4aOYGG4CPFI08lsGgqy4kD9VqFYaSxAkU12f3zTOEuAO7e72eZ+rsKzCCB3bD23hP2QGO9GKSWkSuriWK4Wq6wpEIpXuAQ6eU5vvgjb5UMRVebQPH+pqOsFXS5LrqSM5iOnJtoIq+3oK9KxM3xtuKKiHexhnxmigR7ybtk7jQaMqZVg+um9jNVadP1+xxor5BDaYdauORrmtwHQSJZtqmZ5FbOrt9jEgdEyoGwwJj8OCzb0FkunrmTOGDX72PqMoEgyhK2J12CRCQqRTlVALG6ZiggVnI9YeyGwjlJQuJJwiQOSmNx2yjKT4c4TbhuapGO16Ie+/QU/+fqaJOnN5+h4dYZIaCwH/tqzK6snWe2z/DI2pO0vQar1XkMXWehNsPt2fUMozFRGn/DWBH4Zwgu4b87pQE4/+ZoDmAdbRJ/dQS9wd6L4hhGfbAtogcnWL0/Jzn4NHkEfPIjcAz+ZOoAf3ZLj8xe46GLjwqFfuYIPPI73Hffz/BnJz5FPxjimPY3aG0glxs8S3AMB8gEdNTYS/+zVDIbw1K7OGpRm5CErDQWcHRDsJv+ugSWgfqzKLPyVG5iuwJug1ea3l7db7ly02fx3gIrukdokkHoBqnlYBT+HprOWI3qLLRMFeU/0nIVqFsEq2s7PcFAtX5VeZNEUraYyu6yyDySa7Eg9Z01Xb5zmsix5Xl5bN+QucS+fE5xvnQdTF24LLqii+fsUceTaA9MViAtZJDlvO8lb6Nu13nrp35ZbjhVCqR5hqbc4otM4t9/6zs42vkTCWLF+xT4lStGTWUANSQYEAVyTeyqXC/DAq/JJAnYUKVi1+/jq7ncmpZ/g5F24WW8v7XM1eEmg1AG6s3Vpmk6dYbhiJpiznqWS82pMo5EuAiUavcojRkmMrCtkJKMI1+Y15pBy2tQs2U88jiaEKUxg3CkZAQZUZYzjn2s1GSsrBnEGkJ4UZoqP0WNL/OvCg5OATQXgKtnTtA0TXkBibh3ubEgpMlLX6eeZ7B4I3qWkGRSptqmzfZ4VzkGyMSHwnheV7o4UzM4NnO4ZJ1X1KjfS6pF/98//nnKouKR6mhuBFEoF714DORCkOXQ75I8kmA39vO1H/9Fbk8T2HoOfu9XaR6tcKS6xFxtSiQA97wB7/yD3Oa2eGpqPztqRxjHE8IkYsgItzpVOs9ZhrkH6IIsGLey144tdtQs2Uv3B1fFGCgJJZOZ7EqACVSrs0jPlRkRhiUpt2nLze7WJeiYNiT53iItA0IKukGoPHSTLJHjVK3mUO1QaZaRmTJd0jQNCpZsyUo1THlPNZak7ARFvsKKFMu2OiWvSxNZbNFkrzwybTHqzjKIFXZRtKDzHGztGvsCU5VPuQSUAqjWzb0yqdD56CqoxGPQq+r3BR86kgHjXX7z5b/Imz/+LmUcLXT6qumWg+f/+LveD+ce5oGD91K6heeJytoUnlJkK4ZiSBeGUddmUXYV33Lp9bYJ4qD0NC4AZk218AGxfkyiUkvUCwYyT9lwuHnuBtaGG9SdGtOVDokrzOY8z9kcb5ft4wILkS5PypXhBjoa49gvvX0KhU3DrZVeMZqmUc+rotpXM4eyPGOiRhrXLK/MRq4tp0DFViVfMHUT8kTZVZqQiXVCmqUEsUhg5muzLFTaghl2L8G+2xlmKY2tM/SmD7DUmOPKYIO22ygDv604WZf763QqLTRNK1XXt8wdJc4SesGAplvnV/8HI1//+YJLITjLNcy7ryP5wpP/8DUjlc0cvY5X3+hy50d+DLScT7zsV3jZJrz51a/iaxunODot0fGpjVPcdfePwVf/kG++6/v4oyf+qxgTJ9LyK+j/MqFOVMyuYYvrnakChmHLTgyCFZiqLEJ1QzRdZTeJZDyDdSkzNGOPH6Ibapc2hT9hVyWwWK50J5JIyQEMlTGoBYn4ZOUFQ1OjdLQvRpAW42IzTTxkRGIAJShMfk1JpQKBZkCqujuZ6gJZHszfQKirIWF5LoFvvCMdsbJ7kquSqcjgxhIw7Ir83bD2ujF5gc+E0pHRVGZUYDjhSM6vYYJeA/S9jKkA1A0bLIcZv8/vv+ZXqVoVmYWj6fL7qLLvqY8JQGt56nzapaSgLI0K0FY3VRmnAqQpQD26AV6DrckuvWDAIByJWDBLGUc+uRpoX2AVAWG565/eOUfF9sSDx3KpaxrLjQXFcdEwDEf5r4RKMhBK+xbKAXeDcESUiqF34Uo4UlR7Efyl9IJBaQtZtf8bee8dttlZ1/t+Vn96efv0mUwmyaSSTAKETkCqStiAbsBD82wLAiKigHhAA/soiIqCcunBzkH3ls5W2iaRCKmkEjKTmUyft5enl9X3H7/7XusdTBBCAu5z7ut6r3fmfdp6nmfdv/Ur31LEUZymtVE780+quWVJXBV3J0pihtFYZCstN9MCtpQveskpZDiYhieuFhPFOs/Y80SmSpO468dh5bD05gB6K1QjH7ZcSBIMKTklLpm9gPVhi3E0zljgpiHcpqi/jmVY7GnuUPY5ifLTcrOy6aHWoxZcsslRbGI2+hhXXkH6zTvPvpPuAxw6wj9ePCK9az/u9hGH1o7yvASue/yrePE/vYuJYoMrt15GvVCFb30e1o+w57aPUaxM4alOeXvcVU0qSzGcAxqWIyel7nPYakJkIIHDEf0VTENS/FCNY52CnLTdRdlghiklDsjJq82eFKUfpyCv41VJLYcIA2dzlpOSNSNdw8RPEwE9qfHzMBxlQKeOLydbou5LEuUTmERlGbabN6OzzEGNhmN1Rdt5IZ0kZOSPhHdluZQKAuEnGEF3CU22JI2lp+KGwi0pT8r7jCOwo7x/E0d5ME6RwBKrDAIjz6bSVPW31LEnKuNAjeZjCRbl9jwkCa5hqPFycnYJZhhZr0aMx1TGmOalrwQ8RWlwinIcpqXKwpTQLrA+PKkwGCLWNFKcLymL5CJoAEkswTtMYgpOQUHmDeIkJHVKeGnMTHkyQ3RDqozVbKJkcBakH/Lyrh8M5fRTEho9pUYnUhzQSrr0gwFbqzN0/QEb4w4avVu0vYwA68chg1DrOYtwt6YtxGqDl+xCVqJgwESpzn+++CeYfPDrcPhrUJ6Q7DLdVLak6lxuL5CW6ky6FYaJWKSMoljhycSVo1kUs8GNVptdjW3Md5dwLCf7TL+zz7J5PaplUVrrQbuGEdk4s22Ch7tjwcNZ2kIwGpMGDtefvI03/95H4Gsf4l1P+yUOrj5Ie9xhtjLNfrcEYRcOf4qd1/w2HaWJUnaKjCKfYSgq76NoTCFSLOJxXzFAPYUwNeVETFU/Jk1U2aZO2jiUoKLBZMk47y0EQyjYgJUHDkBvfiNJBFCmjNBlM6iTLvLBtPFISe0CRgq2LWQvQ6FYUQAy01BXaMh7IZsxM8FQlSUqy9IlEykUasLzUGNtMBSBUT2naeYnl6YE+F3J0KJAykCdoTklCRq6v9Ffl4xCB1sd2OIQ1o9JJleoSZaksytdfnplOWa7oD43FXwjnywzi4OcuatIdLhFOQYdmNyKAiaSl5sY+edkmFnfaLG3rFCyfTrjPkma0PeHlN0iCSJrmWFUjRTLtCk5BU62z7C9toUNq0OQhIxjn6LlZrKTD6UeUHTEjVRnQtpPKVZNao1TqulJJgYYksF0/D5df6CmVwlTxUbW1BaFuwhtc+OrPpqe+GCAq2gAM+VJiorHBLB/+lwmj98qwwgd5HWvzSnKAKZQzbLayWAMvVVKM+fRGXdJEpFT0GPymhKmGoYj7l66n5JdYLYyRc/v81MX/9bDBhb4AUB0D7mMBMNKxLcmMWHnbvn7/v1n3288JrinBw8+SHhkxBfvGHGw1IDHv4rL7/2sIvMpjoflgS1cjB/b+xR2N7dTdkuU1cRlY9Tm6MZJWqMOa8NWVrOelb5n2UYhnxw5avJgi9ct3UU15lTTnjhQOBkVOJIoT+Oz+jfN0a/KkzfLMHQwiMYShMbC44nS/OrZGrUxgK7fk/fqqwmN3qRxIK+pNTg0KHDYkr+H6vjGXWrjvpRDCvNQ1X0k1XjMlt9XvBzySZqlJCNtT31WioGdqJGv31P8qQ7c/zlYPixgwmxaZ0v2EY7kPmPhqmQr1Mc/lozJH+SlakEsTbMmeqRG+frU3BRzs3LRUNIL4Vg12t2MeLmz2KBge9S9Ko1CFc1u3hjJe95sfqaN6jWy9K7Fb3NOcwcVt6TQrCklt0iapoohL8G8rhjR48jPXBVbSgZDBxQ59JS2YvD3gwHz3WXmuyvMd5czIXehIpBxyXQ5YpviGhonEVW3RLNQo644SZaCSUwquIJt2bi2y2Vz+7lm4hzB/oxa8v2P2moPFKG5A3Y/EabOYWS5UN+aB/FwnB1DiljkJCrT82yXa/c/l5+9/KeYqUxRccv/bmCBRzlzCd6xYLj/9/bU6FcgsnAvjUn3Xkh4w/1n33HYlx+A5WXirefz6v/xG9z2gvfCjZ9mz/Q+tlz6Igprx2DpIDR2w+AIjev/gNql1+KoMmV92CJMIpqFOiW3SFspepXLk5ImK3wITjHHuyjciZRFvkrJXc7qcfhDuT1NJa1ME8DKs58MyJZsmqqk+QRGB7VkUwajCYWbAou2ech2j+5X9FZysJrWkRmuS29B94hGHbUJEyHznbiV+s4rJJMINYJWQfc78/K8GWp408Y31Pu2ZZyP7cntOw/I37/9BcEmabDaxF5YOwK1rRKovKoci+3mo+MkBAqbgjB5+aSDZhRIhpLpyNg5kE9nNjrQ616RBsZp0JwaSbdUQ7foFCgo9OvJzoIgVZW7g6X0cje7WKpvW24zUp608wC7Gtsp2AX5uFIoWx49Jd71nSuMRVWupcoa3SwO4kCNqkUfpjXuZfExA0WCcgdIlByFQZhoAmWCqbytGzr4ksfZOE3EFN4tM1EQe1eAp+x6vPSuFr8ln2FztzywUINdVzFSkiTxYB3HshkaFuVCjYJhktoedjiiWWqw1FshRQiVfX9ALxjw7fQwpmnRGrX5uSv/4N8NLPBoT4tQkpj/dUdqDMrgu4Q3nQC3cPb0aPMyDJLQoh324Z/fCk/7T3DXn0tjdrAOi3dBNAJvCxz+Jx6/7XK6FzyLby0fEstV1ciK4oipYlM2r2kzSEIBKjklRmlMqjIWy6vg2Z5siGFLpf5qzNxSfQm/m9f7/iBP6ftrkgH5fdlMoSeP81QPwu+Tgb5I802grS/U+NU2rQx4lKQpZbcor1edycfLgzUJKqOupLjt07DjgFxpRm2V7how7EJvVTbaidvkOHSczNDCCGcqSWTj60yMFNJNOBy/L41S05LPR4PkUhWYCzUhtfVXVf8lVBu8lAlMEQ6hNCtBvdSUrMcfiByCDvSWC0VT9XRc1aRW2QhIiao2uIzGVbYSR3n5ZVoi1oV4FrdGbeJEMpLjrVNEsQgxjUJf4VjEqUHLWdjKMK+sbHyrboULik0i06FZrMlEJhyBKnVAgpcoCspH2yhU6foDJgp1WuMuCSkdvw+IjUg/GGUC7KZhULILyk9L3usgHCkbEoNhNM4umlESkcTCL6qoJrNeGnDY84fUvCrdoE/fH2KaJm4SSUZ5+nqo7YHyNNTmwHLwyxOs9FZEyygYsq22BccyWRuuM1eZZWO4AWqcvaO+lUEw4ObTdxIlEeujDq5p8/NX/eH3FFT0etSDC4DREwhwmhpYB/YR+zbceefZV0y9xkO44w6OBJfAe98Cx74OpR2w+qCcqBt3ymDDRKT9b/sdahc8m3OaO+n4PdaHLUbhmLJbEvHqyKfj96TBZVoc2TiJH/uZpoakuB41r0KzOk1FcyGCHbBxAsatvDGb4UzUsTpIgAmVYptTFKZu5Cv/HAVg0w1RvVl0uRQMMN0SNadEVIgZhWOCOKDn95ksqo0Yq7FzsS7P01+BlYOwdgfsvFIykmAom62/KgEh6EOxqfAvSNkTh7IRDSsf5WYj+k11hu5ZRD4c/Dhc8HLJfjqLcpsuxzAlyDV3yE/WlI2lYazkDMDIuUidBZkymZYExEJd3lehxsgRqwovDiX7G/fkfQ3W89e1C/JZJLEgk02F0UmHeTMZYQiDwfqoRVvZtARJmAHdQIzRokSU51zbJU5iURh0y9QLNfGXKtQYhcOsoBtbjtirmjajcJydO4ESjKp64qOlXQVaqjSNEumpSO8kVXwy8TIaR+JXHqusVixtIqV5I5PDUTwmIc2+Jn08Ovhos7Qz3SXmKlO0/S7bq3Mik3DkEzAGiqKPS7EB1Rm8NBcXSxVlwDUdmrZHqlC92nJFZGRPKlXDiDc+4UPfV1DR6zEJLv7vHjS8d+9NjcTEshKsWkJQa0Cn9fAPOnQYPvNamLwMnvUOuP1j0F+U8zVEgksMBDGsHGZiy8W4tottWBxaO0rZLTEIRgoXkDBbmcYJBaE5DEesD6X2HEcieVl2i9S9Krsa29kzvVeukFN74dhJCNbBUONOu6QChUrHg2GOAdENX6eQl12W+kg3lwTJpmZkMCQtNsQD2yuTpiVSwE9CPLecSx7Ynsg4ukXY+3TY+QR5Tn+Q19GWuuzYwngAACAASURBVOoXapIBqD4BvaW8b7N51K4bwBkXys5Lut4SxH15X63Tcj+N0vUHeRnYW1EljCcBq1hXkyHk9nAsWKEkVlMlaThTakKxQd926QcDhv0uo3DMbGWacnmCopZ9KDXlNZIon9KBmtqpgO2WoFAncYuE4ZiyU6Rt2SrIkIHkAt2ERnoheuxrGYLIrSqFfQODqlfmZPsMrgooqUKqVr1yhk0Jk4iC42EnogbXGfeoeGXGkS9ZhC8gun44zDRzHdOmYLvMlCYIk5jWuCMWIqpXM4oCCoquYoBy8XSUbKqZvR/PcjP5zYpbYmWwwTAccfWOKzjTWeQZe66Gr74fIgSKEbcVcluVzesnKOns0CnKbaUJiEOM2izNQp22MmirF2q0x138KOB1V7z/EQUWeIyCC0A4eQZr1MQcl2DsYl+8g+gb3yW4uB7ewk7+7EnP47Xv+n0+/ksv4OVffo8cYUr+OwVu+2u8F1yH59XwLJfT3QVWBmtMlyZxraI6kRxipyhqb16Vhd4KvvJsNgyDtaGkyov9VU5Upnj67idhWo5c/Y9+BkjA8CDqS5lkubLRTdV3iQKwwxzqr+UL0kJudaH4REAOrktE5LvqVjJUpakg8RnhEGQjhb68TqGmlOHUZokUWXKwnmNC/H7exNZTFs230cem6yWnnGNb5OBg9V6o7IeTN0BxVt6/qQJKooBsTjFv7FZn8h6Ifg7SXOog8iXLiEP57OpbGaUp64M1hU7tkSKSoEVbfWY6k4qDPLCd1Vy2pbwqTTBIE1q95ez0KdgeVbfM2mBDhNwT6WVgkjVNHdNWADEXyzCpFSqACI7vqm9nEA5xTJtRNFZeWjEGFRzTZro8SaNQUyJLwlFybUcg+wqOr4mPgNIh8pkpTeBZLrsb2+kF/czOV68EUb4r2UVFUEwyAStd9nu2S8HyssekacpcZYql/ipP96ow14Rv/7Nke0UyCBNpqvphXg64DEdQmyUDlSqJD8e0lANBnYXuEl2/z0A33R/hesyCS/Im3zA/ZKepU8UKpzGLATQmoL3x0A8YdAm+1OXnrU/AsMAr3v23vPwvPg23/g3c9xnoA+dcDffdDBfugb9+Njzh1+HiH6fklAjjiGE4EolLwI98Tg/mpateamIgiMxR6AsOIZW2Xmskthn9YMils/vZdem18kW0T0L/NBJkzBzenpV2KhhoPRTLyYF3cQCJGl1r/MbmsUfkY5BiWC4RgrYkVhIROhjpXg2QjbZ1CWLaki2ZtqBj/X7+OcaBbMg4kLIyDmC8JjKSAJ5ijtsqUBgWLN8FpmrIDk6Df1qdeM0MDJj1oGxXYWRKOWlwc79Ev77tSVZXakJ1mr7SdtU8HENhOeJEELRlr5KPqYORYmVrQJ+nRtoOlCfZCMUjqq0kH3RPxLVdym6JMBFMSJTGImegqlzPcgmTEEuB0apehT2NHbTHvax31/V7CjuVb400TTFNAwxDlVOOcucMSBG2ez8YqJJIslQDkUhwTJtGscb6qEWoslnbtBgqZrzGvlQcgxQjI1Pq96T9nstOkalSU0p7hNKwu7Edvv6n0mTXMIipp0D3OCQKdxQpTlZjuxLdagGmAkzaWelvOQUadpFGOOaOUZt+MHjE5ZBej1lwAbD7s/KP1CBNTCiVHj64qBX881GY2wpLC3gf+HWue97TeFsHicgz+2HyZjlhAW5+P+lFL6DkSKNMW6cWlT1HwfYo2gUeWDtGs1inPe5Jn0ZxTYJEfIYmCnLbcn+Ni2b2cfUz3wyHviJ9n84ZxddRBEmN1tUCURpvofEamnHsFPMmpC6VdJDRmU8Ktq0mVZGfl09aiwZydK4S2Mar5P8uNaSZO25LduWUVRBUhM14LP/2FyEaqiuamnQpQzeOfUICN8jZoONg+w4YbQevCU5FMidNmTDIZRccRX3Q0x3NpDaUBIXtQmmC3qilgGBRBj5zlOyAH/sQqywoE0JXr5FNS1JVqpoMw1HmiqhpH3p8O1WeoDPuSj/HcpXQuvheaXF00cwxsQ2bRqFOCvT9AeNYpENrXpWR8vYehqPMebPn98VFwLBxLJuyI4GsNepkPZOiXRDIP0iAMGCq2FQKcfI+ct8kIxuVYxgYaYofh4win6qaLFqGJVMpx6A97lJxSziWQ71Q5dl7nwLHvw5+Rz678TqU56C8Tb4Dryp9rulzGTgF+n6P2cqMTA/7q2SAR22Voy6G68PWWcH1ka7HNLgE75g3vN84P03LQ9iow0RD3sD86e/+wKUFAJLmBh89+Fne9rJ3wcevg41jcNEr4TMfhllgFarjLpfM7ufoxknKbpG5jdMw7hOVJwWRCcomxGRj1Kbj98XaIQkJ4ogwkcZqglilzveWWRu0uGLnAbbtvApOfVO4T35fcVts1VsYSk9Ed900WEmXSYq4l2U9Wu5A66tYEjRM2yMLOBloTvdKFBvadtG2EPJc5FmU35Xswm9DoNjRicpIkhCCpSyQkQLRKrANTv6LfPs9pFGeIj2tTQMmOANRG4o75L+O0m8xFVlx3Nskb7FpOpbECqaSD2BjpaofKvcBNQTOtV00iC8cyWcXx3m/KJNeMEFhPMRAXiJh2Sll/QkDg4lSk5XBGiSJAsEZGZJbS3CW3GKmpyxEPSlV2uMuZaV9PI591oetzHpkFPqZuV4YR5TdkpQ0yjUTFM5FHa9niYNia9whhQyAttxfx7NcDCPMfI0gJ1E6pijNOZZcKDdGHboqM3JMm73Nnbywvg3u+qRcPFLUBcfKYQ+WJ4Fl6hxobGMYDLEtWwLLwrclcA82JAMOBjKVnNlHa2InUXeRV1z6nh8oa4HHOLgARDPzYCRY3SrOlEnoTn/34KLH1obJC7bvYW9jO9z7D+Ajk5xj/yKb4BQwDfzLH8FTf4mdjW1izvXJn4cQ7C372DV5Prt2PZ4bSg3OdJdYG7aUlm6YaZGCnjSLyrrmjayPWuyf3scTzn2aXD1P3ippeVarNuR441BB6nWvI8zvkyZgGRJkLEVGxM43cJrmGQrI5om0cJKRBytH4U+0Vozm/PiDfOztVGVkbwCmB6lmjpvydIpOhdWEU3fDCFBtDmykaa5bRJskaTE98FfljuUZKad0Ou13VWaiUh63IM8VRzlADyAKZUyshJnkQq09utUUxLTzMtIt5xmg7clnrd5nRJo3XdOUqldRBu3C5XEsm4lAhMzjVC4cpmGqiY5IGJTdEvVCVWRGlXGdp0TKLcMSRnM4zLyTIw1uJLdcFWMz6YE1ipJdtdwO9tii4hRJEf9o25CMuhcMqdcrmehULxhmNjhxmuCa0pC2TRF314pyBignyCqDYMhkqcnlWy+Bv38hDJGMfvpC+fxtT8qesJ/3wiIfuktMT+yS82fj20rStSM/pQl43LVguTywdpQHTt7GT17wjh84sMAPIbjEb+5L72V6BXN9GtNJs37TQz9AbYqKfGEf/OwpfudDf4V3w5OhdY9siiLw61+AdzwfNr4K4xbOFa+UDGMRGADzR8A5Arv/BxvXXJfJ/Y3jQGgDkc+635ETyPKou+XMt/lEZ4FBOMoYvFfNXSBfzGBNvixHSS8k4aZGbZpLRFpGPplJyWUYIMeexDGQ5CA5DbrTz5cmeVM01ZFhU+9GI2O1vKVXVdOjSIIMtvRRkjqkvhxfD9EY7sjNBMhkIUUyjYg8sOiXigdg1yFSGjJpLNgYyyVzRSjU1ChalS6oJu5mlC4SUAwFW9fe3/r/+P28aesUFWAxlezQdjPRbcMQPeGKW6bkFJkpT0F3Eac2pxqQBjWvymxFjMc6455kK6aFtmuZUmV1zatmIkd+FGQqcMNwmKFn4yQWp8FULGM85X4ptq0iiVn3xKC95BSpONIkLztF5dRZph8M2VIpZ+VZXVn5+nGAY9qK0WzhKKcI25Qsa763DBjKU0vG4Ntrc2wtTcjFQsnXMO5Kg93xBIaw8S1o7JfsZP04Oeu9KOdtcydYSzIhrE7zjfl7aI+79P3BWRO2H3Q95sEFIHljZBgfNlOn4WCOXJJLLoX7vvXQuBcdXEYDPveZLs52h/JbXk/0pt+EP30v7AeqYP/5y4l+9h3wh78Dd9wJpSk49WVQfUve/v/w5aDHcz76Fl5y9Ovw1Nfzxps+yiAccaK3yK29ExhRCSOxwIxJ7BGXl7exozxD0fY401umrnyBzmnuYnLmXFgMJDjoK6uuS3W2kmzSSYmCXNLA0FmIqj80UjgD3LEJ2EbeKNZIYJT8gu3kkH8txaAzpHAE1S1yoi3eLlgHF8n4ykggGSMBJlF/V0+NzsxN9aOVNi3AUscUtmGwrCY2TXBdsIoSbMKRnLha+0UHzCyoCrFOZwIg/BkLaXoaGAq1u6nvpImJXlnS+8ndkKb4KqNxlFh1RruIQqVnYlIvVBmEQzzbxTHtrFQRDyiHiWJDOWBK1lh0igyCISPVKNYWwEEcsBpu4FoORcfLsi19/FESZ7iWqVKT9WGL2cokk0kja8RqJ05TMZhXBh0MQyQZtOtBLxjiWg5VT5wdKm6Jhe4KruUImztNWR1sMFVqikGfYcp31kIutpPHReK1UIfGTrnAGkbumlDdJGVam5MpZGMbBAP8mfMorDwgmrvRmNdc/r5HJWvRp9MPZZlxgbC0glkIsGpGzjt6uBWFcPwo4TfPEC/2MN7zcV753KfCncAD8Jpzr4JbfwdKwBxw25fhDLIh6kDrFB+797Pw2g/A8S+D6eDHAb1gwG2tZezuFuz1Oay1Waz1OZz2Vu5pdbmvfVzSa9NmZbBBZ9zj9vl7xB50ao9kCKAkCJCf8oR8mbarayw16dG9FJ2rqaCgVd8wci2SbNqi+jNat8S08n6NxpPoUWaoyJdJLCdOfwk6D8olw0KCSaJ+D5GMZYjKYIAN9e+huk+EBBat86WDjuFAGkD/GPQWck2cjO6wOcj5+bFqTZokplmsU/MqWcZRdAq4pui91r2qAt4p9rVCVuMWZWI1dQ5YDoEh8Hpb9bXCOMTHgPocY3Lx66JdoFmoM1mUzSgKckXmKjPsbuxgrjpDs1AXFXzLzrzGkzRV0P0EP5apVj8Y0BrJ+DlOYnrBIPO/Mg2Toi1yB67lsG9yN1WvIpINCgluGxaWcklsj7uKZBhJsEK0dUtOQfrX4ZiaV8E2beqFCiWnyGSpgWPZRKnILiz1V7l14R4o75DAUgfWD8nn53clW6mdB24daluynguWGkcv3Afzd8Pkbr5l2dw+fw+jcEzRKVDfRDV4NNYPLbjEvzw0yqZJMrmOVRpjzDbAK8HjHvddHhRDsYC1pQpxzMc/cpDrf+uPIIWP3vgVuBnJVO4CTgLzwDqyST71Hop2AfOL74AQ3nPXfyNKYr7VOY7dnyBdbxAsFAlOpARHDYL5aaz1aU52U+5vn+CzBxfpBgOSNOWQkvhjYnfeF7ALUJmRK4YelWoOjE7IMq1aNpERVZmTauRvmpdKOrPZLLmgWcyR/x1gOHJFuFAhdu0CYIJjQAWoIoEmQjIVE8lW9P9D9XukfnpIwLGRoBSisCo9Ka2CNgRdEYVKU9U4dBRFQjevQznB0zTXLR61scc9ZstTVN0yM+VJpkpNpsuT1CJFxNTuC6ZWywvIfJXUshQ7eaDKFs1vHilfcJkWWkpPVmQeJ4qNTT91yq6UK7OVaQDqXi3TL9a8H8eyCeMoew7tjZ2irFyAcyf3sLU6S9Ep0CjUqLgVCrbHrvo2irZHGIf0/YE0etOUU53FzPajMxZ1ujiNKTtFhbw1GEc+D6wd5+DqUaS8K9MsCJbrvIndFG2PIA5Z7K3AgdfJ91tW31V/GVYPw5nPCzarsVPK1Z0HpAF/5i6ZgJ68HbwKN568jc64x5nuEq1xh0ahxov3v/NRy1rgh1QW6dX/pdCwf38yTWs9rLGLcfWeDH7xsGttBWP/NPaeKtGNJ3nWK96K86ItBINF2A2881/hwRsxv/LbJKOAwa/cyD8d/io//c6/xB2fxBrv52efOc0rdlzB/WvHOB0McUYzREdasLRIFgkcjyAKwYBv79qNs6PIpw6f4BOn7uPPnv5fuG/lAeqFGvtmzhMYvFafM9RoWgee9plcy8S0yRwIMjV7FLBMNUH11EcTKjHyDEV7D2mJhaw3E+VmZZYjV/fhhjzWMCTTMALJXopI4EiRoNICaki2kqDUrMjLIEf9TfeDB0BJAbHwIViTur7YVONOpfauTewzLd5xTrDUE7BhS9ThDSMnPPo9mVQEfRmra1Z0ZUb1WRpZFrQ+7jBWn2fBLigvbZGT2OxoCOKVrX93xj3qhSpFu4BlCiHQMi1qXiWf9qjPvF6o0hp1JLOyHIqOPGYYDKFoMA59Lpw+Dy9N2dPYQWvczcbromc7VqBIIRgaBkpBcUQKjCLxNIqG8pi6yoTP9JYZhXIxunTmPEzDYKLY4FRnAT8K6QUDdtW38vJLflL6Zn/7HPnOWsATXwndeVj6hlwcKgvQWBWpjOO3yNj54F/JxWNyC8ee8H+wvHCvqB+mCRW3zNU7fu5RDSzwQ8xc9Ip+dd2YafYxiwFpaMG938p7Eg+10pToRA9jeh2mZ3GesZfwxFb+7NpfgRn49Rs+iPmhT2DP78UseJSP38JPv+8fYf40wf88RPT1e/nLj9zHfcuH+ZMXvJtdXhnMRAJLNv5NZUKVxJItnT5N2HMxhiWMyOXnvv4nGIbJ8z7xCzK5MJTimg4iGombpmJXov8dhyqzGOdYlWwZeQajezAYOQpX955ASg2NdtVZkd9Tvkl+niH5Zzs0Zj0UG8lkQK52DtBUd3PIMxW9NpdRoLIc9Rqml5dA+n3qDEZ/h25RAms4EmvQ1Qdh6X7YOCmiVYMN4XFtnJC/rTwAa8eVT1KQN4q1prFhEqkMxo9ClnqrLPdXWRtuECVh5tJgbMpyXMsVbR9ElKvkFDPshqmmea7tUnfFhTEFUlVWmYapgHIR+yb34Fo2tUIVA2gUapzpLhKa4s/sKeCeNr/TAS6IAtVj2WCpv0aUxpzpLXO6t0R73BOFO9NiabBGa9xVZn8iFnVk4yStkUhuCmZGgsAFU+fA714F73+OTEvbSHaZJnJuVXbCju3ynZqWILjn74b7/woWkMBz+Wu5Y+FehsGI/3zJbxuvuPQ9xjP3vPFRDyzwQ85c9DrzCx3D/LCROit7sZ98IdHRlmBgDh46e1Ppdeok4bwNk1OE93b43B++mmv/+L+z+1c+wG03fZT0/gXCJIVRiHX47+Hgd0g8rC3z5jf+KW954k2k+4a87vEH+Mv1GunBB2E0/DevaT/hAqL5DmlDmo+pGVJ1S3zkWe8kNE0xfNdkQMNSYkhJvuF0Ov9QDWsgs/9Q6TBGqtTCNim+6bVZjNwt5VooOngN13IOj9+SlFgHFx0wdEayBVgmb+RWkKwmVPfR/C0LAdbpEkoDcE3Arub9oIndQmIcbuS8Ji1faagsyh9KhlKezH21tTZOmsrodNjK9G+zAOWVpdzrJzCxizC1GYYj1obrIl9JSr1QYxCMqBWqOJZDotT2S4oyoCdSMtI1iNI4U3QrOQUcwyFIIjZGLWFQR2Mmig2lOgdFR0CY50/tZaG7LBQNQ8BsR9aPYRlijDZbmRJwGoIC1pOl5d5ydqymYbKlMsUgHFN1S8RJQhCFdII+vWCIaRhUnBKDSLhSHb9PnMZMl5rM91ZoeFWRVFhT36OJBP8EWLlPzpmnvx3u+SQUZ/IBQmkSahdD5z54/rv4YrHG7cdvxo8DXv3QZ+ejtn7omYteyRtSI26sYVZGWLsmZVS6Y+dD3NNQGq0WrK9Co8aLPviP2JNd/u7ez/EvL/swv/y2Z2D+pwQqNZKDJ8ArPvRr3nI36d/5/MV1/5OXXuNR+6kxbNsOE9NQb0K5Bo5LdOexrB/y6vMv4w17n08vGAppzbRUL0FNNyIlgBT5+VW3PJEDzrQDYhwoWc1BLlcQDOX/+m/BQMoD/bzaiyiNACU8lSY55UBjTcZd2aT+aUgGkH6HvIW56aeBNAFNeUoiJKD4m35H6sdBAo1uEDt1wblkJmapNLq3XKyEm1KZaLlKfjL0c+RnGsOOK2D342XalKa5wFSaSIajA47tyubQIlzjHo5l0/P7nOkssahU+oVwWMGPfOH3qBE3kP02gEEwYHmwxsn2GZb6K6Cav5aiLWjck2Y668lSGMvI+YLGTq6Zu5AdtS2sDtY53VkgTmKOt06x1F/hVHuelf4qaZrS8XvSfA1HmZNAwRaHTcsQfVyxFBkpmQUN8hPZBYE/ie5vx+/jmHbGd0pT1c8rAruekgf+JIQLr2V9Zp/8W3PHkkhY9d374Km/yKGtF3Lz6bs401vm7tXD38dufWTrR5K5VP/ETW3DJDVtSA0MO8GeNIkC6+wrvrbmKFehUoZuDw4/gPO8fQRfPsXfb1T5hxveilkISPvnQ3wY98nbCOZtOHRoU/r+b9c/vu9m+Ud5HfuFBUy/CoFDsFjDmRyTzCzy+KlphtGYlJR/fvBG3njVz2BEgbCGLUfGenEoDbNEpaYZQASFQUkgGuTNXMvJb9cja/lPLvGQSUX6+eO0wHYwzBumliMBZ7gA4Qr4CXhR9nSAZCUgQcMgJ7aVkdIH8gwnIR9Nl5FRti6rnKaMnsercgIvjeUEbu4i07bdPF4vNXJVOr8nvRlX4Ve8Sk6m05IRuoQyDAF2lSflNn8AnQXs2ixFp0ijKAr14sXjKgV/ae5q2xL99vUEpj3usj4S0mzRLrA+bNEsNpRqn+BcUi+l4pWpe1WmShOMozHtcY+NUYfp8hT22jHO3XE5vWBAySmyPmrTHndxLVeRFMW8Pk4S4T35PZI0pTXusqUynQW/WCvPmSaTxTqjyKfrD1RDWRC+SZoyjMbUvQprozZzlSn8KOALR27gpe/8JIPKtIykv36lXCguvJaNC57NN+fv5rmNnYJfMZCMcPmrcMGrYHI3zUKdUTQmSVO+9qpDj0kptHn9SIKLT4yfxtnLm5UR4XpVsBOOlwtLaSW3bgsGvax8CVZr4BVJbrkbgLg5hX3RFqJhn+ArDwg3yXEVR+dhlXxlDXpEnxjAzt0453i4W7sE08d5UvUc9lS3sqM2i2VaPGXnAWYqU9A6BSuHpDu/5XEwe55C6RZzjk1mTB/lV26QQBD5kCqFe8PYVJIlZwcb3cjVUyLDzCcqGlOTJlBowEghnjVuTe8slfAAMk0zgJ3qfh55H0arUsZIMHKRrEX/tgCrDHEXoi6Ei/Jk4UDg5OVpmNknzxWOpZHolqB1RjV123LMR74GWy+G9rxcUdun8p5RPJbnK1Qls5nbL8Fl5Yh8TuGYRqHGRLHBZKnJRLGBY9r0g0GWHejyQ2JcIvwh1Tj3LI80FY+evrJ0Fd9lF8d0aFbrOKrh7jV3ECUJpzpniJOY+5YPcc7sedRaZzgwdxF+EnHrmbuwTVtIhW6JiZKUU45lM1uZYn3YzvhNuiyDNPPPbhQqDMMxbVVCGoZBkiSUnRJRKse4ozaXOYu6lsNt8/dyqrPA7sZ2djW2ceCZzwXT4puz57Exfw9nOktwyU+KmbxhSLk6BC75CRi2malM4Vg2o81TzMdw/UiCS5gmmBhYwwakBiQGdtkn9B2o1QSivrJ0ds9Cm3m5BTh0BAYKjFGtw+z02VYmipv0Pa8kgRPHCE+o/79GxIVeuO/pXLntElzLZaY8SeH4bXDoS3D6S7IZ12+B5Gdh5rxciV5D/5MwT/lD1SwGcoFqcnFwICP76ZRDc0QiVX75vfyxOvuxHMVU3oQTyej25BMgBQamgwSM7eQlkoUEmhYyJUqQ7MYlL4cKe0THeNjKU3ErhWADRg2p6zWgz3KhMqm0XmrQSoW2cdXPwN2fkgauYcB518DivcKHCjsQLAqiuDglx+9V2DBg4vxnZs3kRqFGVJtTSNoaFadELxgosSUTx3KwTFMN38xMWNu1bAxDtFOkH5KXIp7tUfHKOBhgSuZlAmvDVaZLkyz2Vyg6BWqjLix8C+pb8dKUYShWH5Zpsbuxg2I4xnAr1BXpsVGsEiiJTU2eHEeBUusfMQp9Njbp7lqGRUwiGBpTXEU74x7X7Lmae5cP0fF7JKncfqqzID2nJ/8XpksTRKsPMlSAwJtGGzypPJXrGttIUJ86h5X+GgYG7UDTGR7b9SMJLukbUsP4sJlaXQVIM1MML8SZSgmNrbjTI4Jbx7mZGuTZTLkMrfX878UiHDr40C/kFcF/BJoUXz+Xm/dFXPe0Gc4tTcL8vaLydf2vygYdIxuwhKjEVWeksaktRzPuTz/vregeCYba9E4unQAKPLaJGRyrvkY0JvNgDgMl3KREq0CCjlOHUBnRa36QgQQBDfQrIRnKUP2eUH+vIxlMgAQYj+/IWIDitAqYg7Obv+Udggp2yznTutTMg6nlwsU/Dnuu5pfu/iR/cum1WS/gv3fm+anREoxPbwqIgWQwKjsbhWPGTkBBWcXYccRUaZJ15V3sxwLZFzsOLawkzfQiIucgrGkpf/zIJ1EwekDKl3EHSyGJbeX9XXAKWXnVKNTYWZ6C03fCxE6OtE9zsnOGceSrrMdma2UaBusUvCoTpQZdv0+jUGMcBdgKMwMQJhGjUALO2qgDpApAZ2QeXP1wSMH2GIQj+sGQilfOpCYrrhAqHdOiNepw25m72d3Yzmxliva4Q8kpMFOahPHdchIsKWufI1+A9fOZndjNBVPncGjjxPe/Jx7B+pEEF4D0DYnhvX1/mgJGYoCdYHgBTtMAK4Zz98ApB9ZWzn7g6DuCxcrSw7+IV3hkweXBIwS9LTxz46N8/pdfwZN2HqDhD2SC4iObs4tc+c+ZkvHq5DnKUnQjB70FQ2n2hsNNU544z1KcgvQfLGULG/mKvOjnI97NvZhwJNMh3a9IQgG1xcrpMG3neBbdsNUbt4kERhPBrrjIWBr1N4W/Q5OcdVByt0oTLakNogAAIABJREFUV0+yEqBkwtw1Yre784CMi7dflivIGR4LtsPW2Qv4wsLdREnM03ddxd+tHydOYzzLpVlswJN+WSZtX3hDNuAiGsOFz4XVo2yb3iufR+jL7UEfuzyFq7yrwjhShEMjIyZqb+pAGXu5yitKw/VTJTuprTNs02Z1sIZlWDSLDYHkaxQwCC7HtATpmiR0+iuSNJriOR0mIcfaZ6gXqlRSmVTVvAp+PIFj2gyCERvjDq1RB9uw6AaDLJuyDJOSXcjoAKaSXCjaHq7l4Ccht5y+i/nuMqiSr+8PqHkVReEUIam9ox5HDANXSUOIbcga9I7K+To8KkH7wRu5bNeV3HT6ru9/TzyC9SMLLpD3HLNlpBjlESQm7lSfIN4qE6I0lat66Ivm7ve6hoN//z4Pt5YXSZYXeeH7wWn8DcF1HxRcwTKSHcwiuIG1g7Se8RGa/kDS5sq06OzGgWKetgR05g9UX2EkDV7TAa8OhWY+PbH0FAYy6DyoSZNPZnfitxW2YQxhD5KeIiuyaZOq35reU0GyF4287SJguhAJNhPkZZMePacIOtdXeJoEaEzB7ucIj8UpiqeQW1auA76818Z2+W05PH/PkxmlKWe6C3T9Pn4cMArHbIzafGSwQWvc4zf0a5lA/7D0ZA5fD0Gf1tRemv1VuYNhgVfDUZMgjSuJEimLDMPENE1ipZfsWg62YVJQGxhSojTOnAJrXhXHtFnsr2BZFgZQccW+o+wU8VQ/RQiUgugdtk7Kx6rYzmmaKka2BLqyK97MY4UaNg2LKI0ZKSO8qWID25S+h+ZJybRKviZNWqzYJTZGHR5YO8ZYlVdtpc7n2S5mYtJJe5zpLnJg99Wkp9eoFSrsq2+VMn0FyUaHqP6ZZNOzlSm212a///3wCNaPNLgE2qURSCpdzF6dpN7CHJUhMTGLkJRrYmJfKMLWrXDy+Pf+AtG/08z9XtbddxOWa9zTXeCyNQTlOAK2IRv01BGawAcPfpE373umbAotIdCZV4pqA2lWjpZgvCAb+sV/AV96CxgXyeuYjkxN0jQXcsqmRSoTikbyPFFfspZkDIkqvTQPaPPEB84ujabJeUObQQgDdfsskp3p5m6E8iy6K5ds2P703AHAMPnL099kS3WGnevHuGh6Hxw9AQe/xNZhS0Bc5SmK2y5h39aLOd5dpj3uEMURG8MOa6O2aBtr3a3XfAmKdfqRj331aync8EGa56fSp3HLGUParc4CkolYpliGaNYziA5tkqbYCo9kmRYF26VRqGfkyS3VWRxTMiDPcinYBYJN3K3JkrKU0eeQYbI87mTat1qg6tzJ3exubKcfDLCU15BnexQcaTAbhqFkPMZYRYvp8oRqKg9YHqwTJ4lyB5AvquKWMsHuklNQmjOieFd2ihn9IEpi4jRhobvC507cwvbaFq5wioLILdZh+X75Xsvqu7TLEI6YiWMJmD+E9SMNLpDbwDarA9qxjTmsEJc7WHETqxCSbJmDDRdmpqX34JUEmAUwuwWWFx/7gxx0edyvvY/0ug/BL75RrgbHyPk5f/lsnv3SjzHvFtl2/Guw5XLlO9SSgBD7Ur6MF+QxHbgxGvG0YQcKLTKbVX1yh+r9aQJg7KuMZyTNz6gLyZBMBjPm3wYX/aP7IymSudjquFMk0FSR8q6PBB9T3R5sej49XXcBy+UdrZPsrm9jqtTkrpO3YRomz999NdzzaTj+DRhtwGge4hYYBfDeDLZHoTJNwfYYheJHPA59PjixG378g+BV+P17PsWvlqd599K3mS41efsTXwP/+hEJZpEvo+zVo7jKq9tWQdgxbbQXkd787iZtX4OUmfIUE8UmnuVimiZFFYgiw6RREM5RlMQYka8+/02QAoA0YaSYzJgpda+GHwfMVaaxE5FR0BmTyCc4JLaUMlEcMVlqZPgWfR9AbDsiN+MZuWoCBRAmhpLQHNIo1JQ8Q5B5KJlq/H7Z3IXsOn2XQCTcMpy6BVZul+9eQw7sspxLGyeZ1FpEj/H6kQcXkABzzp9Npp1RCUJbMvlY5fP1CkZDUtV0MIZL9kO7C+st6Pcf9jkf9XXwfia+8Gts1MizgR7SBH32z3HxP/wMvPEWGByGtYoYUmXOjSORLND9Dxuedvcn5TmSEIoTUl6kSd6zSRXPKAPMBYoaYEi2EqRnK8gpcHD2O0Vu34xhgbxZC3lAKqr7GkiwMZDszFD3KarfJmA5fPbIDVzc2MOTt1/OL1z5ci4ybbj5L+D4DTA6kU+ePKA4ljKnNkdz+lxxHuwtMQhHjGOf0xc8G892WR+2uHT2fNjzZHaPWuxp7oCDX5Gsb9yR0bVW8avOgGljNreryZnIVmJAnIj3s+V4aHtb2zCoOSV57LGbRL6hvgWSGNs0aRaqEAW4wVD6ZEr8CSWZ4Ci3hyRNmCw1GUc+eyd24ViOCI4pRrVG9uoAIk4D0gtyleC3bdoESr9FfKx9yqqBbJu26NVYNuMooFGoUrA9zp/co9DFEZ7tUvMqTBTrGIbBga2XsP2Wv4XOaaGeRD4s3C7f9wTgboPhPKzfK+eZ7VD5/0vmotexn1833LftT2lI3p56Y8zQxi6ERCMH006gVCDtD8FxoFmHhSW4YL9Mi76b8dqjtF5zzvPgGVfC638TdiA9iyWkH/SEN8AHn6i0Um6n+6L3U/vkm9T0Yx2SETQuhsoWvrj/OQRxyGDvU3h5dYsSW/JkQtRf472nv8lvdhfzUXysSiLDkL5DnObIWp1Z6P/ri637HQevs5fN0CkdZDaD6wzyVFqfg2qgxdxTwHL4tQOvolGoctX2x7G9twpLd8ORT0rQt5CMqEB+dpUnwXazZqtmCydpynVf+zAffsG7CeOQXY3t3Ll8P4/bchHT5QmhFzz+Z/DjAO8r7xd+kmFIqVKby5HQppkxxGPVZE3DlHE0puyUsC0bq7sEa8eEWDruyO9iXexkQD7r/mruagAitq2b6sMNkjRha3WO5cGqyBNEPtgey/1VaoUqnuWyNhStYJn4DOkFfQbBkEE4FKIg4vgw31tmebhBUX0uetqlxbstJSxvYrCtNodtWpzpyvBi//S5bKnOUHHLTI27ooNcv0yy21FLwQgsmH26XOCCBRik0H4Qlh/Arkx97yf9D7D+wwQXgOB9Bw0A649KqREWwUwwiz6OFxKeimEwhLU16b9YFgRjzHqBZNsOWJx/zI/vDz9xgvf/+duxD/ymNMy2I5vy5C1wza/JOPpjbwMLap//DQi7InC9/YkCHqvOQGWG3/zE6+nHY44Fq8y94IP81pffi2UY/N6zfo3pyZ28dmqPlBjtM0qGwRAKQBJDsqmPpPVXYuSEGpAD57ZwNhlRN0x18NFZf0KeiUEeoHQD2ALc7VDdCZVZeOJreO3J22H3E6SBffouOPplwR3p55lSn4ueWFUmoTpLZ9yVja8PSUmNeuMe22pbGARD/DjgWOsk7VGHOyyHA/P3cnPQZ9+T/k+23fdP4jw5amWZB+FIpiMqEOvSYWPUpuv3mC5PUnHKFE1TgopTkAAdKR9urd8bR9KkdkvZh2NrtLECSe6tztEat9lR2yqMZ8shTWKKToFjGyfZ09xJvVClTpX57hJ9fyg6MGmsxKeky77QW2F91M6kLYU4KQb1huESxTFb6zM4pk3JKbA+alF2ihmNYK4yLcr/R26EE7fKd2YYIuaVxqLnUp6Dusrskmvg5Ffl3Fk+RG3yeT/INvie14+MW/TdVmpEMgWYmSdptjAsFVgKyg/IscEfg+2QHD6Fc141R/M+Rsv9sfO5/p2vw3nhtXzxF35PBKq0XOTq7fBff0o23P/1r4KDOXozbHsi7565EK54Ga85+EVe9o0/53ML93Jnf5FfvOQlxEGZZ33+V/nG6jI3ri5y5adex02n7mAQDuGiF/Crbol/uuDH4HnvlkB1wYvgwpfC414P5/+kBIEQ6Ze0kNfVPxq2rzlFm+UrvxP4najn6KjbQiTzsIDqZVA7R1JqwxRR6DQRYNbaMXFV3FBNdp2tVIHmZbD7RbDzeYIRGXcleKgGqTZaBwT13D5D2XKYMCwunb0Qx3I4YBe42zQZhmPuXz0MsxfA1LnQ2CFo6MGGHMsmaxULKFguYSxugZ1xV3AshiWlZ7Eu55Bmn4+78vj2fC6DkcSweD+c/KbSm+2CP8AIBjSV+Xtr1Ob+lcMcXD3CQm+ZjVGbM91FlvqrLPfXGEc+gZLHHIZjUmBe+QGtj0Rn5NLZ8ynYLoYC9KVpSj8Q7d6l/ipRErE6bDEMRBnPs0U2c09zB5y4XY65e0a9jzVY/za0D4kwlFOW9+oUBSax57mChzrvWZkcxWO9/kNlLnolbwoMAONDVmpYLmm1j3N+AzAIC7vhxCnJXqYmwA8wrERONt3Zn5nD2L2V9LY7H9kBFCswOrufE9x4imdteQusRjz/Z6/Dee52gs4ZUb+LkenRm54BE3DsbZ/knL97Cbgl/vXkrbx49Qi3tx5gIRjzycVvYoZV3vr1/4YVViGpY6QGqZlgRC6vvP79bPU89pW30Q761L0KT955FY3LX5eXSYN1uQLXHVgMc+3bLCUAjgLnkgeVzbelD/E3i1xQqqwfZ8pG1FwgrXznD3JY/4nPS3lVOxd6D0J5uzQPp88XakI4zDyvE68ifYtig1Hks2a2SUl5x6Gv8DvnP1sxog1ct8SByjScuoPHNXdxJBzysqlLBE+khaQw8oa2dlBQUdMzLOaq08xWpjJP7rQyjdHYKhlKMJBNV6jK59ieh/l7BM9TrAv5crAur9c6LVOq2hxUpjDiCEvhbCbLE4JDLNTwo4Dl/ioGhtKCcSFVtqmWw2JvlTXlPlG0PfZN7FJHLJrAYRIxDMf4cUCzUCOIQxljxyPFmRLw33P2Pk0C+7gPrZOwcJN8Z6MzeV+s921o7lVNeJWJ2Z5kLmmCYzp84Bs/l771yX/+mPKL/kMGF73SN8YGgPu7W1LDSkhrPexogrh8HlYhJI1NTFeMwcyrLiS5+W7MJzwOuzomuP6eR/7CDzXC9kekH1dNimGP8HMj+MzfwO++Wng7CaJdmsI5v/USOLAb6lv59Ms+zOP+5qUs+gFmVMYMCxi+B4FDGtoQmySKvGl6AUZss5B2acVHOK+whaOt09x25i6eY3uyqbV74mANrDoka3JCbRZ9Arhw07838430mHozTaA5AVOXw81flUZuRd1+1Vsk3V47Kvd1ywr050ipMH+nnEHnvkI2rV2CyX0CCtRmcE5RkRJX2L7zAEEcsLu5nXqhxvpQvJ1XRy3JXoYbopdTm5Hft/0pXPoqXjZ7nmRJaSJ4GqcoxMjqTC4Mnr0ZIE0oGlZeBqUpsWVg1eYkWHjVnLkdBVLOdpel3Bp15LYkEQDkuA9b9kNzu9w/TTHSlEahhqtrS2UhovlBmmJgKc+piWIDx3LoBQOetOMKwkTEw9eHLXY3tvHgxskMAFh1RcjbMsQKp+wUBe0b+tJYjkM5xvUTMP+v+QVB1yAmUL1YyV72FLF1AwYrUNoC4YhttTmev+8Z3/+++D7Xf+jgolfw9sWzIqz71ovlLDIAIyUeeBhGiv3USzALIzBTrCdeTPzAImysqnPu30D2Hn49HNkx2qSMHkcYv/XHpC95Kfy/n5BMoQisIv2O1ROwcph6eYoPPPXN/PQX/wBzXMYYlIgHBeKhJWhjLcJkWRi1Gk7DxjISBpaMozfGXY62Tkmqq0fUcQDr34IH1iSo6MkVSBbhAfcCB5TtSBLnU6XNS0suOA25sr34TRIYtK5vf1WyE0vhdgrVPGisHoH2N+HiX5BexagNc5fmGB/tLQ2C4Tl5C8XqDF5zK0VDVOT2NnfSHvf4g+lzBd17/CZYuEF6R6j3cccHYNe1EmycgijXz+2XrKKr7Fwn94g6m6uM6LRbgkEGRLQiX+nGKCGr/ppSwy/mn2lpQrk7qIC180ppRs/sUzwd8WIJlEG7YTk46kJU9coZlUDb9Na9auYouau+jXOaIimyMWrjRwHrtBgqlwnXsml4VSzTouqWKdoezWKdulfFMgz6ifhQf2v9KJe4ZWidgNGKjJl1OeoA3rSUgEEPWtqoT8EY6rvAKTDrlpitPO//m6zoH3QFH7hPMppfvzAFMAshxII/0Msqj7EONEjGM0QbhhKievRsEwC44w68iT7+j50H3z4so7/jyEY/CST/CLP7ecmlP8ErH7ief7jnFFG7TLLShnZHgkswVs1Wk7TWINi5Hdep4AYF4lLEIBqLl/LMeZIOayJktJpfrfSkSL89/bu4FxJ1aYtHMFw8G2QXINiW6naZuvRXyLyUYl+JOw1Fj7W/rCQeFKBv9RDM/ZhkD1EgmzAOJLvRCn+Q0xSUSt659W10I5+SU+TJu67klUsHJTMatWHphpw8aSC9oxQ4+Rko75Nyy1TaPu0FaRQ7xVx2VGcSxbocj5bgNExBRWt/KS32tXJESp7WaXnPWrq0WJfAuuViyZC0hs7yAzLGdoocXHuQmfIkOy0P/D47y1NMlyY50T7N+rCVTX8iVbrZlo2tPJFs08IHSk6R9rjHztoc66M2Vbcs8gqxmKWVnAIzCnQn2i4Oi70V3KndnP/j74V7Pwdf+f2c7xUjkhhuUw0A2tC/XwKQCVS2wLZLuWPtKAe2Pu1R2gQPv/63DC56Be+/X6ZLf1hL7dYMRBYUx6RGipGYEFuYGLhzBlH5IpJb7370j+ErD2BP14nOm5WGGYf50NPegGWYvP7jfww3XQfHbuRvXvwBqu67+ZPPLsLpM/820CUxtNeBlKBVxb3I5b7eCs+fnaYz7omS+6ida/LOXgNcDyeQk0uD33R2YiNlSuKoE009TsffACl/CrukTDBQU5ShSqk7sqEqc3J/t6JkH4ay8fx12H6l8ptWUa5QQ8AmCviXqChhuVDfCTf9MQx/hlqhRs2rwP/4eZU5WIJO1oFFc6NMcpuT6IhSyvPFXtYwoDwr2YZuxNpezgPKpEPVG9YlRaEmfy8oDV+nKPcftfPp07GbRRNloKZS9S0SwPrrcO+nccvTXHn1a8lM6+bvgUKNolNkV2M7BtBSaN4kkdF4FEc4jq2CRpFx5DOOfBqFKuvDNjvrW/Asl5oyPwOYLk3ix6HwnQwTyzTxo4AT7TOcP3WuiG8deInAALR7gw20D58NT2gj3LL6dkZOiXMn9jyCM/37X/9bBxe94l/pGgDO+2ZTI3JJvZFoFg1q4ISkY4+kNRIsTJrICah1c3/Q5RaIP5dy7stjDr76b3Fu+WseWDvOn33lFK///Zv4o9s/xi+fuQs+cCXv+sUvsjh4N586Vn14z+z2hgKLCbhkGPl8+sj1bKvN8tJSUyYzQV/eR+0yuDiG3n3SK1kjJ1fGQOcuhX/wBR2sx8xqL+IAkxfKJEgD/rTjQO+ENGejsRJ4UixHX3EFdl8j2YHGgaCIjdpSBfUaWq8mDsGbgFt/D8wirK+rgDiSq652JNDcJ5AyU0/EdBLfPwg1VX715mX8apjSjDUMmSSliZRiUYi2gM2OwVeWt5HiaS0dhG2XwtZLJYNKYpHYXOvK57B0P6wcFinJJJYs7PQ/C7Vj8hy4/KWw4wqG4Yj4f7H33vGSXVe95/fEyrduDh1u56RutVqybMlBkoNkbGNjW5gwwNgYTBgyPMDAwHuAhyEPPDBv3gOGOM8YsI0NTsJZlmzLyrFz7r657r2Vq07a88fa+57T1/YAplsW7t79qU9V1z25zv6dtX5rrd8K2nSjHq7jUfJLLHWWtXcmNU+VXBnf8ehGfS0P4RCGEZ7j4tkeeS+/VoyZc32COMC1HZa6q+Q9Uf73HI+Cm+Po0gn25Mpw9L1yjXQXXDlPUgXBuv6dPaA4RKPfXKvNutLj6wJczAjfPi8g81vjSnRpPSmGtBXuhE9U2APdnoS0G00482+oU/pKY2oK/1DMuZMl/Fe9Fvs1bb53+21gwc9+4nf57T99jJ/dbdP/9l9lvFXjrTe8kWe+7fc48p5xqC18+W3mfHBilBVTcHOMFQYZKlTTFhyxdjWMBGZuIyQXJTyuEJesCzwMHHo8naAma/bOd8CHfgmGDkgS2cCkTMj6rJB/jZOy3aAhxZWRlrGzrNT1Mapzusth2gJEpWJZ5vsk1lGnClQPCB+Q1OR4bNKOA11SYtqULZhCSoNZCqg/IdZQbpNYZ4ZgdnxozIr7MrkPEguaS1DdkOaz9DusdVNIYglng/y9syyWWK8pUhKtBTnW2sNSBGsian2gcw/U90B1iv6eOzleO81YaWSt93SURCQ6PaLo5Wn2W0xXNzJVHifSvYo822W2uUA1Fg1gR2vQ+I5Lo98iiEP6UYBtOeRcn9Veg13DW3lB0IET98HRf5SERfOwiJH/Z9MNXLQ8yCh4BWab8xyaessV51vMrr/uRvizCxZA7pd3KCIH/ADbifB8DxX4REsRLCxenp2dPQ17dxF+6iiohOTv8tz31if41pfs4tGFo3DqJMEpyNl/ya7pDk/d/U4+t+0Ofv11fwf3bJQneBKnZLGXkxC7XwdbUs8dy2bv6E64/4+gcRxKG3UynQYXtwLFaYjq4s5MI2rvTeBxUl0WBewGzj0kk3bqRjjwjRKJiXT9UtAUbRinqrOLm6x1fkxisGIBGMtKNWUsMtnEAZeIlRsBLScHdhf8Cliu3PDLpByQacxmwCU7sgLhJjHPCSA6peUipmU//iCELXj4L+GWHxDLCltAszwm1owHqJwcu1cUotaE1SOjY9wWC6e9CL2alssg7e3URML11lFYPEnuulcDivnWIvV+k6Kbx9WFja7jMpArs9JdZbYp0gllv8xIYQjP9sg5PvV+E8cSIjhKJMdrsjxO3vWZby9hK6lPum5sFzcObYaP/w6c+YgQ34F+5fXLHGdRvwL924dLUDvFcnnkq7/X/43j6xJczOj/8kkhfv/PTcoKfKzIlYfikEfsbYdnDl8aAfoqR3DPccmNiBIIehx+eAPHdj/BcD6E6jDUlwk+fJLDtx7i5blf4tapA9w9fgvBd0Z88JMTEARSK9Xrw1AVbzRGeQFb/Sqx1mbNuzmoH4fjwPRFrcVigeVLHZblgl2EYhl6F2ELkoPT4tJUyZPA8gcki/bgN/GrD72L/3zda4RjCDqShKUM+ZEIx2K7kBvQesEOKAeUldEMRlsymshVirSNrSXrqyAVLbfdtFlbVx9jlpA2hG6bFBhN7ZMJqRuLxgaic7KeezYNt6tYIiR+WVYKOkLUZjsr5MvyuzXnM+DSFXdJa+7SPZWG7tuIZaCL1gmBJXmoeI5HL+zT7LXou30GchXGSsMUdNOzKImpdVZYbC8zUR6lrlXoWoGIQzm2jWO7azIOju1QcPNs0x0iu2GPLX4RnvggrBxLJTVMWxjDZ/sIoBipZgPGMdBZZqVn+sVc+fF1DS5mBL9wwcq9Y4sCsPwI21JYjke0aTOsrEKv+9WJSmVHFqQeeoS4dz2LrgujLVHLK+ZJztf4VGuQT9lH8Dd2GNwwyx+97XsZLlT50Il7+bvHLoATkxQWSPwWQ94E9bBNMcgLuBg5hPNI6YEu6CRuiliUZUOiNMEJTEe6b7A+LmMRbJmCYJb3zD3DvtEdMuEbC7B0WIdoEbDCAhVq2YiWbi+rQ82gk7ZsPeGz4KKtlliLdhtZPMeXSe+4KT9gXB4DgIZzyZF2IQhJyxwMYW0qvI1LAKksZ4xkBW95gSarbR2m1q6czn2hq8WD548KaHZXBRTN+YVN2b/KvBKkitxcz8IwEUrnpjgU/QIlr6g7MVap5MqEmpS1LBuLZE2eodFvkXdzuqtjrAFFelJ7tlg+lVyJ8eKwuMSHPwYnPwH1k3Jc+udf46rM+fvIvVIgBZkYGN3JieWzX+7uvSLjqgAXgP4vnRUr5u3XKaUQWc0dOeLmFpLVDhw7enl3+JTW9K1U8V8xQFi5iFefJDgew8UFgieWWDhwPU9sOsrr9rycX3v5T5F33smP3vJmDnol+gMT/PXj7+PBmafYO7KN0uF/lqe8eYL3AX9MQs1xW0BA6ce2pdu8OgWY9sHOyaTxKjraMgAq5E1T18Ppz8OJz0LtJMw+mQpHqVjWiwPhNXorul2sBg/HA9cIhZMWVRqlPfNKlLZWbIiTVJvGRgBE95VbI5oNeWssG91kkhg5f/OkDrhU69dYLGa9I38r0aPx3brpejdV+zMSFl5O91VaSXs/hV1x4VDQOJLKahhS1BzzoN73xhtE99bJEcYRA7kKA7kSYDFSHMKzXZbCriZoQ0p+kcnyGAU3z6NzT1HxywwXBql1V8i7OVzbpeDm1yQYRgpD0jzu9Bfk92kdTQHPAG5Emu+kcXyNgzHcFUBrkeqzJBQFVxG4mBH85jOW//brlJXvQS+HU+nhlB3C2ji0mv9+C2b98HPQz+H40rrE29MkrOelFkgl/Pn9Z/nzh/4HWzY2+V923sXBoAef+xNyYYe3NU/wtn3fIeb9h/8PcYUU4vMXEPBQiQYWbQpYvn4hldhKFxh5FShtEAuktwjuAHzkfwcnLwBlucKBmIlkL0l9iuXJZLP6mo/pSFp/zmgzaGTQ6e4iYpUBGBMCtnUnNsuRSZ8bgtJKOgkCpMoc0m4EbX08pi0KyIT2uTQ6YtwonTMI+v3UhySNf/pmyXnpLOsaIv04r0xAx6j66eN2PNl4EqdhccNhGEvAdEbY+AqYOoBlWWsN76v5ihQjamsmiEM6YZe8K43nC26e0cKQZO7mB8GCzdUNgAhQFb2ClmuwqeYrQjBfeEx6PTfPCziaa2auj8pcI6N9nCMt/fCAsRugPCZ1Sc/SuOrABQRg3N8ZUY5dwbIUtIs4u6eIZ4pw9szl3VltgeBRF8an8Dd2SAotCclu3Y573RIv3TTOxxcusGdgN+Vcic/EPWp77uTugSn49e+Dk/9Nnt4RkqswgNzYF4HkGWnopowDbh7fmr3HMUrqAAAgAElEQVS1c2D7OvLTls9xH4IL4AyDNSwEa64iFbYrevUmMA6MtPWNvCqV0eEi9FxQu6B+CsYOyj4dN3WHTBsUowdsokWJjiA5nrwG90HvczA4Bt1FmRw6I58CciwGdLzM9+jzN9aLiTgZgMk2cbM8IbhXzgugxcFawiKDGyUq1FrQHTO1heXmwenINRm7GdyHLrWQEgRchw7C5H4Y3kKSJBS8PDnH19XPAY7lrIlDhUnEQK6MZ3sEhvC2Haom36cxx2R5bC2718KSJDzds4nWkhxz1JLjDxDye5nU4su6bcbCyiHAWCrBwW8h3PMKNtVOXYab+l83rkpwAYh+uiYJeL9fUo41glPogVUlvhIu6dwMzM0Q5m7CQ8ELl7DCPDeM5fFsjx/cfTv7RrczXhqh0W8xWR6Tp+oOYHgTPHVBJB58ZNJMkCaYRTrqZemmRYXNOlTdh7glAAMQzkFzJSUnw2XwlyEYlhYhJo3ctHddIq2mLgH9Cxqz+hDpZMTORgGKXCXNkTGq/0kIKiM2brsyeR1PJsrARlgpC8j4I9BfEPBLOlBtpCBxhhRADLiYyutl/bcb9kPrabArkN8gHEoE9OYgPyZuT2VSjq1X18SyLVxSvirf5yvQt1K+ys3LOeSH4Ia74ZF3y/UNWrL+xD7YcjPoBDfTLUCKDGUb3ahHrbNCwc3j2i4lz2elWydUCf04ZDA/QKkxCysX8ba/UKwny4HaKfncrklovdeQa+jkUxfRuEJt5KFjLBhjzZlUFhuo7IbJfXjOrdb+8Vu/qlv4qxlXLbikQxFXFrGiAnYnT1waSHsiXe49HT9L7G/kjj15RvNVjtXPc6RxluV+gx1Dm2n229iWTS8OOBp22POGPxCx6+i7oNaRiW/6N5tG8mth20Bcm9KkgEscQG9ZQtaWDd4E9OfTG9D47AXSWp5J5IkYIpaDScDqIZN9kNSPHxiXPBjPsIo2a5ozkJK3xj3yyzpyYxZ3IdE9pd0CBK7mirQVZtTvjAtkrBP0sReQtigt4MU/qoXQdYJc7hNw7os6kfBJ+a6oEwXDrhYW1323R3cI4PglsW4s5H3DAclELgzysJfjebvvlOWCjkz84S0S3m7XoDwqiWlJhK3QYKPoRiLnWc1XKHpFyWOx7bVGbiPFIcmzWTkLJ3Q3TpPL1FyAHS8RiYlzX4TlZ+TaZJMg86RcSzbRsI9YLSZHNOrB8N3PSm5Ldlz14BL/REeXEFQUWDA8dMXAhV4Xp9inG8WMatmBi51Fnmyf5Q1JTK27qmtPXM6sXMDZ8gLOrZ7n5fu/Gxb/mzzFQYsDkYZiDchM3KFrfCJNxOZ172l9lxm3QSEcQoQUuk2MwPzx9G8Deh8d0kiN8bhs0pYkRuPXuEKmBicJdYm/BhfH1y1ddeQmSQR4KjfId70ViJd1SN1P0/ZbpLoyAam0hJk0hlcIOxJGVgkc/4Rs2yPNj+mcg8VBqG5mrQe1pyufK6MSojaggRLQGNpMd2KvCH/PH4Etzxf3pHZaW1+OFE6aOqTSiFQqD23G8fIkcUQQBRS9AkWvSMHN0Qzaa1GgolfERl8zy4G5p6Ucod+Q323vK2F8p1Rl73w53P9psUINt+QhANtALDgLcWUVaZTNeMitr9DX6wqPqx5czLBiX6ItrvMvL/zVjqBPcEbxULXN9SMxYRKxd3ALN47uAaRJV8vqEMQBN0zu48PHP00Yh7y8W5endJ40n6G6SxLqzNPcQkx1w2+EPblJ+03hHXqZ5UwEwQOiJkw+HwaOX8LPrvEMJlvWhICNua368iSN+xgdljUzSsXSpSCJ9TLaKukPCLGqElh8BoZ3CdEbd8WyCmuSD2PZYGuAMA3bjCtgsmRNyLoM3PtrcNMPyL4rU3D6H1OXTiGTUimtPOfI9WktSoW1kxOL58Jj0KlJhXVhkA9dfIzVbgPP8dg5vEVLauZl8veaYrH0GlKXtHpRwLS1KNbL2E5ybo4oiQjigFgXLxptl7xp4haHAlSVcZHQ6OiswgPfJJbT/DGpaTr+Sf1A6acRIYdLyVyj7UzmWpncocsco/jXjmvgoocd+Rr11+sSXMaRxHDuPKGzlT89vwC7TvDmrbcwWRrhbH2GyfIoA7kSlmWxoTJBO+jw87f/MPzDfxIph0HkJqqMS4UriRYAimHT7WIFmByTUIdcHY+0h/U0tM6lN6GTg/4MLD6tiUpScDGkIFwqj2nU7XC0pWLscpWSokksESx0Ml0UC/cRGDcoD6tHhUMwbVRUBKqX5rJk9WmMKxBljge9zMFvkG0+9R7Zv69DTmZy+chGwrb8PepqwAvTeqqF49K3urUgFtDwNLOWxWqvSTVfBgvON+bY3FkRayfoiHUY9qU6uzEnQOOX4MLjAj5bbqbsl2kFHfpRn2quTHENrZH9q0S201kWsIq60F8WRcPls0Lm1s7AmftTvs2cv6vPTUfQ1yJt5nfKllE8O62hv2RcAxc9rCBPEuu79kqKffe7cPoMCeAPTNPbLA2vTtYvUvQKDOYqnFm5wIunb+YN+14JRz4ulodp/TE4CEP75EYePyhcg60jMAMTMpl7TZ0VW5Ante2yVrzXOJf65pYP9KF9QsxrI4/pk4pyA2uZnuazi7YGdMHP+rCzARWlLRel/apIlwu0F2RfK7ruqTAmxLSxRvoID9QjTabLk1b9mkSxHLDzDvjoL6QTzuS6+KTuY7QAdV3HZa+hoxQllsdkEge65iiQlObhQpXp6gbybo6bFOISrZxnTfLCdgQQHFdI68as8DpBR6715D6GClWiJJI+QWF/LUqEZSJrkUg+nHlA+JzuAtz203JsrcWUIN9+O8zem/4exnLJI65rH0mWnOJS4DVW7mVWGvnXjmvgYkZsY7kJ1uQIanX1ynYS6HdhbBKV2LTDLu94+m9wgkHiJGaiNMxSZ5Vmv82+k/dLub9fEaulAJS3iYVSHBFuw9MdAVUM5XGdBKafkF1dxeZ4sPA0rDwiE7SFJMpdbKYFgQFproRJFMu2e81qrAAwK0WNjl4o6UNsgVPUeTeRuDgmh6a/BLlRoAurj6SFihEwtajzS8I0h8MjrZ3JkbpxhvMxKe8f+wWZYIZDMpPLTEC4NFHPiWSZ9ik4vwrVHQIW3UWJrhWG4KY3cffiSZ5EGqYxsh0ee69s1II1k8rVhZKdFYnuBR0Bi/YinLgXSiOM5UpiDbVr8kDIaxNjcq/8bp0VAaO4L5bc1hfoHk1FmNovVk1lDObvTcl4w30l+lp0kOhetmDRiKsb7uVrMK6BixlODJbCKQREpRI0r2ANhpeDRh2SYea7y3itKQDuPdXEtb/IXVtvZa61yB6vIBm0rfM6x2W33IC5ik6hj1LxJNeXDM4tL5C+O0kMy+e01KEmd4tbITwjv/oyaSKaIWwt0qxYQwaa8K+xXkyq/kVgcz3zBwVxXUhZFUudU9JNw9BxDLHWYTGFdudILY3xMAUvY6HYpGHoMin3YyJJIVI/ZSiffOYc1mqMSCdlNl4StSG/UdptuDqS5Y6L/MSZB8XVmdpLguJI7SR7/ZKuBkfAKNAg3r0o1s7EPvkN/KJYOWc+L79LaSzVivEKEmXKD4jlMr5LlqmMSaqBqb2ybHlQLJ+V361QTUHFuHst0jKJjYhmizZEgXRm57jmFn2th7ITLDtBxc+C1kUcQpgQPjbPI+MWcbHJ6yau56PHFjnTnmNAC1lHN34z7n1/DK1RyWidPCQTNz/AWoFgFOjohQdbb4XZpwV0tr5AyxIMAE0Y3KLT9y3gdOpymMltnoZt0pT3HmnGp+FdSvrzMNrt0JE1Y3EY5jfOVEzHEdgJBIsC2onejgGABuLyrRXYkXIIJt+ljzyNy8DgECyvpPxCV2+vSwowFqnVs6q/N3U2BmzywxCVZFLnBySalC+LVovlcH0U8rHWIivdOucLVeZbS3zXrpcJuCyfFVdm6WnIjwrYD0wKMMw8BcuHpcyiW5ffzM3LMqsXwVmQqNPkXgGb9rKUWAQtYqVwbDvVkclXNTdD+jK/UVefX5k0TcAkzumE6EuSD5/lcQ1czHDFMbV8Xc3ruKnQ0eUepg1KEGB3h4k+ZvHRF9dQXsSpc2Vyz/cpenkW28tMHXojPKo5jcq4JHuVx+TGs2whEh1tvcwfFdNcKajPSRZqaynNil0LF4cQX0gboa0/zWxCVrai2lgIkEaSsjUt8KUmuDHfvQRUPQWuEKnMXkImwwVkgjSQZSIEYIwVYjpFekjouoS4BJumhP85ckbAp6GXCZG72xyfkR5YywsC6odh+AYY2ysCUb5OrrNdcFwuVMaYWTjKtxx4LVES88uf+n0uNOZwLJttQ5t50+oFWD0N7mnYdhtrPaY6SyJxEMyAcwgmr9dJeTpTevWcTg+wJAJlfr+xvThGuqI4DJtulOV6rZSLgjS931h2XSSaaCoyyqQha9ad87M4roGLHsHPzVq5n9uncBLszeMknQ7UV67sTqc3ES/68hSMquzYEHNyoc9kZYyc46NIpEXn2C5Z3ssLwAxPiw+v9UGkYFCJC4QFGw8ImGQrgeszrEkhRH1pRWEsBWOVGHckGyX6cqlX2fB3su49yqyzxnOQul9mfz0EDDYhwNLM/N0UZxp9ErM+mWXWUt4jKeDcURRr6aRer0Vae2MmnEk+MzlCQV339xnUrmSU5gglMZsac4RJxN888QHefOObUCiWOitU8xVq3VUWb/sBxg6/DwqTaWZvuya9sqNuOqkP3Z3+Dof/WVyk0rC0lvXy8tt4JS2A7ujK6w2yfLsmBL2LgEj29zI8mQvMk/awGuJScvcauDwHRi6Avo9T7pEUi1ceXGYXiJsluPFGgjk4UWmgig3e/dSHedXOlzBcHBR91sqYjlC4QvAaVTXblSbtOf2o8oswtlN8e8uWm7PqiaVklOL6TeEXBvZCdCS98bIWh3GDTPNB+PI3qCklMJfJtCQxVoOpb1km1RlxM3+zMi8DbgVSRToTDjcSLIZMNndtBMSLQqAaq2QEcYPawH5S/uhiZt+mOFMBi4+KxGWhKlmxSZRWUDcXedueO+nmylhY/Norfprfuf+PGS+NMJivMHbyczCyU4pCLS1K1WtAZya9Np1Z2bal2eXKhPxevSacf0iyhL285NgMTGprWT8w8mX5TT/0Q6n1ZUYDAZIZpLVwm9SyM+AL8GPqWc/MNcP+lxe5eobytGuUD3C2jqbh2ysxLFueUjMXpdbmyaeJ/rHNLx56nW6IZdOLAigOYXoSr0Uazj8mfMrApKSvj+2Arc8XeQFfK+Sb/BZfNzQz4WivoHsP5S/lUrJ1KZBaIWZk9VaMxRMjE76GkLOryGTukIptg7g/TdInreFyjFm/PsrjAjfcJse06QUpcPRIwcsk1plJ1iRVsnv198AufVwt/f0QaUq8yryac1p1TktczjwBy6ck6hb1oVtnqbNMK2hR7Lf4z/tehWe7jBWH4eJj0D0BC4/CY38BM49Lpm04lya3tWbg8ffr0gJbwCLqi7sT9QXMuqtw83fB/ldr14rU6owCydExnRosfU6LiLWyAhxF1AeNZTaur+XXEFjgmuVyybAA/BCcGGd8hXhwGFaWLv+OKoNQKcP4qOjonj0Pno9zaCfXj+8h7+YYKQ5xbOkUvcENHOy15KYMOpLJWagK7+IVxBx3fJTjYoU9ndzVlcQ1W2eBqozZYSwer5g+xde7Hdkujeb/2YQ2Y2VASrRmBbbN381nG3m6niEFpgQBoRm9HcPhmKzbBz8r3z/9Rfm/EY0ydTMm0c8IaKH30wcuPJCmxSvETSoiADiaOT7DBz39V1DdL8Rr7SFpRTuxD0ojJKM7iFbPs9xdZdAtwMJRvmfhOHz+t2X/N/4kHPsILB2B1j+l1yoL2LNPCh9WHslIivYE+Ed3wun7YPomiCMilZBg4ds6sPD3bxVA7JCCpakYfzkCMKv6/HqI5Wdygb7G45rlkh2RCHpjgXIjGKpemf0MVnG3D4vlApAoyOW4+/YCjaDF1sFNnKidYbXXIEpiDo9uFVDoLIu/XhmXfjpenq7tcKo+w7Glk9RC6Z+9lsAWdOUmbs7r1HIrJaktB/wNKb9hLBIzzESHFAyizLshZY2pHpJqn2RTz7MKchtITfY66STJprRnQcwck7F0zHF1EEulnTnOvv6uDpx5WsAlu/8Q4XdGkIl8+0/Ca39Xtv2GP4eLD4ibsgIsPC7XZ3AjjX4TC0u6Hf7Ba3h0dBtHnvftco5NBDjivuxj80vh+rfJNdC65QxuhqUn4PA98NDfSNLj8mmxmFbPiU7Ltts435yHU/fj2q50cnzfj8OfvUjyaCIkOvftvwS74V0/84sCmA8B9wMHEI7FcEk+8MNfW6sFrlkulw43gtCFxAJL4W4qEl0J+YsL52Dbfmi2oFgBz4P5Wea6yzw2d5ibJq9juroRT4s8t4I2jxQq3FQZF1fHsolsjzAOudiYZb4t1lXRKzBiO9BcZK2PMpZEjpJQ8jFiLVupEqn3MRPbI20Nms0JyYKHMbuNlWImr7FiWqQgYIhdkxujSEHK7MdEgbLWjukEYHI2OqR5OTn9f3MMWfLXQSJPG/V7tnzAWF+mXmpou3Aff/+f4Mc/mnZdDDPrAAxM0umKoJXv+NCGRr9NnCTp8R35OExuhzt+RtYpjaQWlYeQvdO3iLVYGoFnPijV5P0lKRrtL8HCJ9m86QbhbOYOS7h68UE5hwbU3/5Jqv/zm6X1yYaX8B1Kn+MsopX8BX1uJor0NXaHzLgGLpmhnEiaqelhWQpyhcuvTud6xD0Pq+rjbpwmfPgi7N/PA0dCSu45Do7vwbUdbK0VEls2Sin6Q5uJVUI76NBtzgHQiXr0owDHtsm7ecmriHrSwCvJ5H13GwIuSaiTwJrp3wwImHwSM0JYE2Uy4GNu2x4ZbRjSSW54GDPBjBuTtUay65kcm2xUqUPKyUSIhWIqfddvx/BGMakEgzntLPFrLK4ecO4eIcZf9Xb401cJUFSq8MMfhD+5DYYPiaVYn2FscBMdU1X+js9xR123Ltn8KrjwUdne0il4+Ldh6hukj1KWvyqOimzGzts52Zhhx9474eIT8NQ/wfkPppKV7/9fYf/bpKvmY38q2+0BY1BdvQD9FfjAu2Sb996nLSWEa+np67wR+M/PDWABsFTWH7/Kh//rGxXKwoo8lN/Dag4QPLD0lRuYfbUjV4DNm+DEcdzbrseyE8Ljdf6vn38tc60lJkoj3DC5T3RA3AKu4xInMb5ppBVHdKMeFhbdsEsr6BCrmK2Dm9lVHhO91b6xUvopORn1JSphOip2Z6C7lAJKnRRQskCSLZgzXIIBCGPZZPNIzMsAlgEv48ZkE70qZKQ1M/swxK6xjEymrjkeU/nbRTiZFpcCj0caHTKJeKZjgLFg8sDtb5fOhff8krS1LQzLNZo6ICUWlTGobuCdxz7Bj9zyFony1M5InVEcyqvXEC6sfh42Pk+4rnv+ME3oe9EPcuq6u1jp1qnmB9iJBcc+BY+9M+WpshZcpvkCu14M5+5PBbk/R1pkOosAyhTiNlnATzx3wOUa55IZyolAWWLB9PPgRTj7roDmaL8LJ44DYOcDok4Oxkf52fvew2RplIX2Mufrs1hY+I6HrZtlRUlMPwrohF06YRcJWcq9ZGHRN1XQ/Za8WgtSu9JvCufSWpDPvWVJfw8yZLXhQrK8R9ZdyZbxZ783kz8LQAYYbFI+xHAQFilwKYTjsDPbzLpQJjpkpovpG7Sq341qXk2/VkmjSJCCGqRlDdltdIEznxNe45W/IglunZq4MAtHYekELJ6Ek/fzI/khHpl9SvJOikMSmRvaLOT6yDYY3gorD8EDfwT3/6HsW/9E1E6wvTxOJVemmquIDkxzHko7BFRGtqdWmgHrg6+Xa3jf/QIkRpNrq77WRkg9jwC0x3MKWOCaW3TJsGIXLIVyA1AWVqKwnCTNbL0CQyUWan5ZOkAOTPH2T32QDaNtfqhwN7a1DwVU/CJzzUVsy6Ib9Wj0W1T8Mr2oL7SF42FZFoMmVTzqS3izVxcwMfq13SVpGlY/naaHh6QT0FgKWQsAvvQRZCa/+ZtNmngHKYGaJWnXvyCtezGuk8kIXm8dGbcnG4Eyx2ZCzZOkYFMidZds0mQ8Yw2YsoAImP8snJ2WrozLD8DICyUcnETQnGHx9b9LN+ox/anf46aoB5sOgepDY0bIcaWEI5l9HOwSUE+zZ03F8umPw7Hb2b3jNuHMikOw8QadGzMhF+qG74CzX4ATH5fzO/oBed+QOW7zO1QRd6hL6oL+yHMLWOAauFw6LJ1spuw0MdVWMDYOC3NXZJfheQXnzgDwqudv4cNzq8wsDlH2S9R7TWab8wwXBulFfTZUJsCydCsLX3+2yLvS12iyPC5lAWFPP4VXJJIRdYSHaR+VJ142k9ZMZgMQWW7DRHJMEWN2GA4jz6U5KoqM5gtppMisY4AlCzDmb8bdWZ9TYyJGJtXdQSyVbHlAjdTlyR6PcbfypJySOV8Tuj75Hth4lw75zkkdV/1R6CjG/vobROEv6kq9T+20hP8n9urq9CG5Z5oT0N8BnEyLXkdIQ+VBR6xHNyfvXkG6EkTXS87SzpfA1lukluip98p6FX09VhBA6ejvTB2RAV8jU/ocG9fconXDapWxloelujd2QVlXDFgAmJ2Xd9djwC/xjVN7iCtLREnMUwtHmWstMd+uESWx0BWOz1C+SsGTCEfezVHNV6QNBUqyPcujWrckEeK2MwONo2lnw+wkN1IGbuYzpOX62VBu9oY2BXF2Zjt25v/Gcshm6hoF/az2igG17H4hdY/MZ7OsKcIbIrVkzPEbAMnSiGZdsz8DqOblAd0+zD8k673g+0VXJVQCZMvAzGdEWGtiD7Tm4Oi7BSymrpPr7eZg4yHY+xoYvyXlWhJgaET3ah6ShLjSiCQ6Dm8Rd2pqv+QsRX1xsXbeATvvSqNmBX2uJvmwThpyN9xMhpt/Lo1rlktmWJFH0smJK9QVziUJr3A2UnNV3vfsYbG3yieWjvMz170Ox7bZWJmg6BXIuzk2Vzfg2i6rvQZFL08Yh1RzFZpBC8/2sLAI4pCPLp2kUKwycf1rOfDgu6B5QfoUZWUusyn9NlDcJIV2Vi+diGZSGvfDTFqHNDJktme+M1ZQdt2ssW5Axew7m5RnrVvWRINMmNsk2a2py5HyPibsbI7V8EfGujIaNpBWdRu1u7pe7uwcvOjF8PGfT9XbzHEFwOJnoXkOamdh/xuhs8rpsMti0GbXphtwHY9KuwanPpvqyxS3Q3mjAHx5XIjexhyUhkkGpoiTCE8lMH9EGp8VqpKFPX2zWErz96UEr+GKVrg0Wc5wMc/BcQ1cMkPVK6jYFnBxEuFd7Gcpmtbt8elzHZx4jJumrqPslxjMD/CCTYeE0AWaQYdGv8WFxhyrvQY3Tu3nC+cfJU4SHNtea//p2g6dsMvpF30v2/7u7tRVyWbWmpEbFAnK4hS0L0Awqy+G/rsJlRoQMZPc/N1EYgwZbCZjNrqTBY31vIv1Zd6zAJfNHl4PQOh9GF4nCy4mHK3WLWu4IB9xJ4zrVwTO3w83f7+EqdvLQsyaJmwBcPisuCef/AfYt4FNL3wrHz3xGQYLVYbyVdpensmx3fDEZ8R6efGPCQE8fwRIuLe1SMkvMl4aZWXhCBsqE4w6OXGLnvkQdBbgwJvkgNxCCrDnEBBZALYDT5IKl3+l4tLnwLgGLtlhGwJXSSKdsok/9/izs+9TJ7AOTvMbL38NvuOxfXiawfwAri2iUO0ooNZdIVYxcRJTcPPcf/YhlFIkJCRxgmM79KOAgpunHXSElxm/A+Y+I/tYz32YxmH5QYkyxQES38yMbM5IktnGl7Ncsm6JAaWvJNlg3J4syGQtGcOTZCMo2XMwwxiWBpAMyBiXbj34gTz1C6QRJ8gIWN0Pu14JH//D1KWzc+K2bCDVjZm6BW/lPN958A30o4BEJUzaLhz+C21dlVic3McYFlQ3UBua5oP3/Xc822Xr4EamKuMcrG4SAe72EizdD32lORxb97kuw+l/gEPTUDsnx/MAaWO8or5Ov/7cI3PhGueyNvzfmFLE9lrxsEqe/d9LOREFL8dgfoB6r0miEmaa81xoLXCufpF20Fm7kYM4QKGIVUwQh/TiPmEc0g67dKMecRLTDNo8+aLvg8nb04mdTUADXZypZPIEq5ceUBYIjDVgTPT1bk0WKMxkzgKQyTPJ9lw2bVGNHm4emfTmqWzWy3Y8XL9dO7NtAyRe5j2bN6OQiTlCWhVtZbabIHrCjg+v+0XJnylNaFJcr3Pgu+V4BqZAKQaikLHaGSYacyKnEIRwyzfCHe9gubvKrzz4V8wOT7PSXWWmtUgn6jFZGeO1W18Ii8eFII4jKF8HL/sVmNwnbUwGpmi89Meovfmf4bpvhue/DY4gZO6gvka/qaznKrDANctlbSg7EYMlsrHtBMtWxJ3cv7zi5Rr5Ei/fMsYtm26k6BUYLgyy0F6iHXSodVfJuzkGcmW6YRfLsujHAf04IFGKIArohD1yrohM9aOABEWSJIRJyOmX/iTb/u7e1MqA9Emen5CkuqABrbOX1hGVkcl4LnOcE4h8wfqoTtZayUaizDDAAulEN+n2hlw1JDKkEz+rD0Pm/1kLx7hOat32zL6MVWWavWXXN8BklOrskohj5wfg+rcI6B55t4Dqi74f7v1jeONvSFRu+axkPG84wO888JfsHtnGN73q92Xd0W0sLxxlprnI//3g/2S4UGXX0DRTlTFeueN2aRty9kE4+l7Rf9n77bDxIGeSkNbKOQ7MPsXAu38Ebvs++OSfwMMIAI8C73zuAkp2XAOX7HCSS/R1njWXCHBu3MVN43tY6dWZrm4g5+aodVZY7TU5X59h29BmKn5JpBjCHs1+myAKUSiCOKQVdAiSiJzrizKD17AAACAASURBVGUTBeQdH9d2CZMQxm4XBXljEVjIzRoHEqpunbhUNDtbDb2BFCy0HtUasbreEjLuThZYFJe2xiCzvfVu0fpEvvWcT4E0Z0WRdimISWuWjHtkiF2zjAk9G+2TLCCZ5cpb4ehfQy+E6nY4fCpd9p4/lmWWTsG+u/R5Ffjjx97LsdoZdgxP886lEwzmB/jCY++lG/XYP7aTn7r1e2D1IsuFAS42Zllo19js+jDzKJxdlu3sDqG1yNZcWVqd/Pl/l2N99E/k2EeRXJ4rJI54JcY1cNHDUvruNgTus/xwcMpdNlbGGcpXcW2X5e4q7bDD2dULOLZDlES6PWgMliUPc0uO23Vccq6EUJIkwZR0KCCKIyLbgde+A/6fO1IrwDzde+ekY6HRVTHhTRNNyUaCssRqNl9lPQ+SLRsw5Gk2wmRGloPJEsSQ5txkt5ndnjl+E5UyYelspMpUCGd5GeNe+aQRNKPmVkZkEMIQTgCDp1JLp0zqwh3+M9h8I2rLzTx44TEc2+bgxG7uOXEfpxoXma5MMlka4fnDB3hLYQhOf4Ezw9P83qf/K9VchVs3HWLzrpeB5cixnQXe/174wHvT8ohsiN5GgGUK+KH/GFYLXAMXnP9aUMpKcOMxIXKV9TVh34PFIo5t0w46nF49T62zwlxrCUcXMPaiPq2gg1IJURKhAM92sbAouNKg3Hc8Cl6OvJcTIhglAIQF7/lhKG0VpXtlpBwD6F1I0/qzcgmJ/r/hI4z7AZe6HcZysTPvWR7DfM6O9RGi9d8ZstnkephcFiMWDumky9ZC2ZnPxhqBlC8xKncWadTI6O120fVJNky/GhY+IvKbxqUaRBc4Atf/bzC2k1pnhYnyGLFKeGL+CMOFAb5x9x0kSvH68jh87n/AnT/PBxYOM3fiM2wemOKnX/x9Yvk8/g8w84CEwv3M8YyQhppN9fkwIgC1/638RxpXPbhYcR6sGCtx5OGp9VwAuIJN6b9kPPYojf7zaAZtlrvSt6jZl2o8x1K0gg6JEhs+SmKUUtiWRcGT1qAFN0fRL+A7HhVfwtGe7eHaLvvGdolGq5MToei4B25J3KEsoJj3Pql7Y7oEGJAw1yarswupm5MFFjLrZJPXspYPmfXNy1gsRkDcRIJyme2abWTfTb8luDQnRyEgkietujbkscmDMSC2eD/s/Db5PMalAJcDNn0jTD8P/BLd9iKrPbk/pqsb+IHdd4o63TMfgn2vgTf8Ln/21D/hOx4L7WVes/ulfODIP/P6vb9gMfqtcOjXvuyt8PUyrnpwscM8yo4kaQ70Da4gsfFuniZ86CxE0eWXXVg/CiWKXp6n5o8yXBikFXToRD2ZM44vBK1K1qQYEhLyTg7XchgsD+DaDnk3R87JUfDydMIu143vxnNc+MefE2DJVeQ90ZoucT91dcykzE5yU71s3IYvl0RnOJL1wJLlduzMd+vHeuvFWBMG3LJRHTPM/82ED0jriUyVMaQAYpbPksYjiBVyDAGbECGq6yHU/1/Y7IM3CtEK9LraxRqA7S9mbmCCpeXTjBWHmWnOk6iEF25+niTYOTl41X/hvqWTqNkn6UcBX7z4BIP5Ad53+B4a/Rav3/uVboKvr3HVgwuAFXtg6wJFyKTGJziHthPXAjhy+ModwOAI9t7NLHfrKBR2r0GkpAJa3KCYEgUacUjRy+M7Po7lUPaL5N0c143tpugVqOYruEkMrUWaw1upPPy3UmAX6TJkJyctK6IedIS7WTvfbLp9CeEYFGkFctYyMcP8P0eajOZmvs8m3mVrmFi3DXsAnCLYHvhDUJ4UxT2lpKd00pMNhQtpSNwQsUVS8KiOQL0m6fLoc8gNiRZKlr9xECAZRtwdo60LKbG7980SFl45J+HikW1QGCLe+wpyvRajxSEmchVetu2FXKjPUu81Gdx4PWdWzzO/fJpKrsxMY45e1CdWCTOtRdphl/d+6xf+w3Am/95x1YNL8PZZCyD3q1uVFTt6glkoEilgtBMoXtmuUu6rQNmnuO9izMunn0+93yJKIqIkJkwiil4ex7KxbRHtLngFSl6BG6cOMJgfIO/m8S1LZBY6KzD7DJX6RamM7tREcySJpFhOxUIkxgH0li51T4xlki1iNMLWikutAsO1mNCzqdNZ7x6Z7xy5ruQ2C4jEfQ12kymo2w4MTusOg7EUYDoua50MVAyLj4C9nGbvGn7CBqJlkS9AiaTE6/5A8lH+4g5Z1sgxGH6jgYTWIQUkgO2vk7qhwU3Sf3tyP1RGebi7yuOPvodYxewd3cnk2G7yfoFHZp/mWO00BTdHohQXm/PcOHUdBTfHk4vHcSybv3rjp64aUDHjqgeXS4YfSMGisiB0wEJqi8IQ8iXoXYHy0+owb7/xLnpRn62DG6l16yQqoRcFxEq4lUQp+nFI3vLJOT4HxsVScSwHz/HwjSTE+cdg5ayoznVXJH8iDqDfEPI27ouSfBKJ/EJ39kuf6Fl3xrhCxrUw1cmQ1g2ZBLus25Fd37yX9kJhPC2oBDlmLN3G1Jb8kOGtohHsFwUsByZZ69/TXYUJ3Ut56UGIW2Lx5DcJp1SahJf9hCxfO80jYRd39RwHR18MQ1vg6LvkGEukrVBtxHoZOSBgZ7uw9VbRvh3Zynx7ibg4yOfPP8KplfMopcg5Pl+8+DjLXSHdP3TiXj48cwLLb3JLZQcThWFWek2+ed9d/NnrP37VgYoZ18DFjNBDBa54Cm4kygsoAZeoDxun4OSJy7vPkXG8lwlXMpgfoB8FVPyiVpZLpEjRzYNSKBQWFvvHd4n8pe1Q8oui7aqUVOn2m9BegeasuD5JJEVzrSOplbFeCiFreZhkNAMQWa4iKykJacWuCf9miVa4NMJUug4qG6V4b831TOQYLVtKD7DESigNy99tR5rBTewRK+bcw/L9wJRUIucGhDtSCVQ3Sap8aZj7lk5S667y+h23cVMSS8XyHT8uleI3fosINUU9+NQvQG5KWqwc/Fa5fqaP88hW8PIcXjzOyeWzDBWqxEnMhvI4Jb/A6ZULHF06zRcuPs575r+A+gpaKq9/9/PVfeceVr/1yvdclQBzDVz0UIEnvaKVhRVr5lGB7SYkrQ6cP3vZ92m/usVbtr+EdtjFd1wKbh4FfOj0/bxh5x0stGtrZK4FlPwCtmVjWRZ518ezXe1tWKKmBqnQkdLI0TmWciqGq8gOEyXKAok+90vyWrLv6z+b5bPDAFR+GiobpHmb6bvkl+T44khybOJIRLI3HJRzUYmuEC6Dm6cfB+RGtkoNzvBWAQEvL5o1tgcbDwoo9Rq8RCmOlkb49IVHuXnjDZxfPkOtu8JA3GeiNEpxci8nV85y6C33iDU0MMnTSydRKM6tzkDQort8ijiJuX5iLyW/yItXLsLINlZGtnJq5SyfOvNFgiTEwvqKwALwgW9/8KoEFTOugYseKnTAtQRgEm3XJzZJs39FgAXArW2guCdHohKKXoFEKY4snWKiMEScJPTjkGJGxX/P6Pa1Sb0mfWzahGKJFeAXpDYmCWXyJfGl8gUm6LU+gS2rAme+izOfWff/9RbKV8pZKU9DZVJapuY0S+z4AgYqs9PKhOjVtmtrVshKr0kcdnEsh1xJ906uTgl3FAXSB3vvnTC2gwvNeTbZLvhF9uTKLHWWeeji47TDLq1+m1Ox1DC0gy6toM0Hj35SwNmWWHU37BPEIZurkyiluHPHbex433ez7+6/FO6ltUQ5V+LpheMkKuEv3vDJqxo4/jXjqgcX76euV5YFcc/DdhPsQl+7BDqhrqdJBtuRiXoZR7RSJlGKlW6Dklcg5/g8vHiUh+rzvGDqAFESEyuF0m6RMmSzUliWRWw4C8uSCWtZQtYqTYL0F9OoisliNWLcxmXJFvZlJQuyIGM+mw4A2ZR9sx0n813WAiJhrW81iKVRGZcJ6/rCrRgZTpWIRWI5hElCo9+iHweU/SJDxRFxXZQicn3c0rCc79guWLnApn4TCoO0vQK19hL9KBA50FyJnOMz01xgsbNMrbOC7/jYloVj22wemGSwUOXY0mm6UY/v3/pCIZL/4ptFt6W7Isc4uJEzK+d4cOZJ/vA1/3QNWP4V46oHF5QlOBJZJImDnTdpnvI9Fy7KcrkCdFv/Pxv6N458CWfDAp+eWeDW8f20gg5P1U/waPsMP7P/bppBG892CeOQxM0RJ7FEsHS+S87xUSoh0dq+dnkUFhzWWrgmCdh+Cg6mUDDbUzjLwWSbo5FZZz3QOHxpTkx2fTLv3rAk7WHJ8ZDI8Xl5EU4qVOkmEQU3p10j3Zy638S2q1iWdDdo9JqU/RIDhUEA3M4qy5tvZDhXphF2GbAsaakyspUgCelFPc43Zsm7Ps1mi+nqBhr9JjYWE6UREhRxErN/fDevnrgO2kt8g18Wq+hd3ySWkYdwRH6ZaGQrrkog6FwDln/DuOolF4x+i5NLcPKR1nGxUH2PuONDoC2XywksgH3DbpJcl2eaDfpxyJHlM/zD6RN837ZXMawnUTVXlomnrZUoiYniSH+WULV5YWti1HZ1L2hXyEq4VNYyIVXsz/Iq2WXgUmAxWbJGV8VorZjEuPV1Q2Y7+UlxibQkp4S/G2KBxFGaywLgeARJDPkK+CXmmgtcbMyx0K6BJe5MkCvTz5WgNEwn7HLv+Yd5ZvEYFx0Ptr6Avu1R7zWYbS4Q6+siAB1R8SVJcSBXpuwVuWFyH68e3Ayf/SPU1HVw5J+lbci2N8Ab/woOfS+rz/s2PtNe4LG5p8C51do18qZrwPJvGFe95WLlA6zYJjHhEUuhIodoKQTrypWg2lsuSgRIOcRJTKwS4vyqKPljkXdy5ApSjCjSCgmtoC0CUCDLq5jEuECab8AryGftQl3qopBGhLJykWYYEMlaKgaMTIsQBwnlrid/Tcg5O8aug+FtKcncXBUQVEpI3VyZXL4CKqEThzT6Tbphj1p3hfnWEqd16HeyMkar36bkF/Ecj6X2MufqM1TzFfLaqltoL+E7HjPNeUaKQwwVqqx267iOi0KxdWgzJ5bPoIDh4iB3bTgIpz5H8ppf5s8efjfW6Dacmcf57pu/k082ZmiP7+SDH/tNelGfv7wKc1Qux7jqwUXle1iJgxXbokQHqNgWM/7CzL+4/lc77FYFb+IsQWuUdtjFtRx+cPudTJXHsC2Lsl+gFwcyZ3UjtGNLZ3jR9DBhEhHGIY5lE8YRtm0Ll6HJSSxbT+LoUldlfY3P2kXIfM66RhZirZjiQAfJiDVuVdYtyq5vMnK9vER9yqOSo9KuSZJf2BNR6uEttIMOnuNxvj7DfHuJ47XTLLVX6MV9bKRgc75dA30dbt5wPbPNBRpBm5VunUquxHxLgOXkyjmG81WGClUBHZVgJwLKRU9RcPN0ox5T5XFajsexoc2ceuYjXGjMU3Bz7BndxvsXj7FjeJrHZp+h1qvznm/5/DVg+SrHVQ8uuCGKCMvRlXjKwvJi3PEc0fEr17MhaRbpjIIbuXz49EXecdtrsIDFzjJ5J4fnuGsN0aIkWmvtCmBbtrZcxHpRiUqLEi8hna3UqjCgYnoFfbkpk3WTsu6QIYON2JOpH1qvxZJN/y/t1ASzJYr3Vd0pfeZJsWLcHCpXJuiu0Am7dMMevbDHTGOBnOvjWlJI6tg2Zb9EJ+yhVMIDFx5f0wu2LZsgDulEPXKOTxCFXGwu4DkeraBDL+pTcHOonqIX9lFKsdpt8OT8UWrdFWaaC3TDHqPFIXpRj5HiMH//9Ef4wNFPUO+3eN+3PXANWP4d4xq4GHfIiyCxIZaeRcpWkC9A98oAjD3UwGpOEp4Yxp9u0Y8CupFMgG7Ux3UcfMcjTmIsy8azLSwsHpl5mldsfxH9KCDv5lBKEauEXhKRT0IhTEEmte19aS2QhwDF+qQ3uDTEbCqiTbKdaXKeLVQ0sgYGVEy6v1+Cwe0CeN26cC0DkzC+SziX8hhsvomV3iqtoEOj3yTv5Sj6RUaKg9R7TVzHlbqcJGG8NEytu0oYR7SCTiongSQ6Fr0Cvi3Ld8Me860lCl6eXiThZdVXlLwiiUoIk4iLzXmmKmNYwGC+wnK3zo7hLbz/yMdY6Czz7jfddw1ULsO4Bi6xA1aq9m9EolRkQ69zxXab1KpE5+pQ6IObcjs5N4dvuwzmB3SECGwsPNsl0TILRb9AlERYWGK5gIhFafIXS7t1Kv7S5DabL3WLvlySnDmkOLN81qUyoktGtxa0u1SEoRuluVccSAOw2WcEcFCScZsrE1lQ7zXpRRIbnyqP0wm6bByY5JG5Z9g1vEVkJWyblV4D3/ZJNKktHJWArrkOq0lEN+zTDNp0wj5512cwV6EVyG9Yt013MpFHWO01GC0Oc3rlPLXuKp2wx6HJfVxsLvDd73+5upbH8u8f18DFUpBogFmvCeDnL7/Ugi0TP7r/Cdi2HX8LHNhoiQCUUoyXh3EsUZ4zw7EdLMvCwcKypD9RzvFwbFu3FXFIVCJuk+3KRLYdsVy+ZP9c2nrVuDPrZQ2y1czGJTIWirFgjDJbfgKCeSjuhOIEFIZ0Sj/QXJAe1dUNYr1UxkEp3CRmrDTCufpFLCyeXjjG8eUzLLaXSZSiHXRZ6qxgWRb1XgvPcbH1+feiPpZl4TniFiVKUeuuYmFRzZWpdesoRJ7CWeuzHUluUJJweOkke0e20+i3Od+Yw7FsNlTGObt68ZrVchnHVR+KTvItcCOUE4sF4WjSwVJXRsNlQMLM2DbWWJV4dIH9Q1v5jcN/S8krgIIwCbF021aTQWpjr02WBy8+znBhCMdycPR3naArSWi5kgYJS4oVs25OVlQpn3mVEW2Tgcx3VYS8NVKLFpdKXmYLE4N5GLkVxg7IvpaeEvdHS3Li+FKf5XgSzfKlULHg5tkxvJXx8iglv0g/6hPEAWPFIRY7y/TigLl2jaXu6hqwLPcarPQarPaaLHVW6IQ9EpUwnB9gKD9AEIc4tk3JK9CL+oSZ6vIoibEsC9/2OLlynmO1M7TDLv04ZLlbZ+/o9sv/e1/F45rlYoaywIlF1yW2iVeTf3mdr2Y42joam2D8+mO8aGQ/9X4ba2ErhUM5LMsiiRNinWsvVomF6zgkicK2LRzLIVYxeTeHZdkkSUzRL8pkDjoaRAwakOa0mKQ5Y7lkeRcDQtnwdAmRflzh0oLG7CMpvwH6M9Celezg1nnR5V32RE6huknqgapTAi6WRT9JRH0h7OF0VymVhuiGXRbby7TCLqu9JkopFrsrTBRHcCybVijuTRAH4PgSUU8SCm6elV6DwVxFSpl0BwRxIUUTx9Vp/q4lJpoCglgyActeEd/1eOuN30IreI42Xf4POq56ywWQCFHkiXuUWChlpT2cL/foamtooMKe0mYG/BL/tPA4P/qiG9bkFkLNIziWg2s5uLaLjS1N5x2Rtbz3zAPESbIWNXFM/yHLkmJApcM8hnNxEAuljM4+Je0X5CNuS3VT6uoM7ZJ3o/BmLJb1CXOWDxMvhdIGqWfqndORpkhC0cUhSeoLe+D6dOKQfhzI8bYWYPUCJculkisTq4Tlbp1YxUQqZrwwhAWEcSTELOjWteBYom1T664Sq4SFzjK1Xh2wyLs5fMdbAxWFIlEx/TikE/YARc7xxBK0bfaMbGPzqc+zrzHPY3N/tZ6luja+ynHVWy5W4orV4kYQ27oBvS3SlldiZAoOy16Bg+O7+S/Vjbi2S6ISbEvIW8tKcd91XLDQURIR4rYsm3vPPsBdO24T2YXGnEzgoCPvAGFD9pe1RoobINZP6PJOIX/NqzMn/Y1BgxOS42JErE2YOdLbjIDuGVm3NAW95dQK8koi/FQZh4m99AYmiJIYV7ssTq8h3QY7dQg7rHTr2JYlMp7K0nVVidYCFjIbpfAdl/nOMq5W4rOAlb50Yvc191LrWpT8AkolNII2I/mqhLUtmzCRNiyO7VDyCvzWnlfAygXRjlGKQ5Nvvsa5XKZxDVyCPFaznEaJgLCVh8bKldmhKSNYrPH8yZvpRQFjpWFildAKOuRMvo02D2zbXgMcpRQ51yfn+sRJjGPZYuHYjlQId1fFNYr6IhSFkhPKjwK29IMe3inyB/WZNA/FcgQgRndC7ZTsvnX+0kiT4VeyWi+R/n/vlBC55Q3SobBbg/HrYXw3bDxIUKjS7reEhLULOEq7b6URmLqeluOx0F7CtkQf2Hc8mkGHUJc6eLZLkIRr598I2gx4JSwsbMtiMFfh+Op5xgqDOLZNkiS0213yro+FRa1XZ7w4zPTAFL2oz9n6LHGSYFkWh4tD7At1NWbtFOy8Mj/71TiugUurjNTuWKhAX46HH7ni+3UPTDHXWlp7Ql8/vpsojjjfmGVLdcNaiNm4PJZlaYtFJpStrZeHZ5/k9o03iqURdERmwdFI8OKfgaWTok7Xb0JpTNyf/IC8jDTmmvWiCwyXT6WWiyLVyDU1ST5fysEURwW0ioOy7Q0HiCoTLHVqLCweJ+fmKPtFyn5R6opcHwamWLEsHrnwKEeWTst5acAEqa1qBG2sJKYXBaBzWvJOjn88f4Lnjw5x3dA2APYObeH46nlG81U87fLU+y0c2ybn+Cx2Vqj4JaarUwwXBzlWO8Pzpvazz/YkilUYECW/a+OyjasaXHK/uFuhbFRsoUIXlVjELTsjlnLlRrztKDdN3ckT88foRD0a/SaVXBkVKM435thYGV/LyLUy/4qeCEYBa27B40snGKmMMTKxmziJKT9zD+y5S6yTsCuZu24eJvamGbOgP+vUfNuR8x6YgtoJKR1QpDILph7JuEcBKWPn/X/tvXesZVl23vfbe594wwv1XoWu6lTdPaGHMxwOSTOIQbRogyTGoimApk3RhglRoGVLsCwbsACTNmVAtiEbEiVDhi3BUTAdCNuySBigIJGmIIKkwZkhJ3DY3TPTqaor10s3nbCD/1j7nHtfdXWuru6qd1fj9gv33nPDq/Pdtb71rW8h/MqT3wHbj1NnA149vMzh1a+wsFW/pM0Pdzg12AaTURWGRVvx6uFrfOX6CySxK1YkGY1rSZShSJb1XJmK781+JbxUSOrIR2l8zG4+ceoih820FxZmJqV2LZkWFz/rZa/2x3afwnnHxe3HBOTahQwtAvzGvxr4E//zujS6B3FiwSX/+Y8uESQogtVC5Jr3n+M2P7nNf//9/waXjq6xVYzZUVsM0pLdcoth1+nQK5sI1LK73IGMieVSZlJ2B9skOomTwBYufrc41tcTuYBkLEoLwQrHpfnaxKzFidvbxnnZbbS4JrcdIVsAOt1LN3/ULaZPkC0Dg22OjOH64WVm7YKFrbg9l82Rj26co3UtN6a3GOdDLh1e5ZWDy1yd3KRxDUqJxcU4G3J9dqufBi+SrJ/8NsqwW26zXWwQbn2Vuau5OrvNICnYyIdkOmWQFBRJjlaKc6PThOD58o2vsVNucm60y/nxGRZtxY89+0OcV5p/cPn3OTvc4bs2HpFs7bDb/bqO9xonFlykNSsfUCpx6Gg7EmzxOlHrW0ZnkP02IvuRZ/jv/rmf4Lse+1bKpOB/+oP/o5903q+OKNOCLH4ad080hEAauRag18DkSc7O4BRFUpAa2b5YJAUcXpPn01bi8IYSxaxSoj8ZbMvvBqciGbsTHeusZC46gb0Vv+DV9nPWP61lixsEoOKe6lE2xHpHNthGK81kf4r1jsN6wlEz7bMIH7w8b6Vx8efK1pKZqa5pDD4EEm1wQWbXU5Pz5576QTwBo7R4EOdjXpvcYJiW0QjKkGgj5W58b2fNnI/sXESh+PL15/jN+R7DbMiT248JqTu9BZP3qUt4AuPkgguADtG/RQDGzQrC7F0K5za34XCFBH7iIrzy0utuZncv89jmeXKT8b995VdFBBeFcEaL/UJQIWYqKraaVX+ipSbBKN2vFBlnw8gr5GitSWe3ESI3iKAOEEsGI3xMPo4ZzJb8rvNa6cBl6/yKBy9LTcwOYpG5asfQbVwpiCbZY0qd9JseW28pk4LaNuxXhwC0Tro1qU6YNDNq1/Zq5Gk7Z9FWGCXg+k+ufIE/fuFb+b0bX8V6R2kyRukArSR7e3rrUc4Md/rJ8e1iTO1EgNituFWxhe+C59rsNr/+4m/z+OYjJDrhyuQGuck4qI745rMf5zOnn4GbX3t3f/91vC5OLrhEkyi8FosFFG6m4BuvB4S3jqgv6eLxJ+BKtGsYb5F9zy5+XuAWGcnBHq8dXeN3Ln2B2jaRN1E03oK3pNqg0GQmwXsv5vhGBhS7bYuJTki0LJ8vkoJBWqCUkoVorl2CRz4SPkUp4RRMKoBTbsZZn1gamTTaenoIBew+Bd/20/Dy78LkCixuwuwb8nruFNF1i951AtNb5Fvn8UF2LOmgaF1LmRbcnu9ze3FAZWt8dNCzcYLbh0DrWmbNIgIpvLD/KgNT8NXbL5LplGFSkOmUcTZgkBQoYN5WkqUoWQOzWYx7HYu8J7KtUvQxAnbf2L/E9ektntl5giLJefXwKqPqCOssnzn9Ebj0m+/i77+Ou8XJBRfdDSpCaBJck8D+3tJ57p1ECFBFc9rtXZjNYWsL0pTsOxSndq6xaQY8/0sVYeMCv/HS71IkObuDbVxw1G3DtJnTestGPmKYliQmlgFBRWARQOnasalJZBo4SdFaY7wXYCk26M2jgpdZpm6Y0VkphdIyDjkivy/G8YXEHrPJ6Fd+7F+SzQJXDVQvHLdZ6AYXi0elnb3/KmQlptxCoahtQ+0aWtcSCCxshXXCC4Voih0I1K7BeSkrXzx8DR+CeNlEwN5IB6QmITcZZSyBcpOSmhSthJCS3dmaMi0i4Mjrvjy5TqZTIXdtQ6NaGtfy+St/yCgrab3llcOrDNOS33QNP/BnbqzJ3HsUJxJc8l94OhB0lPsH0IFwbR+a9yCcGw4hz2B6BFvbUORQlvi9jL15yfd+ZofnZ7+DSs7xP7z8W/zQ6Y+TVuE0ZQAAIABJREFUmoRZK3qOKrrNOe/RKBKdkMYLQBsHGzttnfeB2tYcVkdsF5t4pUlNKuXNYAvqmZQ9JhVQSSNRmZby+1xa8ChFrU3s6ASKznQqG8DmOemkdObkzT4c3Vx66Xaq3WwrLlo7gmZGNtgWXU7ki0AxzoYUJmO/nWC9Ff8W16BQvfn4q5NrVLbBBicT0MFRmJwiSRkkJaOsZKfc6ueDXHDSto7A4oPHOkuZRmtQAt954dP86gv/L6eKDabtgkkzY5Dk7FcTJs0coxRbxQa3F0I8r+PexYkEF1QAm8jXAKFNYDqVy7uNyVEkURvJZJIU8hxfS4v1V750E4Dm8wekn77IPwwvMEgLcp2Sm5S5rXrJekdwpjrBBU/wof8Ut971pKdSUMcZmY18LEBkIrlcbtJnLKEr25RkMtkQryQjmrcLmmaG9z52WbRscOwym61Ho0O/A/994H/tuBWFQrIkb0W8V02hXZCaFOudSO+RkYZpu8BHiX/nxvLK4VVMLPc6viTB4JRHB0VmEoZpyWY+ZpSWMgoRnftVJGul0+SFu4kdtQAM05IrkxtsF2NsENGh8465lQ0PtWv4yU9+lsc3z/P1vZf57GDn3f/91/G6OHHgkv/HFwM6rBCTCjd1Ag5t86b3fdM4fRquXY27jRWkybKtHRTuIJKrt28SmrMke4/wD+zXGA9mfPf2s/jg2Sk2GWeRsFQKH8si0Gjo+QTi/x/beIRhLmbWla0ZZUN8CIyz4VLFW27hghhhB6R8aKw4v4UQaL1YFnRlhVaaLB9GjUsp4JTmAi5JJmXX4WW49o/l9agVc6o2bn1sFuSjEUWSsTffhxB47eg6Npo9BQJ/cPNrJNrIbqboYaM7MFSQEtjMhmQmJTcyjJialBA8RJe6jp/pWvBdh816h9GGxrXRnkK8YBrXopRm3i7ITMqPPftD/IuPfRtMb/GZC5+B5/4RPPru/wms43icOHCxmzdIjk7LD85gJyVcv/begAXg8EjUrcUAjo6gLGA4ABBx3iyiWQjossbujUm85si0LGxNHj/piySXZV1KEwjxRJFx5kW7IDMZs2bBs6c/wuPDXZje4CvzfWbNnGk2J4TAUXpEmYihVJkWNK7tT8CuVGmdfL/X+aAUGxgtj6lUSW0byayKDQEZ74Sv2XoU9k7D8IwMKnonpVe5LRlOswBvUd7KzBNS0k2aGUoJwatQXFrcYGhKNtIB3ayyQqFRzG2NUlCYnN1yi3E2jC3rTrtL3N0koGtDoLatkMiI302GWFc47/vsxsfSryuhfvDp74HrX4t6oKlwVuu4Z3GiwCX9z88EHcbQRvtHr/AzD/v3YI5oKq1W2ka2Ad68Dae2hWx0Gl55Ra7/6MehXOCvFbAd8KaSzII06jKWnSf5IJYVs7IAzaGUpfEt50ZnYO8VmNxgHloa11DZmkQnTJpp74qfmYzWt7ROTL210ixstaIracR0KnhmzZwLG2epbI31lspWlGnJMB+Smkelld11nTbPx+7SilFMt/jM1uBaEi2k8835Xu8Y17XUE2WEE1YyQ1C5Fh2vb7wodE8PttjKxyRahhS3y00OqonYYGqDVz4OI1oqV1OQ0wTXW4Wm2tB4i/WW1sv71g0wfmznSbaqqUxmT2/Lc+8Ehuu4J3GiwEXZDLUosfsjTNlIt2ixgLZ+6zu/3bAxA1pMoaoJSUlYNPI4z36C7GIt2hqlIbWMDWQ6JdViyG2U7k8yFdusOnIFOiga1xACfP7KF3l04xHGjzzL0Yu/TZakzFmI1kUnLMyid6lrne21JF2XZt4uoupV98N+w3zIuB6KvB45CU09YZCWnCq3GA53UcWGnITtIk5fd/JhhHsxqbw273qbyiaCWrtSugxMgem7OpJJoWRlSgiBpzcvsFNuoZXmsc1H2Blss5GPeHHvVSZxE2NuMqaxxGudI4SahZVuX2ZSbHxPXWTBG9eyU27ymXOf4Ke/5cfhhd8Q8ZxdwHf+vXWX6B7HiQGX/K88FRQZoU3w1qCdxk4zuHEP14ck2RJcAA6PCGWBKjPCp76J7MItfL5AN4VkNEnLlhlQmIzC5Ayi0rY7WRVEPsThCXgCeFnt+kc3v8ELt15Caxn0a2PJ001Jd3uQu2N0JZFkMJa5XdDYzvFOwCxLMvainywQLRCkY3WwkMysTEvOb5wlLzZI2ig49I5+nUk6gGhF2UTvmhBCT0L3TnFJ0c8c1a7FBofzPmZxsFNsoqP69vz4LKlJmNQzAtKKT0IiJU/oyG55X7TS1FZa25VqyKPdpomzSpv5mH/lUz8Kr35OMi+TwPeugeX9iBMDLrTxpQZQWhafceXavbVWyIvj4FJVmNJjNuaEwRyf1qACvpijsx2CadlMhuQmZZSW4nOiTO8k19kzNralSPN+kDFALJMCzokHTJdtaKUINvTyd+99PO0krLPM2gWta6kj4SnZS0Kij6htwzxer1bA5WocSRhnAxrXUCQF2+UmChjnsvPaB8/B7CbzdsG8XVDb5hi5arTBBkdrLZlOmNua2rdxMtxR+RYfPE+Pz/crVx8Zn+b0cIcbs1tcmVxHoRhnI7RSzNuKU+UmjWu4Nr2NUYrWWXzwzOyCEAIb2bBfxZLqhFPlJkU1kVa8d/Dd/9UaWN6nODng0mUDceguOA37B/f2MYoCZkfLn8+ewYznUFSg5RM5KE9IFyQbC1xas5PtUCQ5eZKSJWnfclbAUT2NbvYV42zAmeFObxgFy+WIMlHsesIzBIQzgX5CWCGkJoG4xqSi9Y7a1v2gZOcR053sHX2ambTPjMZR2bs72GbeLlAKNpqxgFsI7FeHHFYTatvQ+hajDJWtKJJc+B2lWbiaua2pfCO+6EDtW2rf4oLndLlNZRvG2ZBT5SYb+YhXDi4DAp55kjLOR5zVKWeGO3x972WO6lnsBskE9dxWLGzDMC0jMe4pk5xPn3tWND9JDhf//BpY3sc4MeBS/9UXVP7zHw3ogM6jWO5tDhu+7Shy6RZVc9g8RfZogLwlqEBQK4OIuiVsTHDphN1ykzLJJWNZCRscN2Z73Fzsc1hP2S23SHTCTtwj3Xlti4WjvI6uwySc8PK8Cf0CNR/LJBHrifOdxgcnJUdc3RHiChOUorENWmuscxitaawQtVmS9nacs2aBUZrGNTSuZdrMmDWLHqAWrSwta510kK7ObjN3FVl0mGuiQFCjqULbCwa71viVyXX2qyOKJKf1lto2DFPPH3vy29hNCq5MrvfWlmWSM23nTJo5Nxb7fWt/kBTkScqF8VlRE+/+S2tgeZ/jxIALANaACgQX54mObSe8B6GUyP6vzeGxC5DGST+1BDHlDQRNO77Kd40vMkoHlElObrJ+yTzApJ5xc7HPN45e40q9xx9Lv4lJM2er2ABCbLEen9/uujGSyUR7hkiYyu09NoS+VOnu3g0KztqFLGGLs04+iElTNwOUJzKnU6Q5G9UIn8ljdRshZad1YN4smDXz6JUrIsJuPcogyalszW6+Sa7TWMJUeAK1bwltoDAZSbw9wIt7r0IInB3vUiQ502bO7mCb3cUhTF/k3FikBRv5iP3qkOduvihZT77BVj6KM0iazXzMUT3lc7de4NvPf++9/duv43VxosBFOrsaP/cyQ2Tvsa5hPodtaWeasZH5JRXkgnxVNoWg+YHtZzhXnoom0QatNYGYVQTH5aPrPH/4Kl+a3gLdkkYzpdrWBOiXifWvrRfJeTGUin4vXUbUZTciQhNAwUtGJff0dHDVt65jC7d1kuk13pIow6lykzpqUcqkZGErjqpJBAMhc6fNDKMMXnt8JF1VtEK4MDpN623cz+SonWybXLiaoSnYKjbIdEKqDT4EdgbbXNx+jI/uPoWpJjJ0uX8JZntQbvJImrM7OMU4G/LywWX2F4fkScoz248zbWZMmhlGGz6y8yRGGz7zyL++zlruQ5wocFFarAjSHUt7O5OlZ+9mUPGNYu8WbG3CaBNlVksuJRlTBBhMzU6+0e+CBvquR+UaJs2ML9x6nq8czNAUuLwiiX4ts3bRq01ZyV1WwaVyDaN0QKINifaSUcXrO27ERzGdip0ihcJ7UfL6/uL7DCmEQKISatfQWNnPLGWW4vb8gBuzWzKpbVICopINKmC9WHV24jnrHWVSMIjjB3LsAU0kmD2ecTagTAoyIwOHnzz7Mc4F4OaLkTS7tRT17TzBOddQtRXTZkZuMp49/QyXDq+ilGLTjkREGDwXxmfXwHIf40SBi2QR4t2SbmvaCxfgpW/c28c4OIKnnxQCt1sPG+SxQ7IA5Xk2f4TcZNE+Ycm1GKWZNQu+cXCZL96eYRYb+HLKZ8rH4yBgbNtGXgQk31Asu0UheJpYXiXaMEwHRB43Ct5Uz4X4INmKXBV6HqZT8zZRH9MBUa61OL4Zcb2rabCVY39xQONs9HBZmj51PjXd8KVWKu56NqQm6Q24LaJM7iaaiyQHAruDbcq0YNFWXEtLTu08yf7igLPeQT3l9nALFofsFBt447H+iDzJ2CzG7A5OcVRPorVlQ21bloKcddyPOFngor34tzQp3mr06Q38W9m3vAOXOUwCsxkkCUp1fI6CELMT00JS8cTwyd5NznlHiFYKNjgmzYz/59bn0BMxng7akcXrxQNlybd0gCGZggjUOlJ31i4igIi/bJ5kccaInthFKVzkUzoy1wUXFa9CzlrveruDcTZknI0oklz2AQWxUciTfKVrBa1zMultawZp2QNhbsQqYiMbMsoG3F4c9oQyEey2ijHOO/IkY3e4TQhiyXB6uEOmZEzhWnVIKMZM5vtYb2UjQD0lTzIubv8ptbf49eCDCAgrW1O1FWXi+a7HfnaNLvcxTgy45P/hR+RfuNPYRUZoA3zxS299x3fSURqOYVBC2xJ8bORag3KakFXgE7576yLjTGaOOlf/aSsmSa2zfGXvRcJiG+oMVdQE09CGuJI0Suo7LmRZVgBIKSNCsqWLfmVrnHG9G1u3vL77FA8x4+m4nq48cd5RuYZESfaRJylFkpFosXZYZkuxvGKF14n/dWSwWHembJdjMpMyzEo28hE7g22M0gzSEqM1e4tDps2cQVrQONuv/2hcyyz63WQ65dzwNCjFRjNn1s65Nr1BCHA6Go+fKn9QHVa/GXYHp7g2vcFGMebju//yGljuc5wYcOkiBEW6OccelfhTu3DrPXimrmY13ST0eLScuHaaYA3twRDzsdugLN1kchLtFKq2Ya864mZ1wB/NL7GYnkIvNqSbFQnhyjVxNkZARSTyAKJh6ST1nbqVAEaLY13lGtlg6J0YTikd7Qeky+MiuBD1MI1rmbULWfDO0v3ORJABeqBzEVy64cA2LoUPEeg6IyylFN/0yDPiNwM9+J0d7aKid03rW7bKTSb1FKMMo2zItJmJfN87Lh2Jklqj2SzG1K7h6uQG1yY3+eTZj3J+fI4sHh9gs/gBBTDMfid06uJ13N84ceCisjZ2jOx7AxYQYIlkKSFAmgrAqM7hLhWvmNt76MUYvTXBxT1FPngWtuKlyVUO2xkv1DdJ5mdQ+9s0e0OSYYMKCuUTam+5PLvBxfF5lALrRT7XkbhdBrGamZhg5HvvcNqQaEeqHa2WFbGNb49lP91A46RZcNBMmNmKRBkGSSFlikl7ovewOurvmxixSqhdy8JW7C2OGGUDBkkucn7vmDZzxvmQ3OScG5+Og4cJw6xkkJa4uPID4FSxxcIKaX12tMtGPmbWznu9zCgfclRPWdiKM8MdLozPcXq4I858nQfNHXF29MPrrOUDiBMBLn1JpIIQrE6TnrG0Tz0DL379Le79VhGtIQGOJnDmNGYEvjViB/DSPvqxHVR7SOsVtRfJ/WY+4ot7r/Kl+RW0z0kXZ8Gm+FkOL72MfepJsqEBn7Dvpuz4MQfNBIXisJkyToccNJNegdtdxmnJKClxUVuTaINz4ldrtYBameZiQ+na2FIWYnnWVkzbOUftjKmtaELLBXUaozT7sdXcrVh9ZvtxXPB86cYLQgx7efzb1QH71VHcxZyQmgTvZBn8rzz/62zmIwZpyZ/+5h9l0He0pAs1qafShjYZk0aMu+btgtxk0VBKkZuM3cEpmeT20nYfpqXYJsz3Zbd1F+H3wp3ixHXcvzgR4NJHmxC8JrQGO0nB3gNbw+CXLe3FDIJHaUXwATtP4Pp1/NnTkFiUT6l9nBB2li/Pr5LtP0ZIW9TRGN+kuDlweACtRcjg6HHiavaqiQz4BcfM1lIi4ePidk8bLEduzulsk1FSxvJEvHdtXO6exDmf1lnxrg2eRVwM77zjRn3A9eaA627KGTPgViMlRbcG5NmdpyKfIjqbp7Yu8PvXn5fJZG9pg3SaEmv6Se+Frfm//ugfMW3mNK5lnDX8r1/+Vf797/lZmazWCcMkF6I4jh+MsiEomNaiURFBX812uYVRRtzyQCwgOi+WrR9dZij2twPOkuTfv85aPqA4GeDSGXFbg50WokGZzuDqtXtz/M5oKngx6t7OMIXDTzUMhihDlP+L8rV2DbVLSY7OE45GqEFFc0nD5BCyDDa3UIWBtAVtue0XjF0pZQk+8hkei+PIzamCYxEsCsiVYeFqzuXbbGcbuOAZRHK3Dbb3cNFKMbUVCpg0M/E9CY7DdsZ1N0X5hLlqyV3NUTuLs0VQObHxFNWu4g9vfV2sMr0Vnia2t6337DdTGt9yptjmtekNATJbsbA1latlT9B8X6wayg0GJoUAeVqIbSaBvNyC4Km8pUgKARaTyOratoqDouE4sIDsZ1qXQx9onBBwARTYWU5oHKFu4PJrb+7jUg6jq//byG6CF89c20KaolSQRWujFP/4oyTjTqi3nE/eryeoRUEzLUiDgpdfkk/fixfh8UdJT03w5ZxgGkLQ3LITtJpSqJQmOKpgWXiPQkt2EwoCgVo5rvgZdWgZJwN0tGNItawqcQJPgNhD1r4VwPMth3bGq/YA5VOUz5hRMw2HLHzNo/mudJKsEMQhwPN7L/edrsZZDlopZTSKy/UtmmB5sjjLzC6YW5HgV7ZmZiv+yx/5j+D6czA/EG8YFIx2o2nTKfk5yUQsF3crTZsZIH7BRW+JqWDjXzgOIge/El4HNuu473EywEVH97NBQ+sKKTkWsze/T9tAdneCkPEWTO6YqE5zKMpI6FrQAVM2mFJIZBleFHuESTvHBi/u+a2l3dNQxeczKEk3W8JgTkgrQuROJt6iMByyQAWx3dchkawMLTNLKoC2gOO2r5naBZupGF93regO3jquZmFrZq5i6hbccFNUMHLxBkIJoeVWqDnlagqT8eWbX+s7VFcWt3rGqfWWhWvYd1P2fI3yYoA+bA7ZSoQIMUpjlOZ//+xfZ3zrRZjcgMOr4gZXHcGFTwvQjM7A6LSYjA+2QGmKJGOmDIOspFBGSlrXwsZnXw8ia2D5UMTJAJckusIHRRogFAY73pQy5I3ijUYD8hKSu7xtiYHRSL6PbWJ0QKWtfHVCLLrgObICLn7cQhhBuzLj1FpU6ghBi/guxOHDoFFBypvuexH+xr1EQaG6FbA6QNBcrfcYJSXet2DpS5YOWGrXMHcCLvtuTuM0yuWokKBcItqVRF7LdXsQ7RKa+DqE55m6BZftIcqnBOVQPkOHQXwTApe9TEeLuA/+/p/8Gzy9OBJgmdyE2W0x/LYVXPocJIWsRQHJYlwrnEqjZCLcOwiVWGk2cxgv3zqu/VLg3E+tgeVDEicCXOpfeEllf/XxoFSCKqXFy4Xz8NybgIt3dx9s9F4GFO8MpQV0rMMuMtLMopQADCqAk4zgqj0kdzMMCpdb0u1ztAcrGZJzMuDoFcqmKG3BaaDLJqLfi09Ax+0AAUBJluORKezguOImjKt9xklJabqsRYClcg2Nt3y1ui4AFUwsh1KUNyiX9GSyTz1HrmKoZpQ6F3+U4LhkD8iVQdsBKsieItWBYjeoSeDv/uDPkeqU1CR8ewgwvSlcSz2J2wImUt5MXoPReQGc0RlYHAofA/LVNnEdbfz7dLYJz/21QLkJT/y5NbB8iOJEgAuAquMJHOd9zKkMt7F9dye6wfiNx1DejKdpWqgbkeqXKTqNvjFeFrCptqQJUGvpBCmfw6lDjD1Fb/5gDMEqlE1QJkPHckqBpBERSFRQ4FcsF5TwL0GFmNkYCJ7nmxs85jc4nW2S6ZSb9QE37YR5sNgA2pUx6xHgUr7LmLQAIgqlcoJu2fNzBqFlz9cROzLqoNFeJr110DFh0f1z+sXv/0k+u3FegKGZCXDM92S/kWvEvzbJAQ3JQLY9aiPXzQ9koZs2YvDkIw+mlBxnC/i9fzcwPvs2/gWs437HiQGXjtTFadwiw824e3kDYEzvwP+2QwF1JZyLbXHzbdSGiycrYEC3BSEYgmniiZyCceiiEXApR5Cm+MpjEoeKK2eVidDjl3uQAMmMQIYiTViWRkH3ZZUKikv2iACMTMGLzSHK5UCKjuWVHFMvM5igwcelY17HgU/HaTPmkj1AhQS8AeXRLkOA0izLuPj8gtL8hSe+M5Y1sbvTLmT9SLuIHSEgHQmomFTI22JDSHJbC8gkuZRNthbA6bKfbsfQx//yOmP5EMbJARe68kFa0sxnYN9gfeudZO1bHlqDdZDnkr20LaFqYaDlU5cgM0Yqi9lAISVN5GFClyZtbqBzGVEITqOcGGx3YNJPWa+8JJQnaI/yMWvx8fGURwXZd6gCXLZH0E4EGIKSr/2xlxlLByz9Y2mHT2c8lo45l59i4WuUUtxu5NhB+b4UWmY98hx/6hMfjWDgBEw6nkR8RuXvkeTyNRtJx80IWOHa46tk24UcS6m43TG2//+Zv7EGlg9pnBxw8XEFhvYkWzPCyNBuPgWf+/x7P7ZCuj2jKA/1HhY1vi3RxoNLwMSMoDWEJkFlsupUZVbArrufCuI74+NJrgN9zdSd8L6bO0J2NcOyUxRUBIjYSdJWphR6GqQjhZeA1ZPEK6CignjQ+KxCuZxX2yMKnfFIdoo2WG61+2hXyP2s8DQdKKl4jP/xEz8Ck+uxlaziwrRWXicIUOQbkrEkcZe1TiRbaeeSoXSWFKrrS8UW9Hf+rTWofMjjxIBLsAZlOnJVodKWdLyg7fQp7yV8PHvnC5mKDkBd45oxKnECHq2BKsW9fFtuf2oLvZFjFIQ2nkDzuYwNpI5gNcrHE2oVVPyqRwxAQKlACLE0IY48dSchGnS7HO7uukuwzFKgB4TVi88r6Rp5A8rxQn2L7TgRvZkopnXel14dGPbHQYlIzrVy6dCte146gURJBtNfp49zXbYScFH6+IV77H28jvclTgy4+EWOSi0q8bR7GdnZuQwxnnsELr/63h8geAEN7wWsZh7qBp8kBK8ICwt7B3DlMow2ZUl9XqDTBLeI6Ucq60+18aIiXuFUXv94kcuJvIz8Tkk55I2UR911XqO0IaiumxNv35UxKmZKRFDoO1JGrvYKpXN00BywICh/7B9O6O+5khGpQI9otpJ9RiaNXZ8KKOReKpZAwceMxS6nzbWW7MW3crsOXL73766zlgcgTgy4uKmHLEcZj+46vzqQfSqjuXyPHsQ7cF5WjFgH0zk+2ZDfPf+1pW5meijWDE2DnedQxeVip3dQOqBSJ8CXWnoP3lim9ETznYDTA0xsWXsVS6cEpT1BO5R2x2+rBDj6TOMOTkfVOeS1lDou2hk4QNc45TG9hE5F7idIS7ojoEMEmHQA+VD0KwopgZIigo2JZaQsrMc1AiqmoFtKj23l94Q37uKt40MXJwZcAJjN0ds53upYJiEn0+mzcPM92i+AgIiPn7iulhWuZSE/N3e0sLWGRQV1DZMpjDfR2wN03qBSC6klrIj/hH+I1g6rYNNlJrAEnI478Vo6SiqgVCKEr3HxfqwMdB8vh/rSRkWAaVMYTgmmRQWNme0SzAoZrjzBeOlSJW08vobNC/I+BCT7SDLJXHRCFOTErKWRJfbeiZufTuVnEMK3OpLNDbbmnq+DWcf7FicHXL78JTj/KG44gPmCMDCoxNHsjWDvxXvzGG0lmx1tKuChFJiOJ4gnfl5K+RMCXLsOpTjXsSmm3ipxkLUytIjcLXT9pOjFu2xJA6iVkmb5MPK9kgwmmoUr7cFJuRTuLLkisevTGlWVy2O6eAwU5vAUIReQVCoQtOszq6C8iPeieO72v/UrsodZaQGHthJg6DxXmvkSTNqVJ52PJZsZ7gof450cswrSIfq+/3aduzwgcSLARX13HshLOaFnc1AKXyfookYbj3dvQOjunoFbN97+A4Ug7e26kQxmMBDNTACGG5Leb26u3LYBVUqJNB7KeaklQxF+REU8ucv51JcysVS62wd6YJmRqO4+RA5G5Pl0XyOB24NXd3HCyajJCFKLcobQcTlBg3KyRVJbIY6V49/56GfZbGsY7izLOJ3Qm2t1HivVkXyflqJbyYYCRvkINs+JgM7WcHhFfv/9a2B5kOJEgAvnHoE8E9HcoMTkUaCGQuctvnOTuyPUUxcI7wRcQAYekzSWMbIbCO+lTT2bi1sdyPWjsQBQnkFZokzkWLzq28KRWj1O2sIKcHRq2GN1zrJs6mL1fgp5XnpJuvYZideSNc1LEdC52A4PoJQMRwpOGdm53b9ZnqAcf+GZH+bJrQuiqAUpe7JBJGmjtqUjb/OhtKcJS2VuNoCtCwJGJpOf2wo+9VfWwPKAxYkAl+TiGKUDdp6RbszBeEKdLtu62sin5OviXfx7jrt/SKLdpXNy0VqGG52TbAYkYykKyDNUEpYAsZo5xHUod7aJ8ZrQlT2rT7XjU4jnc3dVl+n0z1PFTML0INTNE+E1wQkpHJwWIrh7Ht2clAoE37W7XX/8V46u8jPf+uOShQQfS59Mvu8uzYKlPaiT9z7J5TqTCjC5VpS6Sb5evfqAhn7rmzz4obcmqEElvirDeRwEhOA1dpLcNWsBCC9feXcPGALoCCxjFU73AAAS6UlEQVRN06t2QUnZ5GINk2UyLhA9YACCU/L8rFkBmTuApV9HK/L8PsPwCuxSYRu86u8b4YNj5G13PyeDisrKhaqIjyEAo7SPZRQCLpG8Xcr94ywTgV+5+QV++9Uv9PuQRKfCso3ckbttLarbthYAWm01e7/UxiTZu/sbrOMDj2Pgkvzi5t3Psgc48p//aOhPguECmozmZk57xeLnOVy9/sY7o29cg2c+8s4f1Fk5QdpWOkLzuQCMc9Kidpbe1LvjTVgFDiOq3v7kX3Z/8EpEeV12Ea8Pd1x6YApKMpxIrSwvKxlS9xhegMZX6fI4Pd8TgcrrSOQKeau8PjZyAPDn/7+/zS9/5VeFL/Hujvc3yPvQjQP49jhxC8LFdG3s4p9dZy0PaBwDF/uXDh+qP2T6l745hDqFRSdsCfhpKWK2F57HTSzcvvXmx/iWyTt/YBezk6aVJWlHk6jVcFFkF0un1komYx3eaWmROwPWELqLM9LhiSAgv9MEqwmt3EfuFy8rWUtYVfMCq1mLYJvqL68rubrMKCh8lbEkfxFSt9OyxFIraMfStiHjp37jv1jOEtk6DiG2kYBeeZ8CSxWvbURgd3hVbrM2fXqg46HmXOyBgiRHpZCemi5Pmo0RjDZk0PAtQtkMtk7Bwd7bf2AXQUMrqCpJ/bNCSoTgJQNwXm7TtD3/4L0YiGvjUEaJyZRCNCQO8ArfmpVyqONbupJF9a3hjgoWFFExQYpk7J3dpzvKrlVg6nyw6//sOZX/wtOBoMAmMqntDKGoQPmlyK6zbwAu2wWPLo6EvD1W5nRdKxenneMQIgGq+PsLf2YNLA94PNScix4YWIgJdbdgzC0M+ID+pqelbCnKNz6ASWSd6nfuvvMHd61kJp3/S1cK2Zi9OCuP37YipGsaaBpC5XBNgm8TQpvg46Cjr+OlNbg2wTstzRcbCC14qySD8Uqymgg8d2YlyxKpK3OWgHTXMQOg/cUvCSecNZJFATSZAEmdgzcol6JchnY5ymYom3Px7/yMtJGProv/SnUo9gv1RECly1gWB6LQXRzC4gi+42+ugeUhiIc2c1E//amghyk4j59bwrxAlTUqAbWZ4RsF33gJZm9S9gxGKGfwi3dBKt65+NzZOCsTxXVd1zgEcOlyLskIf+G9AW2iwlYOEVx3e3HGx3cdHNVfQtBgVOQ/tIjy+g7SHW3s7r0aLlayn4AZLaQcA+mqxWh+7pLKf+6jkXlecjYqbjpUTbH0ctEBtBNjqFXCfBCtKuuZcCv1RDx0XSOv4U/9kzWwPCTx0IILgL9xBIMCnKO91IgadrpPcF74j/n0zQ/w5GP4/Dr2c++gJFqNPt2P4Vy0UNCxResicRvJXechi0CTZWD08UaWi1xNB0QAKCm/lJJWdwgElSBJqfi89FabK6DSZTBqtUOtQj/sqLxoWPSgIvvLnwjNX/vqW570alH2rXGlPWQWjq4B3biBWvIwk2sCKNOrkYeaiRPdOh6aeHjB5eBoOaFcRFPtEARgTNSbLCo4uH33VnQ5JD1t5SRsmtdf/26i64h0PiWdorcjOTvBnYnVaqeH6YGEpW6my1pMHFJUcYI4KmJDksTlbFL69CASjpdKogQmCuVW8EOH5byS02T/3idD89e/our/5IU3BJn8P/hY8FVG8AqlA+2f/Vn46q+xtFUA9l+RDtHsKmw+RbefiODgT395nbU8RPHwgkuWCZehtJysq5aWxsin/XzGMVVrF6MNeOJx1GgiJ1xevD4LebfhY8s2eIjEJ85Ao6J+JFpI6mapEQmxBCJmNy52ajqRntJRVxNfZwMoTdBKlPZeSTUWD9VrYKLwLcxKKY3gOMDEsYBgRWiX/NvfEoJXuL/9+3cFgfo/fV6lf/HTAa8JeHjuHwt4eLsEl3YmP/9rX18DyUMeKryBgOxBDvXjT4VeZq4NFHkc44/ZgjGwtw+XL8kd7py0/djHyZ4IhOEMZROaX7v65sbc7/wZLksRrUWFaoyUNSZesmzpZ9KpWb2P5VMnOlNym+44nQo4SWQaO00xmRWTLO37wxBU1LDQq27NZlzn0YFObGn7KsNb3RtSde1pAvj/5vNrgFjHG8bD2S26flOyFh0/0b2Xzo1zoogNHm7fjjd+PbiaUzkUlXRCnLnHwLLymF1G4u2yfd0J7Vq7VPc2jXSVbHcbvyyPwko207W2u/t7f0fb+c7XulIezYuop4kCvdiOdnVCcDp2pzQ+jh0ED+pnvuXh+2Raxz2Lh64sUt+VizikaWVYUGs58YpCTtCykD3Ri7h7qMvcVoYXdW6hyWS39NGbtKrfS/QZY9f9ib9T0erAtksStMcHtZJlxc+Fjo/xCG+hYxnoIsfk1YpurdOXqP7hujLILVK0V+Il04POkvjtlcTBH8+i1rGON4iHL3OxnWALmUDO0ugOl4NS0trd63YVrZ4c6thXPyuwhwPCN157f59v3/2JnEo/3NdGpe9SxYu1d3SKOi5m5fv+WHLsY+StP17W9ErdOJ/k5hmhTQlNQmhEX4MLxzOlLkNq5aJ+6tk1wqzjrvFQZS7q26NYI4sdIROzAKUkWylL+eA/jKtD7vzkzQpoG9ppgckt4Q9fkO7S/YjggGj5GJCvSbc/qW/zrHAskTDpJqk7zgXu6DLJuEDvWgfHVbgQyV05fj/IGaDv4vTdrAhcfcfKE355Tcyu4+7xcGUuqWQnDIfik9LpR+JJasoA+4dQDiMZao7f/8J5OTFvH8jJ1dTiwXI/ois5fNdJit4nWgu5293G+77dfExAp7VkaWUhX2M7OzgILqp2nSY4JXNMLnInDslMumPbleHKLks69vvI57QtVPeai1rHwxQPF7gAFEMpgUIQRzgFanOE2c7Rgxpu74l4ruNY1MpbcGsPPvIRso8kuEsHqG/9JFT3EVw6YPGOvqPkg4Ccj7/vpqm7LlIPNEpMqrJs6SUTAOcILuDbCCzWxPIoQBMJ5OCXwNGRxj2g2OPlWHe7url3+p91PJTxUJVF5IVkKWmnehWlqylbdB6tLKsFyy1hLAnSLmu4fgMubqIvbONvzYAgPq93NZO6x9GJ7JQW0yTnogCNKLbz8lxAfudiyZTEkYLuNXTAQvS0jYK7ZRG40truyq0VngYAu5IhwfEsxkdOaD5HfbsK4XN38+Fcx0mPhwZc1D+/G8gzGI9lMVlc1aoS2WpIamlubC45jWOnQ8wAqjmc2o7zMl58WIA39Hu519Gd8CYaelu3BD8fxXIugkzopovD8fKoO84xgR3LFR7d9ce+Qu9v24XvMiNWsiXi0KWLw5YivFsDzDruFg8NuDAei6ZlZ3vpmxItBpQKhCqDy3dxluvKo9Pn4OY1ODwkVBdkC2JvR7kJR+9wf/S7jRBi+cNxA6UQAL8Egr5JFA3AA0sQ6DZAroKOcsczlC5r6QV9K+VV93jd/bsuFCzLpHpxfCyh/a3AP/2v4U/80hpk1gE8TOACMBqRDBpclRIqoGnxbih6lVkOR0fLyeS+UxQ//a2FM49IJylx6KLFb28CjwsJfD8j7ox+XTdrNeMIdgmMq5qZEEQId0zB2wHKCkisAkO3BkURQUkdJ467sYMQhMht2tetwL3ZzDm99eg9fBPW8aDHQwEu6ic/FkgT6ZKoBpO32DYH51A6CNjcPDh+woQgBGh3kuzdhG/7NtjeRJULVGYwboirSrh06f6/qLcSqIXoMxs8hHSldbzSRu7ApR8b8MsSr+NlFMsMTccVsD3ZvfJedVqXppGs5Y545fAyp7cfvycvfR0PRzwU4MLGWD5RlYjFVOZIRopQDtB5Tbufws1bSxd+7+WkGgxhciAb/py0VrOPLnrRmc4s7oXnl1PUH7boyNUQ66JOE9PvVo63c3HNbMeXECIHE98PHUvAxC+zmNXo7mtjC/rO7ZHAKB3eFXTWcXLj4QCXEGRIL7d0+3p0WcuUsdMQojDMWbGLBEn/R0OYHMKpU3B4CM89B088QW8VmbeinTEJuNkH+hLfNJx9PSB0PEo36Ng54fWG2WrZfVJKvmb5inCPZcbSf2/fsGv2+NaF6N2yjnVIPBzgMl+QPiLSdlenJEWAvCGkLWo2jEN/zbIMCl66tKUs7ko/tUN7/TR89Q/x0xKdt4TRjPYrQ7j4pOxyvvohBheQ19VdkmioDfG1hqU5dh/x+w4sOue8kC6v76mc8JbgMnjty/D0X1yTuevo44EFF/XDZ4N+5rwMGV6/QTh9Hl3WmMRDWRHSbvAvniFKy4mmtQCLMaLi3TmNyhuyC5rmazKsyLCCxJI97SGvaV7cgavv84zRvQrngOhj04ny4K05HJDWso0+MvoOfWVvr3mXyEt48t9cA8s6jsUDqdBVP/FMYHcH3xrsnoK6xi4yMB6VN71tgKoLaUFPZ1H3YZcnjdLQtqgnzwsAZS3m2z+OGVZydVXQfB2aF7fInjz64F7sO46OfLVLPubtTi93JZRt4tKyZllGORtb5P7197uzJFvHOnhAwSX88tcVRS4n0dXr0sG4uSdAoj2ru5ZDJ/xa1YCAnBC3bssA30zKI7NzCGn8dFaQPFKQbAaaKzsfzAt919EBytsElbvdtxs3cHFEYHX0oAulIc0IvzVbo8s6XhcPJLgA6AJ0HiQrcRZu36Y9LKBNjk0Aq8TBcLDsjKwqWeMwnusc7rWHrImlVCA8egl19rYM6j158YN5oR90rM40rUZHApsHtrJex/scDyy4mEGNGTTCm2gtbdKjKaHO6NeUWkN7UPaubMt9xLGLMh5hBkH2IEcw8llss9oEUPhsQfbUHmxtfXAv9sMYWuw011nLOt4oHtyPHRVB4expKYvGIzAa3xq0TkEZ/LyAP/jiUspuMuma+CCAVOTozC0d2ZxBheh94jW6LbDlHn7zCF6tPuhX/OEKkxyfKF/HOu6IB/Zfh6ywiDL+s2dg9xRYS3DiqObnOe72YpnOawM7O5AKv4K10VDKyZCi9oBCzQcQO01qNkS7nFMmRX/2XeyMfpBDm5WLPm5GBWIU9U8n66xlHW8YD27mAtijAtwUDg5lknhjjNI+AsxdyMy9ffE7aaM/yqJCmZTQJlJGxaVgITrlAyiXcttP4P8c3t8X90HHxz4WvwnLmSTn4cWvy29/t14DyzreNB7YzMX+rS+qEJSYP4UAhxOYzft9yWglRO5jj8kdOmLykbPLdqpS0r4ua45tG3QJZK1sRFSg2yHZ953/4F7s/Y6LTy/Xk3SrTkrxygmfC2ptr7COtxMPLLgA8LUXo+dJ6N3nQh3QiZNuUgrMF8v9PwBHExF9AUynwrX0+5SVZDCI/D+krehlXIbdvfpBvcr7Gxcek1mtblZpZQgy/C/PrUFlHW87HuiyiGHcLZwksL0pMn/vUakX87ZJKgACEWC8gEvXPp1MaG6cJzs7kTmkLrr9ytoLwRtAWY1nAp/8lGRFX/rSfX2p9y22t+L2Rgh/53NrMFnHu44HG1zO7EqbOTGoRBOUbClUyUIGFusG8hyI6lzbHrccqCt46RXC5iOooukHFrGJEL2kBO0le/EGHnsC86nXCMkCf3hx6ZUSgJdf/ADfiPcW6zJnHe9HPNDgkm5bMYKqUkJrxYOlaEB5XJ2Br2E0Ev7kYE/u5GyvLKWp4egQVz2OMV52GnUlkpMRApKmb08n3zPt9/6o77/CY3nJoV8wDy3t/70L+7c+sPfiToDo16y8xe3WsY73Kx5ocFFFgwqKNLX4JkEnDSprcbNCzLUPJwIgnas9yFdtRBR34zoQ8IuAzgzogNJenOoSJ+SuTYV/MXJ/nzSokKB8yqV2CgEKo7F/8oDw9z7A9+IOH9s1iKzjg44HGlwA0B7fJLgjcFkOIYPDI1hUUGQwLKVsqWt6O8u2EZ4GYGsHbu0RBqfQoyqK7WLpFIBGdgapTBG0x7hUxHZewwCCtizUAv7+GLjPdpjrWMeHOFR4CPb9qj/7rQHbSrmzfyDAUlWyYqSOQKI1zGdLw6TTZ2CxgN0dQGHODjEbcyFyV7cTWiOAo11P8hKHIv3GIT5dEHRNev0izT984YN8G9bZyjo+VPFgt6IB9WOPB52DKjPpDB0dSVdoUIo5lPeyBK2u43qOOLjYWllOvzEW791uvqijKqwRUhj60QDaRH7vpYPU71v+wsdorhQf1FuwjnV8KOOBLovUTzwTSFN0ZglW4wKAErvGNBEAGY/g9k2o53JdkkoWkyYwGKAL8BWyT7mLNhHTqCiqWwUd1bWslah3efUCfOUP7vdLX8c6PvTxYGcukynkuXAulYaNkWQiCil55vPIrUTj6m6bYTkQ/9yNEXgFplvWLof1i1yyE2cITSLZib0Dh4MiXN/F/+4aWNaxjrvFgw0uAFrhb03glUtxOHFbzLYnE8Jv7Clu3Yw3DITf8wIfZQGpZDB+UqMGGTqzvc7FW41vDK5KsYsMVdb4OhHD7hVao/2bX/zQSOE/LM9jHevo4v8HjeQ7pOVQtZEAAAAASUVORK5CYII=",[[0,-81.333333333],[-40,-34.833333333]],0.5,null,null,"Suitability"]},{"method":"addLegend","args":[{"colors":["#003300 , #003300 0%, #046F2E 20%, #80BF5D 40%, #FFC263 60%, #FF7100 80%, #FF3300 100%, #FF3300 "],"labels":["0.0","0.2","0.4","0.6","0.8","1.0"],"na_color":null,"na_label":"NA","opacity":0.5,"position":"topright","type":"numeric","title":"Suitability","extra":{"p_1":0,"p_n":1},"layerId":null,"className":"info legend","group":"Suitability"}]},{"method":"addLayersControl","args":[[],"Suitability",{"collapsed":false,"autoZIndex":true,"position":"topright"}]},{"method":"addCircleMarkers","args":[[-22.941217,-23.458144,-22.866301,-23.73215,-23.280158,-23.152174,-26.754782,-22.966663,-7.923776,-7.158342,-28.699573,-15.911,-15.803887,-15.891832,-22.578207,-22.455412,-7.923776,-21.740146,-28.059503,-4.529956,-29.422776,-15.800343,-7.923776,-23.487815,-15.650645,-23.7328,-26.816027,-15.738433,-15.736392,-15.936301,-12.987695,-15.766282,-25.473067,-22.953575,-15.844967,-22.999322,-15.670733,-22.784671,-15.733383,-15.76857,-15.736069,-15.768679,-20.523639,-23.603072,-15.736163,-22.436066,-25.452969,-15.735802,-15.671995,-15.670943,-15.779194,-23.450999,-15.67071,-15.826055,-15.767492,-25.914888,-15.735975,-15.809535,-15.825873,-29.892234,-27.38501,-15.779703,-19.751133,-8.170197,-7.907342,-23.296286,-25.417142,-29.892234,-15.735994,-25.417278,-25.512815,-20.386121,-20.289922,-25.42929,-25.417142,-22.10611,-15.779959,-15.779889,-20.759349,-15.777872,-23.610788,-23.610912,-23.610883,-23.010727,-29.892234,-28.23353,-22.322379,-15.819504,-22.952637,-28.697932,-19.899328,-15.769476,-28.060875,-23.578312,-15.779923,-15.779914,-16.016756,-16.016786,-15.765169,-15.813022,-19.75804,-15.777512,-18.790343,-29.892234,-15.764933,-15.765154,-27.192394,-29.687606,-15.940411,-15.79266,-15.7745,-23.489907,-15.77539,-23.153092,-7.923776,-7.923776,-7.923776,-16.010116,-7.923776,-15.735991,-20.499163,-15.779928,-29.892234,-15.766566,-15.761023,-15.766401,-15.760423,-15.835391,-15.737653,-22.96181,-15.765644,-22.518772,-15.859874,-7.923776,-23.610805,-29.892234,-15.634099,-15.729444,-21.735725,-23.609205,-21.735725,-23.611058,-15.795229,-22.253752,-15.540833,-22.253752,-23.610872,-15.996703,-22.453535,-22.996637,-15.735153,-15.733045,-22.993028,-15.799469,-22.253752,-15.834245,-20.386121,-16.71086,-4.8636,-22.975785,-25.432216,-29.895604,-29.895556,-17.734448,-19.899376,-27.919333,-7.923776,-15.834811,-15.729722,-15.799363,-15.729444,-15.806445,-18.758851,-22.253752,-12.60907,-12.771599,-27.914263,-27.392442,-22.822974,-15.755453,-23.405238,-23.428509,-15.948196,-22.836022,-22.835912,-12.951203,-12.95132,-13.002067,-22.034521,-20.403469,-15.825178,-14.134764,-21.778143,-15.797834,-15.771241,-20.530789,-16.700663,-23.360532,-16.84304,-15.934356,-15.861243,-15.76792,-28.059747,-23.704449,-28.059444,-15.893617,-28.059723,-23.55653,-15.769146,-22.236114,-15.640663,-15.640872,-15.745,-26.93162,-22.197411,-23.962313,-7.440104,-27.685113,-19.165945,-27.919147,-23.406505,-23.472799,-15.775585,-22.827472,-15.767599,-15.735862,-15.729722,-15.671121,-15.73577,-15.731145,-22.905617,-15.76738,-15.729445,-15.575667,-15.814644,-16.627077,-16.015952,-23.406283,-12.94693,-22.969941,-15.76769,-27.681991,-3.789188,-17.74511,-3.742537,-3.742537,-15.843318,-25.371704,-15.729922,-25.426143,-23.501475,-3.742537,-15.729922,-15.729922,-6.358796,-22.957163,-26.962952,-15.744788,-22.936803,-23.442542,-26.100315,-12.947233,-25.412521,-27.685011,-19.156859,-22.903135,-20.121689,-4.652812,-22.68952,-12.562097,-12.3339,-7.530823,-12.561728,-12.565067,-12.561728,-22.953733,-23.309378,-23.309271,-3.748662,-23.356755,-21.228136,-20.124653,-15.764006,-15.763702,-15.765666,-12.60181,-12.566366,-12.60268,-19.059852,-22.910878,-12.672093,-22.454118,-12.560946,-23.56565,-22.836257,-19.460771,-27.2791,-22.617108,-21.048041,-15.811054,-27.4447,-25.411477,-20.523639,-8.592594,-8.592594,-20.122745,-23.627695,-27.352828,-25.428954,-7.224909,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-30.0811,-22.676324,-25.676,-25.6761,-25.676,-25.676,-25.676,-25.676,-25.676,-25.676,-25.6779,-25.676,-25.6779,-22.35121,-25.69,-23.304028,-25.6813,-25.6783,-25.678,-25.678,-30.0811,-30.0811,-30.0811,-28.9647,-15.6667,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.109308,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.109308,-7.112641,-7.109308,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.151808,-7.151808,-7.112641,-7.151808,-7.155975,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.155975,-7.151808,-7.151808,-7.112641,-7.109308,-7.109308,-7.109308,-7.112641,-7.109308,-7.151808,-7.109308,-7.112641,-7.109308,-7.109308,-7.109308,-7.112641,-7.151808,-7.109308,-7.112641,-7.112641,-7.109308,-7.112641,-7.109308,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.109308,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.109308,-7.112641,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.112641,-7.151808,-7.112641,-7.151808,-7.151808,-7.179864,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.112641,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.195697,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.195697,-7.195697,-7.195697,-7.195697,-7.195697,-7.195697,-7.195697,-7.112641,-7.195697,-7.195697,-7.195697,-7.112641,-7.151808,-7.195697,-7.151808,-7.112641,-7.195697,-7.151808,-7.151808,-7.151808,-7.195697,-7.112641,-7.195697,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.179864,-7.179864,-7.179864,-7.155975,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.155975,-7.179864,-7.155975,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.109308,-7.112641,-7.109308,-7.112641,-7.112641,-7.109308,-7.109308,-7.109308,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.151808,-7.109308,-7.151808,-7.109308,-7.112641,-7.109308,-7.109308,-7.151808,-7.151808,-7.151808,-7.109308,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.112641,-7.151808,-7.112641,-7.151808,-7.151808,-7.112641,-7.112641,-7.151808,-7.151808,-7.155975,-7.112641,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.112641,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.109308,-7.112641,-7.112641,-7.179864,-7.109308,-7.112641,-7.179864,-7.112641,-7.109308,-7.112641,-7.155975,-7.109308,-7.179864,-7.109308,-7.109308,-7.112641,-7.112641,-7.112641,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.109308,-7.112641,-7.155975,-7.112641,-7.109308,-7.112641,-7.151808,-7.112641,-7.179864,-7.112641,-7.155975,-7.112641,-7.151808,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.151808,-7.112641,-7.109308,-7.151808,-7.151808,-7.112641,-7.112641,-7.155975,-7.112641,-7.151808,-7.112641,-7.109308,-7.112641,-7.195697,-7.112641,-7.112641,-7.112641,-7.155975,-7.112641,-7.155975,-7.112641,-7.155975,-7.112641,-7.155975,-7.112641,-7.155975,-7.155975,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.155975,-7.112641,-7.112641,-7.155975,-7.112641,-7.112641,-7.155975,-7.155975,-7.112641,-7.155975,-7.112641,-7.155975,-7.155975,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.109308,-7.112641,-7.155975,-7.112641,-7.155975,-7.112641,-7.195697,-7.112641,-7.195697,-7.112641,-7.112641,-7.155975,-7.112641,-7.109308,-7.112641,-7.179864,-7.109308,-7.112641,-7.195697,-7.112641,-7.112641,-7.195697,-7.195697,-7.195697,-7.112641,-7.195697,-7.155975,-7.151808,-7.151808,-7.155975,-7.151808,-7.112641,-7.151808,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.155975,-7.151808,-7.112641,-7.155975,-7.151808,-7.151808,-7.151808,-7.155975,-7.151808,-7.151808,-7.155975,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.112641,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.151808,-7.112641,-7.155975,-7.112641,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.151808,-7.112641,-7.151808,-7.151808,-7.179864,-7.151808,-7.179864,-7.151808,-7.179864,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.151808,-7.179864,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.151808,-7.179864,-7.155975,-7.155975,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-19.500426,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.109308,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.155975,-7.151808,-7.151808,-7.151808,-7.155975,-7.112641,-7.155975,-7.151808,-7.155975,-7.155975,-7.151808,-7.155975,-7.151808,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.112641,-7.112641,-7.112641,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.155975,-7.155975,-7.109308,-7.179864,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.179864,-7.112641,-7.112641,-7.179864,-7.179864,-7.112641,-7.112641,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.179864,-7.151808,-7.179864,-7.179864,-7.151808,-7.151808,-7.179864,-7.151808,-7.151808,-7.179864,-7.151808,-7.151808,-7.151808,-7.179864,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-7.151808,-7.151808,-7.151808,-7.112641,-7.179864,-7.112641,-7.151808,-7.179864,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.179864,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.112641,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.112641,-7.151808,-7.112641,-7.112641,-7.151808,-7.112641,-7.151808,-7.112641,-7.155975,-7.155975,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.155975,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.155975,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.151808,-7.151808,-7.179864,-7.151808,-7.151808,-7.151808,-7.151808,-7.151808,-7.179864,-7.179864,-7.151808,-7.179864,-7.151808,-7.151808,-7.179864,-7.151808,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-23.5475,-23.5475,-23.5475,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-16.566667,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-21.166667,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-20.15,-21.166667,-20.15,-20.15,-20.15,-20.15,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-16.566667,-16.566667,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-30.0811,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-3.2078,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-23.5475,-23.5475,-23.5475,-23.5475,-16.566667,-16.566667,-16.566667,-20.15,-20.15,-16.566667,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-16.566667,-30.0811,-23.1169,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-23.5475,-23.5475,-23.5475,-23.5475,-16.5675,-16.5675,-16.5675,-16.5675,-16.5675,-23.4339,-23.4339,-20.341667,-20.341667,-16.5675,-20.341667,-20.341667,-20.341667,-20.341667,-20.341667,-20.341667,-20.341667,-20.341667,-20.341667,-23.1169,-23.1169,-23.0986,-23.1169,-23.1169,-23.1169,-22.366667,-22.366667,-22.366667,-22.366667,-22.316667,-22.316667,-22.366667,-22.316667,-22.316667,-22.316667,-22.366667,-22.366667,-22.366667,-22.316667,-22.366667,-22.366667,-22.366667,-22.366667,-22.316667,-22.366667,-22.316667,-22.316667,-22.366667,-22.316667,-22.366667,-22.366667,-22.366667,-22.316667,-22.366667,-22.316667,-22.366667,-22.316667,-22.905556,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-22.533333,-10.963056,-10.963056,-10.963056,-22.533333,-10.963056,-10.963056,-10.963056,-24.7081,-19.748333,-19.748333,-21.555,-21.555,-18.647222,-18.647222,-20.133333,-20.133333,-20.633333,-20.633333,-20.133333,-20.133333,-20.133333,-23.4339,-23.1169,-23.1169,-23.4339,-21.555,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-22.4764,-22.4764,-22.4764,-22.4764,-22.4764,-5.466389,-5.466389,-5.466389,-5.466389,-5.466389,-22.366667,-21.75,-21.75,-21.75,-21.75,-21.75,-21.75,-11.089444,-29.448056,-22.9706,-22.9706,-22.9706,-22.9706,-22.9706,-22.9706,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.366667,-22.9706,-22.9706,-22.9706,-22.9706,-22.9706,-22.9706,-22.9706,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-21.836389,-22.905556,-16.566667,-16.585,-16.566667,-16.566667,-16.378333,-16.585,-16.566667,-16.566667,-16.566667,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-23.1169,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-16.366667,-16.366667,-18.606667,-18.606667,-16.366667,-16.366667,-18.606667,-16.366667,-16.366667,-16.366667,-16.366667,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.282778,-21.282778,-22.9056,-29.4481,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-10.833333,-11.116667,-11.766667,-11.766667,-11.816667,-11.816667,-11.816667,-11.816667,-11.816667,-11.933333,-11.936944,-11.716667,-11.966667,-11.933333,-11.933333,-11.966667,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-10.946944,-10.946944,-10.946944,-10.946944,-10.946944,-10.946944,-11.087222,-10.946944,-10.946944,-11.065278,-8.409722,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-20.345833,-20.345833,-20.345833,-20.345833,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.6039,-21.183333,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-29.448056,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-23.6039,-23.6039,-23.6039,-23.6039,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.7103,-21.555,-21.7103,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.7103,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.7103,-21.7103,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.7103,-21.7103,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.555,-21.7103,-21.555,-21.555,-21.7103,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-27.5967,-23.6039,-23.6039,-10.383333,-10.383333,-10.316667,-10.316667,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-12.316667,-23.6039,-23.6039,-23.6039,-23.6039,-23.6039,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.1139,-30.1139,-30.5414,-30.1139,-30.5414,-30.5414,-30.5414,-30.5414,-30.1139,-30.5414,-30.1139,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.1139,-30.1139,-30.5414,-30.5414,-30.1139,-30.5414,-29.4481,-29.4481,-30.5414,-30.5414,-30.5414,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.448056,-29.448056,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.4481,-29.448056,-21.555,-21.555,-21.7103,-21.266667,-21.266667,-25.4278,-21.555,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-11.633333,-10.45,-11.633333,-11.633333,-11.633333,-11.633333,-10.45,-11.633333,-11.633333,-10.45,-10.45,-10.45,-14.133333,-14.133333,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-10.45,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.7103,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-21.555,-30.1139,-30.1139,-29.4481,-29.448056,-29.4481,-29.4481,-29.448056,-29.4481,-29.4481,-29.448056,-29.4481,-29.448056,-29.4481,-29.448056,-29.448056,-29.448056,-23.5322,-23.5475,-10.708056,-10.708056,-10.708056,-10.708056,-10.708056,-10.708056,-10.708056,-22.3147,-17,-17,-17,-17,-17,-17,-17,-17,-17,-17,-17,-10.366667,-17,-17,-17,-17,-17,-17,-9.116667,-3.733333,-3.733333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-9.083333,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-29.8867,-29.8867,-30.5414,-30.5414,-29.8867,-30.5414,-23.5322,-23.5322,-23.5322,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.95,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.95,-8.483333,-8.95,-9.116667,-8.95,-8.95,-8.483333,-8.483333,-8.95,-9.116667,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.95,-8.483333,-8.483333,-8.483333,-8.483333,-8.483333,-8.95,-8.95,-8.95,-8.95,-8.95,-8.95,-8.95,-8.95,-8.95,-8.95,-9.116667,-9.116667,-9.116667,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-30.5122,-29.8867,-30.5414,-30.5122,-30.5122,-30.5414,-31.3314,-31.3314,-30.5122,-30.5414,-30.5414,-30.5414,-30.5122,-30.5122,-30.5414,-30.5414,-30.5414,-31.3314,-30.5122,-30.0331,-30.5414,-30.5122,-30.5414,-30.5122,-30.5414,-30.0331,-30.5414,-29.8867,-29.8867,-30.5414,-30.0331,-29.8867,-29.8867,-29.8867,-29.8867,-30.5414,-30.5414,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-20.25,-20.25,-20.25,-20.25,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-20.255556,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-21.722222,-21.722222,-21.722222,-21.722222,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5475,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.620278,-23.620278,-23.620278,-23.620278,-23.620278,-23.620278,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-22.905556,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.217778,-30.5414,-30.5414,-30.5414,-31.3314,-30.5414,-30.5414,-30.5414,-30.5414,-31.3314,-31.3314,-29.8867,-30.5414,-31.3314,-30.5414,-29.8867,-30.5414,-30.5414,-29.8867,-31.3314,-29.8867,-30.5414,-29.8867,-29.8867,-29.8867,-30.5414,-29.8867,-30.5122,-29.8867,-30.5122,-30.8131,-30.5122,-29.8867,-29.8867,-30.5122,-29.8867,-31.3314,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-29.8867,-30.0331,-30.0331,-30.0331,-30.5414,-30.5414,-29.8867,-29.8867,-20.326667,-20.326667,-20.326667,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-23.5322,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-22.905556,-30.5414,-30.5414,-30.5122,-30.5414,-30.1139,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5122,-30.5122,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.1139,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.0331,-30.0331,-29.8867,-30.0331,-30.0331,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-16.95,-30.0331,-30.0331,-30.8719,-30.1139,-30.8719,-30.8719,-30.8719,-30.8719,-30.1139,-30.1139,-30.8719,-30.1139,-30.8719,-30.8719,-30.8719,-30.8719,-30.8719,-30.8719,-30.8719,-30.1139,-30.8719,-30.1139,-30.8719,-30.8719,-30.1139,-30.8719,-30.1139,-30.8719,-30.5414,-30.5414,-30.8719,-30.1139,-30.8719,-30.5414,-30.8719,-30.1139,-30.8719,-30.5414,-30.5414,-30.5414,-30.1139,-30.5414,-30.8719,-30.8719,-30.8719,-30.8719,-30.5122,-30.5122,-30.5414,-30.8719,-30.8719,-30.8719,-30.5122,-30.5122,-30.5122,-30.1139,-30.5414,-30.5414,-30.5414,-30.5122,-30.8719,-30.1139,-30.8719,-30.8719,-30.5122,-30.5122,-30.1139,-30.1139,-30.1139,-30.5122,-30.1139,-30.8719,-30.5122,-30.5122,-30.8719,-30.8719,-30.5122,-30.8719,-30.1139,-30.5414,-30.5414,-30.5414,-30.5414,-30.5414,-30.5122,-30.5414,-30.5414,-30.8719,-30.8719,-30.8719,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.5122,-30.8719,-30.5414,-30.8719,-30.8719,-30.5414,-30.8719,-30.8719,-30.1139,-30.8719,-30.5414,-17.285556,-17.285556,-17.285556,-16.95,-17.285556,-17.285556,-17.285556,-17.285556,-17.285556,-16.95,-17.285556,-16.95,-16.95,-16.95,-17.285556,-16.95,-17.285556,-17.285556,-17.285556,-17.285556,-17.285556,-16.95,-17.285556,-16.95,-16.95,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-21.166667,-31.395,-31.395,-27.5967,-31.395,-27.5967,-27.5967,-31.395,-31.395,-27.5967,-27.5967,-27.5967,-27.5967,-27.5967,-27.5967,-30.1139,-30.1139,-27.5967,-27.5967,-30.1139,-30.1139,-31.3314,-27.5967,-31.3314,-31.3314,-31.3314,-31.3314,-31.3314,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-21.266667,-20.523056,-20.523056,-20.523056,-20.523056,-21.266667,-20.523056,-20.523056,-20.523056,-20.523056,-21.266667,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-20.523056,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-20.345833,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-15.3,-21.166667,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.166667,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.282778,-21.166667,-23.5475,-23.5475,-23.5475,-23.5475,-22,-22,-22,-22,-22,-21.166667,-22,-22,-21.166667,-21.166667,-22,-21.166667,-21.166667,-22,-22,-21.166667,-22,-21.166667,-22,-21.166667,-21.166667,-21.166667,-21.166667,-24.6925,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.563056,-12.185278,-12.563056,-12.563056,-12.563056,-12.563056,-24.7081,-24.7081,-24.7081,-24.7081,-21.266667,-21.166667,-21.266667,-21.266667,-21.266667,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-19.905833,-23.5475,-23.5475,-23.5475,-23.5475,-23.960556,-8.11,-8.11,-8.11,-8.11,-8.11,-8.11,-8.11,-8.11,-8.11,-8.11,-24.7081,-10.461389,-10.461389,-10.461389,-5.089167,-10.461389,-5.089167,-5.089167,-5.089167,-5.089167,-5.089167,-5.089167,-10.461389,-5.089167,-23.5475,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-24.7081,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.4671,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-24.08,-24.08,-24.08,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-24.08,-24.08,-24.08,-24.08,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-9.4142,-9.2963,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-7.021944,-3.466111,-7.021944,-7.021944,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-7.021944,-3.466111,-3.466111,-3.466111,-7.021944,-7.021944,-7.516667,-7.516667,-7.021944,-7.021944,-7.021944,-7.516667,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.466111,-3.854444,-3.854444,-4.455278,-4.455278,-3.854444,-2.934444,-4.455278,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-3.854444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-3.854444,-3.854444,-3.854444,-4.455278,-2.934444,-2.934444,-3.854444,-2.934444,-2.934444,-2.934444,-2.934444,-3.854444,-2.934444,-2.934444,-2.934444,-4.455278,-4.455278,-2.934444,-4.455278,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-2.934444,-3.453611,-3.453611,-3.453611,-3.453611,-3.453611,-3.453611,-3.453611,-3.453611,-3.453611,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-21.166667,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5322,-23.5322,-23.5322,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-23.5475,-21,-21.016667,-21.016667,-21.016667,-7.716944,-7.716944,-7.716944,-7.716944,-21.166667,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-8.05,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-21,-23,-23,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-20,-25.60574,-21,-18.918611,-23.5454,-23.5454,-23.5454,-22.9065,-22.9065,-22.9065,-22.9065,-22.7253,-22.7253,-16,-15.883333,-22,-21.17289,-21.17289,-21.17289,-21.17289,-21.17289,-21.17289,-21.17289,-21.17289,-15.8,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-1.4391,-1.4391,-1.4391,-1.4391,-1.4391,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-7.3371,-27.05,-27.18333,-27.18333,-27.18333,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-27.18333,-27.18333,-27.18333,-27.05,-22.6458,-27.05,-27.05,-27.05,-24.63333,-27.05,-27.05,-19.5992,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-21.1851,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-23.776667,-27.05,-27.05,-27.05,-27.05,-27.05,-27.05,-30.8,-20.390278,-20.390278,-20.390278,-20.390278,-21.22474,-21.22474,-21.22474,-21.22474,-21.22474,-21.22474,-21.22474,-21.22474,-20.718889,-8.95,-12.971111,-12.971111,-12.971111,-12.971111,-12.971111,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-21.55975,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.97637,-22.80574,-27.183333,-27.183333,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-22.01945,-27.25586,-27.25586,-27.25586,-27.25586,-27.25586,-22.01945,-22.01945,-22.01945,-25.41667,-24.87337,-27.21667,-27.21667,-27.21667,-27.21667,-27.21667,-27.21667,-27.21667,-25.41667,-25.41667,-27.183333,-8.76667,-7.06858,-7.06858,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-25.41667,-25.41667,-25.38333,-25.38333,-7.068,-7.068,-7.068,-25.41667,-22.80574,-22.80574,-22.80574,-22.80574,-25.41667,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-22.80574,-25.41667,-25.41667,-25.41667,-7.06858,-22.95831,-22.96334,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-22.95831,-22.95831,-27.183333,-27.183333,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.315,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-27.183333,-5.80021,-5.80021,-5.80021,-5.80021,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.315,-27.315,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-5.80021,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-5.83278,-5.83278,-5.83278,-5.80021,-5.80021,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-5.80021,-5.80021,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-5.80021,-5.80021,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-25.283,-25.283,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.183333,-27.48706,-27.25586,-27.25586,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.25586,-27.48706,-27.25586,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.3674,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.48706,-27.183333,-27.183333,-27.183333,-27.05,-27.05,-27.05,-27.05,-27.183333,-20.89218,-25.76,-25.76,-25.76,-25.76,-25.76,-25.76,-25.76,-25.76,-25.76,-25.76,-26.40843,-26.40843,-26.40843,-26.40843,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3986,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-12.9711,-12.9711,-12.9711,-12.9711,-12.9711,-12.9711,-12.9711,-12.5289,-12.5289,-12.9711,-12.9711,-12.5631,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.87,-12.87,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-12.9711,-12.87,-12.5275,-12.5275,-12.5275,-12.5275,-12.5275,-12.5275,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.9711,-12.9711,-12.9711,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.7794,-9.8967,-9.3381,-9.3381,-9.3381,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-12.7794,-11.1144,-11.1144,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-11.0894,-12.87,-12.87,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.8883,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-11.9419,-12.7794,-12.7794,-12.5275,-12.5275,-12.5275,-12.5275,-9.3381,-12.9711,-12.9711,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.6975,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-11.0894,-11.0894,-11.0894,-12.7794,-12.7794,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5289,-12.9711,-12.5631,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.87,-12.87,-12.87,-12.87,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7794,-12.7794,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.9711,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-11.0894,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.9711,-12.9711,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.9711,-12.9711,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7017,-12.7017,-12.7017,-12.7017,-12.7017,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.9711,-12.9711,-12.9711,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.8967,-9.8967,-9.8967,-9.8967,-9.8967,-9.8967,-9.8967,-9.8967,-11.0894,-11.0894,-12.7794,-12.7794,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-10.0031,-12.9711,-12.9711,-9.1619,-12.7794,-12.7794,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-12.87,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.2667,-12.9711,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-9.3381,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-12.7794,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-9.1619,-12.5631,-12.7794,-12.7794,-12.87,-23.5322,-23.1169,-21.555,-21.555,-21.555,-23.1169,-23.1169,-21.555,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.1169,-23.5475,-22.78604,-22.78604,-30.0811,-30.0811,-17.93,-17.93,-22.633333,-25.416667,-26.35,-22.633333,-27.383333,-22.633333,-15.516667,-26.35,-25.266667,-8.283333,-22.985833,-26.383333,-26.383333,-27.183333,-25.766111,-26.566667,-27.383333,-15.516667,-27.183333,-8.283333,-8.283333,-22.633333,-26.383333,-26.566667,-27.383333,-26.233611,-15.516667,-25.577472,-22.985833,-19.716667,-15.516667,-25.566667,-26.383333,-25.577472,-25.416667,-25.45,-23.533333,-22.633333,-26.383333,-22.218889,-18.853472,-14.833333,-26.433333,-25.634972,-25.766111,-26.433333,-26.383333,-26.6,-26.6,-27.183333,-18.853472,-26.016667,-26.016667,-26.016667,-25.266667,-27.183333,-21.794722,-18.853472,-25.766111,-25.766111,-25.766111,-25.766111,-25.766111,-26.016667,-26.016667,-25.766111,-25.766111,-27.183333,-26.016667,-26.016667,-25.766111,-25.766111,-25.766111,-25.766111,-22.927111,-27.183333,-18.853472,-18.853472,-25.766111,-25.766111,-25.266667,-27.183333,-18.853472,-26.016667,-25.766111,-25.766111,-26.6,-26.6,-15.483472,-26.016667,-26.016667,-26.016667,-25.766111,-25.266667,-26.6,-27.183333,-27.183333,-25.766111,-25.766111,-26.6,-17.886944,-26.016667,-26.016667,-25.766111,-25.766111,-25.766111,-25.766111,-25.766111,-27.183333,-14.520389,-26.016667,-25.766111,-14.789528,-26.6,-25.466667,-25.266667,-26.933333,-25.766111,-26.6,-26.6,-25.466667,-27.183333,-19.773056,-14.789528,-26.6,-6.855278,-27.183333,-27.183333,-21.8,-27.183333,-27.183333,-6.207222,-14.789528,-14.789528,-26.6,-26.6,-22.510833,-26.6,-26.6,-12.983333,-27.183333,-27.183333,-27.183333,-23.364556,-26.933333,-14.789528,-27.183333,-27.183333,-25.466667,-25.466667,-25.466667,-25.466667,-25.466667,-6.855278,-27.183333,-27.183333,-27.183333,-24.281556,-26.6,-26.6,-26.6,-21.022556,-25.366667,-24.281556,-22.9,-22.469556,-27.183333,-25.466667,-25.466667,-27.183333,-25.258056,-27.183333,-5.983333,-26.383333,-25.766111,-25.766111,-26.566667,-27.383333,-22.218889,-25.388889,-25.388889,-22.633333,-25.766111,-22.388583,-23.127222,-22.633333,-16.667861,-14.797222,-25.388889,-22.633333,-26.383333,-25.577472,-22.633333,-15.516667,-22.633333,-25.766111,-26.233611,-25.577472,-22.218889,-15.516667,-8.283333,-23.127222,-26.566667,-21.066667,-26.35,-0.519111,-26.383333,-22.633333,-25.75,-26.383333,-14.833333,-22.645083,-26.35,-25.388889,-26.6,-18.853472,-18.853472,-25.766111,-25.266667,-26.6,-25.766111,-25.766111,-25.766111,-26.6,-27.183333,-27.183333,-18.853472,-25.766111,-25.266667,-26.6,-26.6,-27.183333,-23.183333,-27.183333,-26.016667,-26.016667,-25.766111,-25.766111,-25.766111,-25.266667,-25.266667,-13.366667,-27.183333,-27.183333,-27.183333,-18.853472,-18.853472,-18.853472,-25.766111,-25.766111,-25.766111,-27.183333,-27.183333,-18.853472,-26.016667,-25.766111,-25.766111,-25.766111,-25.766111,-25.266667,-25.266667,-25.266667,-26.6,-26.426389,-26.016667,-26.016667,-25.766111,-26.6,-18.853472,-25.766111,-25.766111,-27.183333,-27.183333,-18.853472,-26.016667,-25.766111,-25.766111,-25.266667,-27.183333,-27.183333,-27.183333,-26.016667,-26.016667,-26.016667,-25.766111,-25.766111,-25.766111,-26.6,-14.583333,-16.667861,-27.183333,-25.466667,-25.466667,-25.466667,-27.183333,-27.183333,-27.183333,-22.975,-26.6,-26.6,-26.6,-26.6,-17.108528,-25.266667,-25.466667,-25.466667,-25.466667,-25.466667,-25.466667,-27.183333,-27.183333,-27.183333,-18.851528,-14.789528,-26.6,-27.183333,-27.183333,-19.862222,-24.281556,-14.789528,-14.789528,-14.583333,-26.6,-27.183333,-25.466667,-25.466667,-14.789528,-26.6,-27.183333,-14.789528,-26.6,-26.6,-27.183333,-25.466667,-25.466667,-27.183333,-27.183333,-24.281556,-26.6,-27.183333,-25.466667,-25.466667,-14.789528,-27.183333,-25.466667,-27.183333,-27.183333,-27.183333,-19.773056,-25.266667,-14.789528,-26.6,-27.183333,-25.466667,-27.183333,-25.266667,-19.866111],[-46.993778,-46.634645,-48.454025,-46.570871,-46.921601,-44.871074,-48.729182,-43.187947,-35.165012,-34.7965,-49.322178,-47.706258,-47.722652,-47.926761,-47.417154,-42.96285,-35.165012,-44.296301,-51.186397,-37.696331,-50.386157,-47.9078,-35.165012,-46.786144,-47.759472,-46.57058,-48.650073,-47.972003,-47.934138,-47.862839,-38.453013,-47.882942,-49.573177,-43.162482,-47.8958,-46.360705,-47.852744,-47.037756,-47.886817,-47.862324,-47.836923,-47.862698,-43.691412,-46.6912,-47.837208,-47.759906,-49.271247,-47.837226,-47.856819,-47.852503,-47.87907,-46.940289,-47.852045,-47.817405,-47.879858,-49.142389,-47.837212,-47.811723,-47.818055,-50.262032,-55.592735,-47.87734,-47.938555,-35.772048,-34.825544,-51.109953,-49.28975,-50.262032,-47.837179,-49.28972,-49.249746,-44.47716,-43.992905,-49.300688,-49.28977,-45.062434,-47.877456,-47.877444,-42.870598,-47.875918,-46.690442,-46.690433,-46.690645,-46.985516,-50.262032,-49.177812,-49.019861,-47.836203,-43.196604,-49.314904,-43.978998,-47.853618,-49.566405,-46.738199,-47.877467,-47.877478,-47.927004,-47.926972,-47.881883,-47.910619,-47.963857,-47.877644,-42.936787,-50.262032,-47.876603,-47.881945,-49.649299,-53.812911,-47.605167,-47.902076,-47.865854,-47.502402,-47.877614,-44.701847,-35.165012,-35.165012,-35.165012,-47.983846,-35.165012,-47.837246,-54.611933,-47.877478,-50.262032,-47.879969,-47.879521,-47.879078,-47.87978,-47.991673,-47.887442,-46.517993,-47.880722,-43.174734,-47.82954,-35.165012,-46.690662,-50.262032,-47.840695,-47.858611,-43.369263,-46.691142,-43.369263,-46.690587,-47.847003,-43.07096,-47.336111,-43.07096,-46.69038,-48.053431,-42.770316,-47.024453,-47.888553,-47.87635,-47.030967,-47.809452,-43.07096,-47.878685,-44.47716,-43.861755,-43.355755,-43.23106,-49.300965,-50.262344,-50.262222,-48.628457,-43.978935,-51.342789,-35.165012,-47.93993,-47.858889,-47.809469,-47.858889,-48.030486,-42.670142,-43.07096,-41.49672,-43.947636,-51.343932,-48.422212,-47.109417,-47.870369,-51.937508,-51.929552,-47.877869,-47.10461,-47.104687,-38.386138,-38.413105,-38.507717,-48.911859,-40.666597,-47.875722,-47.515995,-46.596119,-47.807148,-47.871319,-43.706379,-49.283433,-44.888637,-48.052074,-47.777152,-47.828641,-47.86602,-51.186641,-46.525053,-51.18639,-48.829475,-51.186111,-46.730689,-47.875001,-49.968046,-47.788733,-47.788233,-47.825832,-48.645275,-46.742512,-47.05726,-38.506693,-48.497202,-49.401299,-51.342902,-51.933617,-46.626091,-47.859084,-47.105223,-47.880028,-47.836863,-47.858891,-47.852488,-47.837025,-47.926521,-47.019883,-47.880251,-47.858891,-47.606363,-48.010471,-40.756398,-47.928294,-51.933447,-38.401637,-43.225095,-47.880222,-48.503218,-38.555365,-46.176503,-38.573405,-38.573405,-47.877003,-49.229367,-47.859659,-49.306753,-46.698599,-38.573405,-47.859659,-47.859659,-39.298059,-46.980347,-49.141754,-47.826348,-47.003292,-46.551907,-48.614913,-38.41647,-49.232968,-48.497252,-46.566512,-47.046943,-44.220037,-37.461328,-46.880139,-41.3947,-41.2334,-38.494678,-41.389876,-41.393586,-41.389876,-43.295193,-44.876822,-44.877015,-38.485697,-44.886233,-44.979905,-44.218962,-47.85641,-47.856084,-47.854869,-41.466522,-41.390755,-41.493134,-39.983376,-47.022263,-41.453787,-42.973502,-41.392362,-46.722403,-41.979201,-45.596489,-55.5784,-42.407247,-47.409833,-47.846779,-54.9403,-49.351487,-43.691412,-37.247526,-37.247526,-44.221655,-46.557143,-49.350105,-49.267137,-34.923267,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-51.0233,-44.138127,-54.4528,-54.4531,-54.4528,-54.4528,-54.4528,-54.4528,-54.4528,-54.4528,-54.452,-54.4528,-54.452,-42.326379,-54.44,-47.667581,-54.1626,-54.4491,-54.449,-54.449,-51.0233,-51.0233,-51.0233,-50.068,-43.5167,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.850565,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.850565,-34.853065,-34.850565,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.849176,-34.849176,-34.853065,-34.849176,-34.853621,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853621,-34.849176,-34.849176,-34.853065,-34.850565,-34.850565,-34.850565,-34.853065,-34.850565,-34.849176,-34.850565,-34.853065,-34.850565,-34.850565,-34.850565,-34.853065,-34.849176,-34.850565,-34.853065,-34.853065,-34.850565,-34.853065,-34.850565,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.850565,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.850565,-34.853065,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.853065,-34.849176,-34.853065,-34.849176,-34.849176,-34.819732,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.853065,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.822509,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.822509,-34.822509,-34.822509,-34.822509,-34.822509,-34.822509,-34.822509,-34.853065,-34.822509,-34.822509,-34.822509,-34.853065,-34.849176,-34.822509,-34.849176,-34.853065,-34.822509,-34.849176,-34.849176,-34.849176,-34.822509,-34.853065,-34.822509,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.819732,-34.819732,-34.819732,-34.853621,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.853621,-34.819732,-34.853621,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.850565,-34.853065,-34.850565,-34.853065,-34.853065,-34.850565,-34.850565,-34.850565,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.849176,-34.850565,-34.849176,-34.850565,-34.853065,-34.850565,-34.850565,-34.849176,-34.849176,-34.849176,-34.850565,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.853065,-34.849176,-34.853065,-34.849176,-34.849176,-34.853065,-34.853065,-34.849176,-34.849176,-34.853621,-34.853065,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.853065,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.850565,-34.853065,-34.853065,-34.819732,-34.850565,-34.853065,-34.819732,-34.853065,-34.850565,-34.853065,-34.853621,-34.850565,-34.819732,-34.850565,-34.850565,-34.853065,-34.853065,-34.853065,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.850565,-34.853065,-34.853621,-34.853065,-34.850565,-34.853065,-34.849176,-34.853065,-34.819732,-34.853065,-34.853621,-34.853065,-34.849176,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.849176,-34.853065,-34.850565,-34.849176,-34.849176,-34.853065,-34.853065,-34.853621,-34.853065,-34.849176,-34.853065,-34.850565,-34.853065,-34.822509,-34.853065,-34.853065,-34.853065,-34.853621,-34.853065,-34.853621,-34.853065,-34.853621,-34.853065,-34.853621,-34.853065,-34.853621,-34.853621,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.853621,-34.853065,-34.853065,-34.853621,-34.853065,-34.853065,-34.853621,-34.853621,-34.853065,-34.853621,-34.853065,-34.853621,-34.853621,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.850565,-34.853065,-34.853621,-34.853065,-34.853621,-34.853065,-34.822509,-34.853065,-34.822509,-34.853065,-34.853065,-34.853621,-34.853065,-34.850565,-34.853065,-34.819732,-34.850565,-34.853065,-34.822509,-34.853065,-34.853065,-34.822509,-34.822509,-34.822509,-34.853065,-34.822509,-34.853621,-34.849176,-34.849176,-34.853621,-34.849176,-34.853065,-34.849176,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.853621,-34.849176,-34.853065,-34.853621,-34.849176,-34.849176,-34.849176,-34.853621,-34.849176,-34.849176,-34.853621,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.853065,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.849176,-34.853065,-34.853621,-34.853065,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.849176,-34.853065,-34.849176,-34.849176,-34.819732,-34.849176,-34.819732,-34.849176,-34.819732,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.849176,-34.819732,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.849176,-34.819732,-34.853621,-34.853621,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-40.605469,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.850565,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853621,-34.849176,-34.849176,-34.849176,-34.853621,-34.853065,-34.853621,-34.849176,-34.853621,-34.853621,-34.849176,-34.853621,-34.849176,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.853065,-34.853065,-34.853065,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853621,-34.853621,-34.850565,-34.819732,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.819732,-34.853065,-34.853065,-34.819732,-34.819732,-34.853065,-34.853065,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.819732,-34.849176,-34.819732,-34.819732,-34.849176,-34.849176,-34.819732,-34.849176,-34.849176,-34.819732,-34.849176,-34.849176,-34.849176,-34.819732,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-34.849176,-34.849176,-34.849176,-34.853065,-34.819732,-34.853065,-34.849176,-34.819732,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.819732,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.853065,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.853065,-34.849176,-34.853065,-34.853065,-34.849176,-34.853065,-34.849176,-34.853065,-34.853621,-34.853621,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.853621,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.853621,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.849176,-34.849176,-34.819732,-34.849176,-34.849176,-34.849176,-34.849176,-34.849176,-34.819732,-34.819732,-34.849176,-34.819732,-34.849176,-34.849176,-34.819732,-34.849176,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-46.6361,-46.6361,-46.6361,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.983333,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.633333,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.566667,-47.633333,-47.566667,-47.566667,-47.566667,-47.566667,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.983333,-45.983333,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-51.0233,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-43.4036,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-46.6361,-46.6361,-46.6361,-46.6361,-45.983333,-45.983333,-45.983333,-47.566667,-47.566667,-45.983333,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-45.983333,-51.0233,-46.5503,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-46.6361,-46.6361,-46.6361,-46.6361,-45.99,-45.99,-45.99,-45.99,-45.99,-45.0711,-45.0711,-49.195,-49.195,-45.99,-49.195,-49.195,-49.195,-49.195,-49.195,-49.195,-49.195,-49.195,-49.195,-46.5503,-46.5503,-48.9258,-46.5503,-46.5503,-46.5503,-41.783333,-41.783333,-41.783333,-41.783333,-42.166667,-42.166667,-41.783333,-42.166667,-42.166667,-42.166667,-41.783333,-41.783333,-41.783333,-42.166667,-41.783333,-41.783333,-41.783333,-41.783333,-42.166667,-41.783333,-42.166667,-42.166667,-41.783333,-42.166667,-41.783333,-41.783333,-41.783333,-42.166667,-41.783333,-42.166667,-41.783333,-42.166667,-47.060833,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-52.316667,-38.786667,-38.786667,-38.786667,-52.316667,-38.786667,-38.786667,-38.786667,-47.5553,-47.931944,-47.931944,-47.704444,-47.704444,-48.187222,-48.187222,-46.9,-46.9,-46.216667,-46.216667,-46.9,-46.9,-46.9,-45.0711,-46.5503,-46.5503,-45.0711,-47.704444,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.6328,-46.6328,-46.6328,-46.6328,-46.6328,-47.8875,-47.8875,-47.8875,-47.8875,-47.8875,-41.783333,-41.316667,-41.316667,-41.316667,-41.316667,-41.316667,-41.316667,-43.141667,-50.583333,-46.9958,-46.9958,-46.9958,-46.9958,-46.9958,-46.9958,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-41.783333,-46.9958,-46.9958,-46.9958,-46.9958,-46.9958,-46.9958,-46.9958,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-46.743611,-47.060833,-45.983333,-39.103333,-45.983333,-45.983333,-39.018611,-39.103333,-45.983333,-45.983333,-45.983333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-46.5503,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-45.083333,-45.083333,-44.391667,-44.391667,-45.083333,-45.083333,-44.391667,-45.083333,-45.083333,-45.083333,-45.083333,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.169722,-47.169722,-47.0608,-50.5836,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-62.316667,-62.8,-62.716667,-62.716667,-62.833333,-62.833333,-62.833333,-62.833333,-62.833333,-62.716667,-62.7175,-62.733333,-61.983333,-62.716667,-62.716667,-61.983333,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-62.481944,-62.481944,-62.481944,-62.481944,-62.481944,-62.481944,-62.741944,-62.481944,-62.481944,-62.665278,-63.526111,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.4825,-46.4825,-46.4825,-46.4825,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.9192,-47.716667,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-50.583333,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-46.9192,-46.9192,-46.9192,-46.9192,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.4781,-47.7044,-47.4781,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.4781,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.4781,-47.4781,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.4781,-47.4781,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.7044,-47.4781,-47.7044,-47.7044,-47.4781,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-48.5492,-46.9192,-46.9192,-65,-65,-64.55,-64.55,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-64.233333,-46.9192,-46.9192,-46.9192,-46.9192,-46.9192,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-51.325,-51.325,-53.4869,-51.325,-53.4869,-53.4869,-53.4869,-53.4869,-51.325,-53.4869,-51.325,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-51.325,-51.325,-53.4869,-53.4869,-51.325,-53.4869,-50.5836,-50.5836,-53.4869,-53.4869,-53.4869,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.583333,-50.583333,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.5836,-50.583333,-47.7044,-47.7044,-47.4781,-47.3,-47.3,-49.2731,-47.7044,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.8,-46.316667,-46.8,-46.8,-46.8,-46.8,-46.316667,-46.8,-46.8,-46.316667,-46.316667,-46.316667,-44.666667,-44.666667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-46.316667,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.4781,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-47.7044,-51.325,-51.325,-50.5836,-50.583333,-50.5836,-50.5836,-50.583333,-50.5836,-50.5836,-50.583333,-50.5836,-50.583333,-50.5836,-50.583333,-50.583333,-50.583333,-45.8464,-46.6361,-48.417222,-48.417222,-48.417222,-48.417222,-48.417222,-48.417222,-48.417222,-49.0606,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-48.133333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-42.733333,-45.916667,-43.35,-43.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-44.35,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-50.2697,-50.2697,-53.4869,-53.4869,-50.2697,-53.4869,-45.8464,-45.8464,-45.8464,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-44.583333,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-44.583333,-47.366667,-44.583333,-45.916667,-44.583333,-44.583333,-47.366667,-47.366667,-44.583333,-45.916667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-44.583333,-47.366667,-47.366667,-47.366667,-47.366667,-47.366667,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-44.583333,-45.916667,-45.916667,-45.916667,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-53.4914,-50.2697,-53.4869,-53.4914,-53.4914,-53.4869,-54.1069,-54.1069,-53.4914,-53.4869,-53.4869,-53.4869,-53.4914,-53.4914,-53.4869,-53.4869,-53.4869,-54.1069,-53.4914,-51.23,-53.4869,-53.4914,-53.4869,-53.4914,-53.4869,-51.23,-53.4869,-50.2697,-50.2697,-53.4869,-51.23,-50.2697,-50.2697,-50.2697,-50.2697,-53.4869,-53.4869,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.416667,-46.416667,-46.416667,-46.416667,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-47.476667,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-44.985278,-44.985278,-44.985278,-44.985278,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-46.6361,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.413056,-45.413056,-45.413056,-45.413056,-45.413056,-45.413056,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-47.060833,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-44.713056,-53.4869,-53.4869,-53.4869,-54.1069,-53.4869,-53.4869,-53.4869,-53.4869,-54.1069,-54.1069,-50.2697,-53.4869,-54.1069,-53.4869,-50.2697,-53.4869,-53.4869,-50.2697,-54.1069,-50.2697,-53.4869,-50.2697,-50.2697,-50.2697,-53.4869,-50.2697,-53.4914,-50.2697,-53.4914,-53.895,-53.4914,-50.2697,-50.2697,-53.4914,-50.2697,-54.1069,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-50.2697,-51.23,-51.23,-51.23,-53.4869,-53.4869,-50.2697,-50.2697,-46.366111,-46.366111,-46.366111,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-45.8464,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-47.060833,-53.4869,-53.4869,-53.4914,-53.4869,-51.325,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4914,-53.4914,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-51.325,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-51.23,-51.23,-50.2697,-51.23,-51.23,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-42.716667,-51.23,-51.23,-53.1153,-51.325,-53.1153,-53.1153,-53.1153,-53.1153,-51.325,-51.325,-53.1153,-51.325,-53.1153,-53.1153,-53.1153,-53.1153,-53.1153,-53.1153,-53.1153,-51.325,-53.1153,-51.325,-53.1153,-53.1153,-51.325,-53.1153,-51.325,-53.1153,-53.4869,-53.4869,-53.1153,-51.325,-53.1153,-53.4869,-53.1153,-51.325,-53.1153,-53.4869,-53.4869,-53.4869,-51.325,-53.4869,-53.1153,-53.1153,-53.1153,-53.1153,-53.4914,-53.4914,-53.4869,-53.1153,-53.1153,-53.1153,-53.4914,-53.4914,-53.4914,-51.325,-53.4869,-53.4869,-53.4869,-53.4914,-53.1153,-51.325,-53.1153,-53.1153,-53.4914,-53.4914,-51.325,-51.325,-51.325,-53.4914,-51.325,-53.1153,-53.4914,-53.4914,-53.1153,-53.1153,-53.4914,-53.1153,-51.325,-53.4869,-53.4869,-53.4869,-53.4869,-53.4869,-53.4914,-53.4869,-53.4869,-53.1153,-53.1153,-53.1153,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.4914,-53.1153,-53.4869,-53.1153,-53.1153,-53.4869,-53.1153,-53.1153,-51.325,-53.1153,-53.4869,-42.73,-42.73,-42.73,-42.716667,-42.73,-42.73,-42.73,-42.73,-42.73,-42.716667,-42.73,-42.716667,-42.716667,-42.716667,-42.73,-42.716667,-42.73,-42.73,-42.73,-42.73,-42.73,-42.716667,-42.73,-42.716667,-42.716667,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-47.633333,-52.6756,-52.6756,-48.5492,-52.6756,-48.5492,-48.5492,-52.6756,-52.6756,-48.5492,-48.5492,-48.5492,-48.5492,-48.5492,-48.5492,-51.325,-51.325,-48.5492,-48.5492,-51.325,-51.325,-54.1069,-48.5492,-54.1069,-54.1069,-54.1069,-54.1069,-54.1069,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-47.3,-41.904444,-41.904444,-41.904444,-41.904444,-47.3,-41.904444,-41.904444,-41.904444,-41.904444,-47.3,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-41.904444,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-46.4825,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-44.416667,-47.633333,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.633333,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.169722,-47.633333,-46.6361,-46.6361,-46.6361,-46.6361,-46,-46,-46,-46,-46,-47.633333,-46,-46,-47.633333,-47.633333,-46,-47.633333,-47.633333,-46,-46,-47.633333,-46,-47.633333,-46,-47.633333,-47.633333,-47.633333,-47.633333,-48.002222,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-43.220556,-41.39,-41.39,-41.39,-41.39,-47.5553,-47.5553,-47.5553,-47.5553,-47.3,-47.633333,-47.3,-47.3,-47.3,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-50.726944,-46.6361,-46.6361,-46.6361,-46.6361,-46.333611,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-42.944444,-47.5553,-40.189167,-40.189167,-40.189167,-42.801667,-40.189167,-42.801667,-42.801667,-42.801667,-42.801667,-42.801667,-42.801667,-40.189167,-42.801667,-46.6361,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-47.5553,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-56.4886,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-54.2558,-54.2558,-54.2558,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-54.2558,-54.2558,-54.2558,-54.2558,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-75.9709,-75.9972,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-45.481111,-43.905,-45.481111,-45.481111,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-45.481111,-43.905,-43.905,-43.905,-45.481111,-45.481111,-45.766667,-45.766667,-45.481111,-45.481111,-45.481111,-45.766667,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-43.905,-40.921111,-40.921111,-43.885556,-43.885556,-40.921111,-44.235,-43.885556,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-40.921111,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-40.921111,-40.921111,-40.921111,-43.885556,-44.235,-44.235,-40.921111,-44.235,-44.235,-44.235,-44.235,-40.921111,-44.235,-44.235,-44.235,-43.885556,-43.885556,-44.235,-43.885556,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.235,-44.763333,-44.763333,-44.763333,-44.763333,-44.763333,-44.763333,-44.763333,-44.763333,-44.763333,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-47.633333,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-45.8464,-45.8464,-45.8464,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-46.6361,-48,-47.366667,-47.366667,-47.366667,-51.183333,-51.183333,-51.183333,-51.183333,-47.633333,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-34.9,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-44,-55,-55,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-48,-54.57656,-48,-48.277222,-46.6379,-46.6379,-46.6379,-47.0637,-47.0637,-47.0637,-47.0637,-47.6416,-47.6416,-52,-52.25,-46,-47.80151,-47.80151,-47.80151,-47.80151,-47.80151,-47.80151,-47.80151,-47.80151,-47.916667,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-48.4975,-48.4975,-48.4975,-48.4975,-48.4975,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-47.4626,-52.4,-52.38333,-52.38333,-52.38333,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-52.38333,-52.38333,-52.38333,-52.4,-47.1961,-52.4,-52.4,-52.4,-54.11667,-52.4,-52.4,-46.9394,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-47.8002,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-45.409722,-52.4,-52.4,-52.4,-52.4,-52.4,-52.4,-51.83333,-40.496111,-40.496111,-40.496111,-40.496111,-43.76667,-43.76667,-43.76667,-43.76667,-43.76667,-43.76667,-43.76667,-43.76667,-46.609722,-54.9,-38.510833,-38.510833,-38.510833,-38.510833,-38.510833,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-45.43465,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.27777,-43.21033,-52.383333,-52.383333,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-47.8894,-55.5339,-55.5339,-55.5339,-55.5339,-55.5339,-47.8894,-47.8894,-47.8894,-49.25,-64.38975,-55.6,-55.6,-55.6,-55.6,-55.6,-55.6,-55.6,-49.25,-49.25,-52.383333,-63.9,-36.57816,-36.57816,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-49.25,-49.25,-51.45,-51.45,-36.578,-36.578,-36.578,-49.25,-43.21033,-43.21033,-43.21033,-43.21033,-49.25,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-43.21033,-49.25,-49.25,-49.25,-36.57816,-43.27721,-43.26636,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-43.27721,-43.27721,-52.383333,-52.383333,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-55.533,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-52.383333,-35.21067,-35.21067,-35.21067,-35.21067,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-55.533,-55.533,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-35.21067,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-35.21194,-35.21194,-35.21194,-35.21067,-35.21067,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-35.21067,-35.21067,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-35.21067,-35.21067,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-57.633,-57.633,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-52.383333,-55.11994,-55.5339,-55.5339,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.5339,-55.11994,-55.5339,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.58091,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-55.11994,-52.383333,-52.383333,-52.383333,-52.4,-52.4,-52.4,-52.4,-52.383333,-47.59014,-56.41,-56.41,-56.41,-56.41,-56.41,-56.41,-56.41,-56.41,-56.41,-56.41,-54.69463,-54.69463,-54.69463,-54.69463,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-40.5008,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-43.1417,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-38.5108,-38.5108,-38.5108,-38.5108,-38.5108,-38.5108,-38.5108,-41.5589,-41.5589,-38.5108,-38.5108,-41.39,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.8589,-39.8589,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-38.5108,-39.8589,-40.3069,-40.3069,-40.3069,-40.3069,-40.3069,-40.3069,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-38.5108,-38.5108,-38.5108,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.17,-39.0264,-38.2547,-38.2547,-38.2547,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-39.17,-42.7192,-42.7192,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-43.1417,-39.8589,-39.8589,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-43.1417,-39.17,-39.17,-39.17,-39.17,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-38.6786,-39.17,-39.17,-39.17,-39.17,-39.17,-38.0844,-39.17,-39.17,-40.3069,-40.3069,-40.3069,-40.3069,-38.2547,-38.5108,-38.5108,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-38.3242,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-43.1417,-43.1417,-43.1417,-39.17,-39.17,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.5589,-38.5108,-41.39,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.8589,-39.8589,-39.8589,-39.8589,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.17,-39.17,-43.1417,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-38.5108,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-43.1417,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-38.5108,-38.5108,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-38.5108,-38.5108,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.708,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-41.8275,-41.8275,-41.8275,-41.8275,-41.8275,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-38.5108,-38.5108,-38.5108,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.0264,-39.0264,-39.0264,-39.0264,-39.0264,-39.0264,-39.0264,-39.0264,-43.1417,-43.1417,-39.17,-39.17,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-42.5044,-38.5108,-38.5108,-40.708,-39.17,-39.17,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-39.8589,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.9667,-38.5108,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-38.2547,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-39.17,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-40.9708,-41.39,-39.17,-39.17,-39.8589,-45.8464,-46.5503,-47.7044,-47.7044,-47.7044,-46.5503,-46.5503,-47.7044,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.5503,-46.6361,-43.30904,-43.30904,-51.0233,-51.0233,-43.79,-43.79,-47.2,-49.25,-50.883333,-47.2,-55.966667,-47.2,-40.9,-50.883333,-57.666667,-35.966667,-43.210278,-54.666667,-54.666667,-52.383333,-56.510833,-54.783333,-55.966667,-40.9,-52.383333,-35.966667,-35.966667,-47.2,-54.666667,-54.783333,-55.966667,-54.636111,-40.9,-48.774722,-43.210278,-44.566667,-40.9,-54.566667,-54.666667,-48.774722,-49.25,-48.633333,-46.616667,-44.566667,-54.666667,-54.8,-41.942861,-55.983333,-49.233333,-56.821472,-56.510833,-49.233333,-54.666667,-49.016667,-49.016667,-52.383333,-41.942861,-57.05,-57.05,-57.05,-57.666667,-52.383333,-48.176111,-41.942861,-56.510833,-56.510833,-56.510833,-56.510833,-56.510833,-57.05,-57.05,-56.510833,-56.510833,-52.383333,-57.05,-57.05,-56.510833,-56.510833,-56.510833,-56.510833,-43.230583,-52.383333,-41.942861,-41.942861,-56.510833,-56.510833,-57.666667,-52.383333,-41.942861,-57.05,-56.510833,-56.510833,-49.016667,-49.016667,-47.306139,-57.05,-57.05,-57.05,-56.510833,-57.666667,-49.016667,-52.383333,-52.383333,-56.510833,-56.510833,-49.016667,-44.577778,-57.05,-57.05,-56.510833,-56.510833,-56.510833,-56.510833,-56.510833,-52.383333,-47.504306,-57.05,-56.510833,-39.049333,-49.016667,-56.011111,-57.666667,-49.05,-56.510833,-49.016667,-49.016667,-56.011111,-52.383333,-47.933333,-39.049333,-49.016667,-35.492222,-52.383333,-52.383333,-52.45,-52.383333,-52.383333,-52.702778,-39.049333,-39.049333,-49.016667,-49.016667,-43.184444,-49.016667,-49.016667,-38.516667,-52.383333,-52.383333,-52.383333,-46.741417,-49.05,-39.049333,-52.383333,-52.383333,-56.011111,-56.011111,-56.011111,-56.011111,-56.011111,-35.492222,-52.383333,-52.383333,-52.383333,-47.460417,-49.016667,-49.016667,-49.016667,-44.318333,-57.616667,-47.460417,-43.233333,-44.4475,-52.383333,-56.011111,-56.011111,-52.383333,-57.576944,-52.383333,-35.933333,-54.666667,-56.510833,-56.510833,-54.783333,-55.966667,-54.8,-57.266667,-57.266667,-47.2,-56.510833,-44.963083,-54.957778,-47.2,-49.2915,-39.034444,-57.266667,-44.566667,-54.666667,-48.774722,-47.2,-40.9,-47.2,-56.510833,-54.636111,-48.774722,-54.8,-40.9,-35.966667,-54.957778,-54.783333,-47.833333,-50.883333,-50.785556,-54.666667,-47.2,-56.5,-54.666667,-55.983333,-44.576528,-50.883333,-57.266667,-49.016667,-41.942861,-41.942861,-56.510833,-57.666667,-49.016667,-56.510833,-56.510833,-56.510833,-49.016667,-52.383333,-52.383333,-41.942861,-56.510833,-57.666667,-49.016667,-49.016667,-52.383333,-46.866667,-52.383333,-57.05,-57.05,-56.510833,-56.510833,-56.510833,-57.666667,-57.666667,-39.7,-52.383333,-52.383333,-52.383333,-41.942861,-41.942861,-41.942861,-56.510833,-56.510833,-56.510833,-52.383333,-52.383333,-41.942861,-57.05,-56.510833,-56.510833,-56.510833,-56.510833,-57.666667,-57.666667,-57.666667,-49.016667,-49.243278,-57.05,-57.05,-56.510833,-49.016667,-41.942861,-56.510833,-56.510833,-52.383333,-52.383333,-41.942861,-57.05,-56.510833,-56.510833,-57.666667,-52.383333,-52.383333,-52.383333,-57.05,-57.05,-57.05,-56.510833,-56.510833,-56.510833,-49.016667,-39.266667,-49.2915,-52.383333,-56.011111,-56.011111,-56.011111,-52.383333,-52.383333,-52.383333,-53.605,-49.016667,-49.016667,-49.016667,-49.016667,-43.815389,-57.666667,-56.011111,-56.011111,-56.011111,-56.011111,-56.011111,-52.383333,-52.383333,-52.383333,-41.949361,-39.049333,-49.016667,-52.383333,-52.383333,-43.970556,-47.460417,-39.049333,-39.049333,-39.266667,-49.016667,-52.383333,-56.011111,-56.011111,-39.049333,-49.016667,-52.383333,-39.049333,-49.016667,-49.016667,-52.383333,-56.011111,-56.011111,-52.383333,-52.383333,-47.460417,-49.016667,-52.383333,-56.011111,-56.011111,-39.049333,-52.383333,-56.011111,-52.383333,-52.383333,-52.383333,-47.933333,-57.316667,-39.049333,-49.016667,-52.383333,-56.011111,-52.383333,-57.316667,-43.964444],1,null,null,{"interactive":true,"className":"","stroke":true,"color":"black","weight":5,"opacity":0.5,"fill":true,"fillColor":"black","fillOpacity":0.2},null,null,null,null,null,{"interactive":false,"permanent":false,"direction":"auto","opacity":1,"offset":[0,0],"textsize":"10px","textOnly":false,"className":"","sticky":true},null]},{"method":"addScaleBar","args":[{"maxWidth":100,"metric":true,"imperial":true,"updateWhenIdle":true,"position":"topright"}]}],"limits":{"lat":[-40,0],"lng":[-81.333333333,-34.7965]}},"evals":[],"jsHooks":[]}</script> --- class: center, middle # Agradecimentos ### [R-Ladies BH](https://rladiesbh.com.br/) 💙 ### [Grupo de Estudos "Niche Group"](https://linktr.ee/niche_group) 💛 <img src="https://media1.tenor.com/images/ceadcc4af571518e125e7964e43bb714/tenor.gif?itemid=5459333" style="width: 20%;"> --- class: center, middle # Acessar essa apresentação em ## www.mmfava.com